data(penguins, package = "palmerpenguins")
penguins_sub <- penguins |>
filter(species %in% c("Adelie","Gentoo")) |>
drop_na() |>
mutate(species = fct_drop(species))
pairs(penguins_sub)

DS3 - Explorative Datenanalyse & Data Mining
Saskia Otto
Universität Hamburg, IMF
Wintersemester 2023/2024
![]()
Image by starline on Freepik
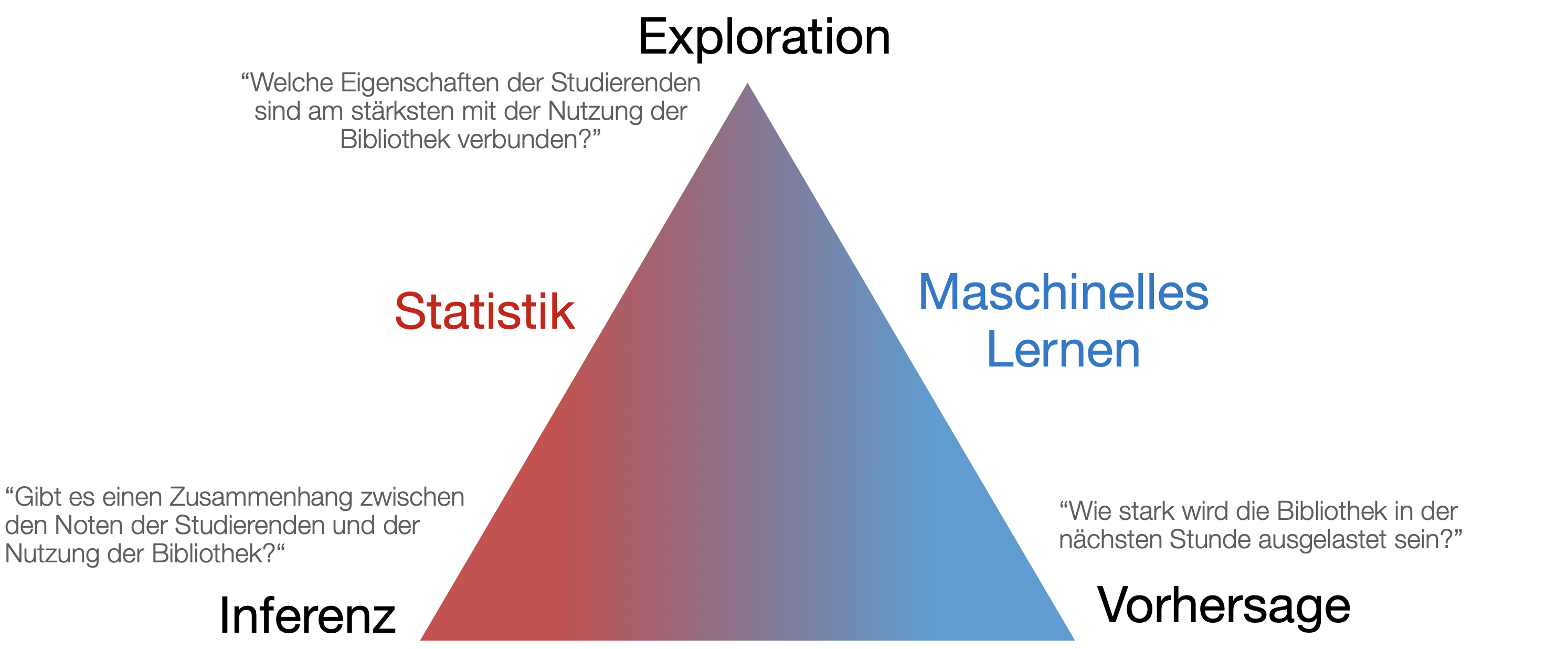
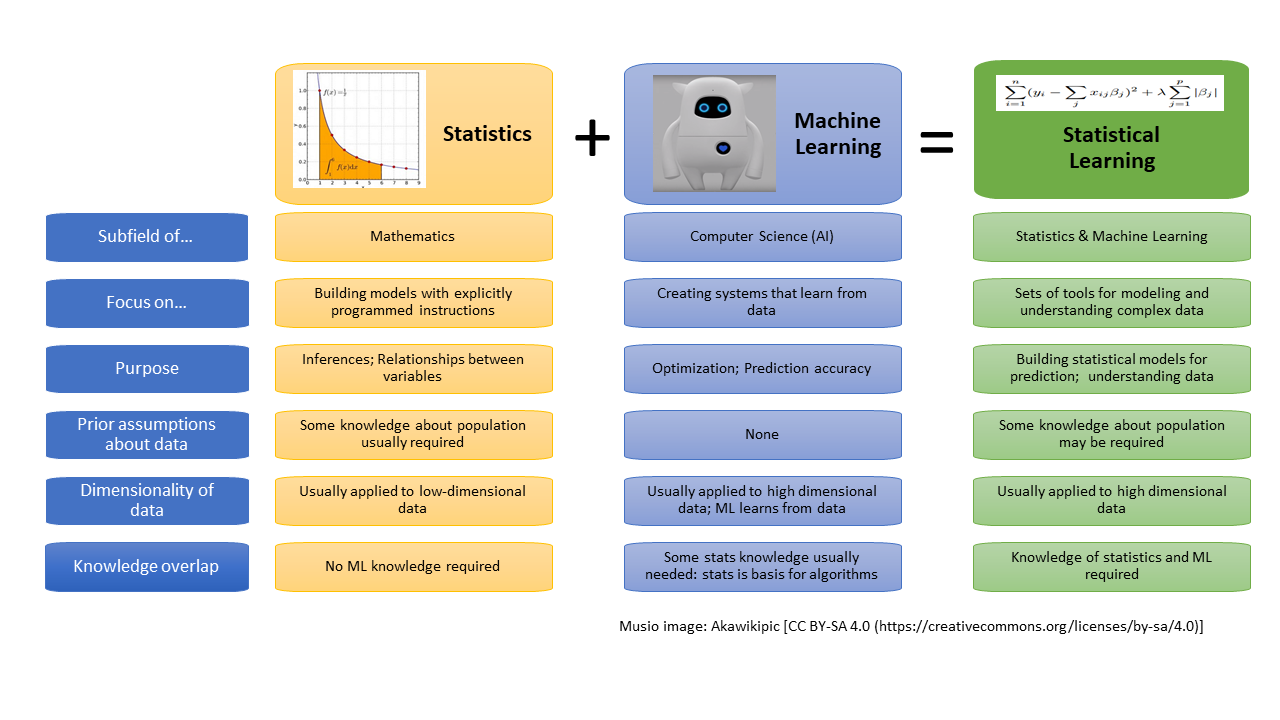
Die Explorative Datenanalyse (EDA) und Data Mining sind zwei verschiedene, jedoch miteinander verbundene Konzepte im Bereich der Datenanalyse:













→ Die Wissenschaft des Lernens spielt eine Schlüsselrolle in den Bereichen Statistik, Data Mining und künstliche Intelligenz und überschneidet sich mit Bereichen der Ingenieurwissenschaften und anderen Disziplinen.
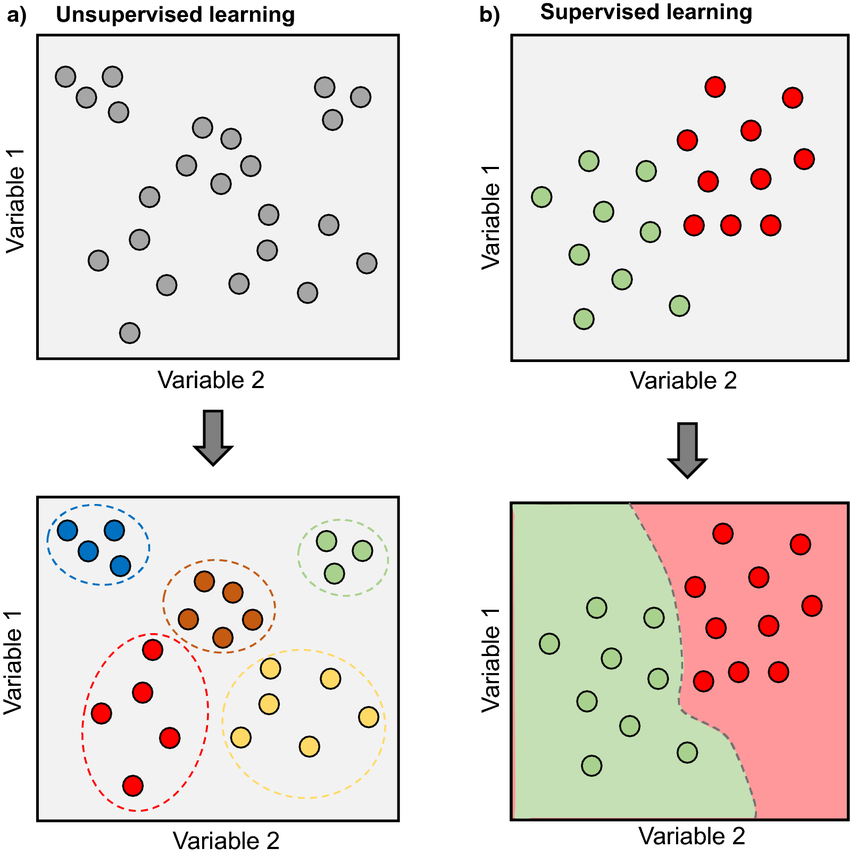
![]()
Bildquelle: Morimoto & Ponton (2021): Virtual reality in biology: could we become virtual naturalists?
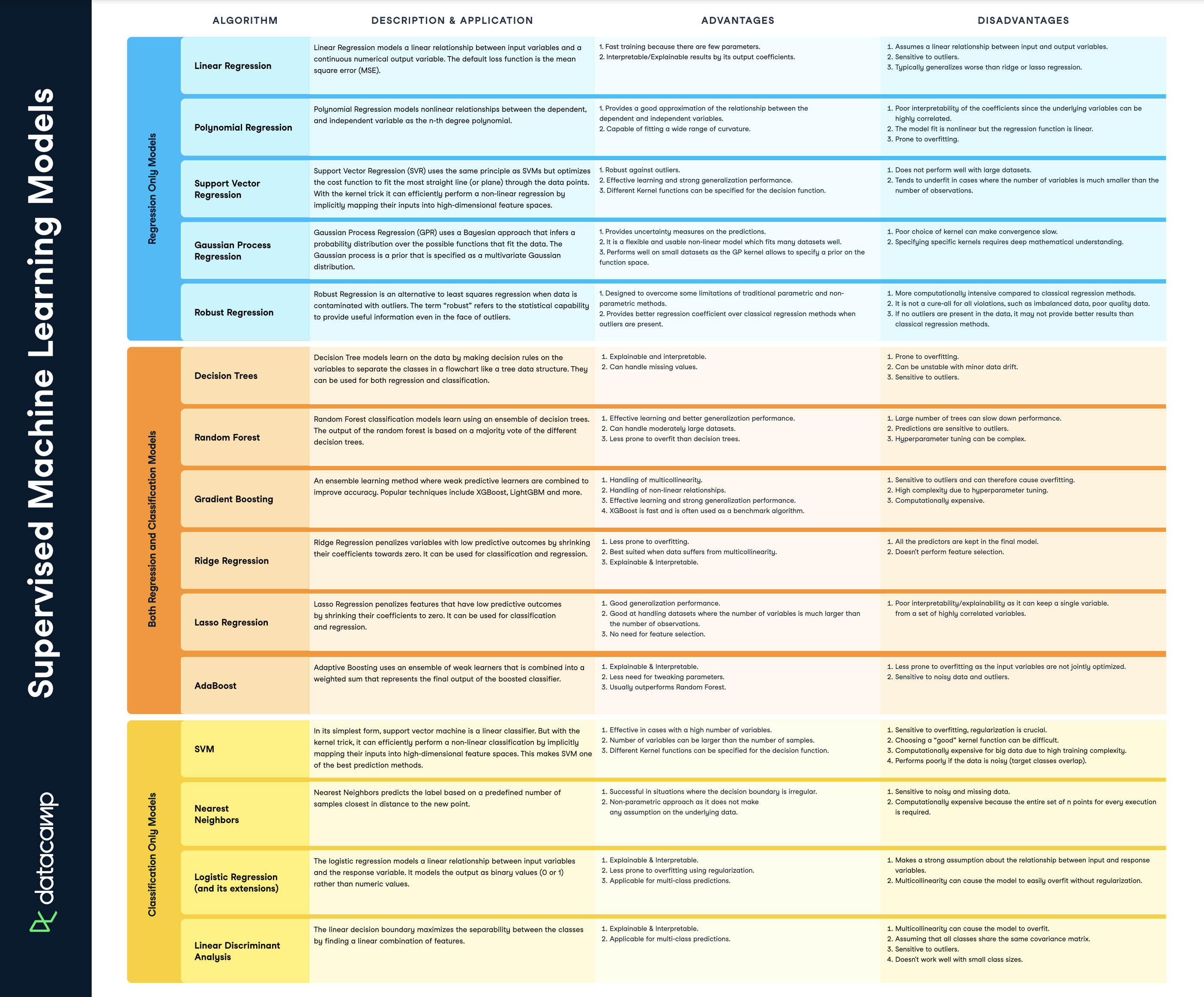
Download-link dieses Cheatsheets: [datacamp.com](https://www.datacamp.com/cheat-sheet/)


Einfach für ein oder zwei Parameter…
Nicht einfach für >> 2 Parameter…
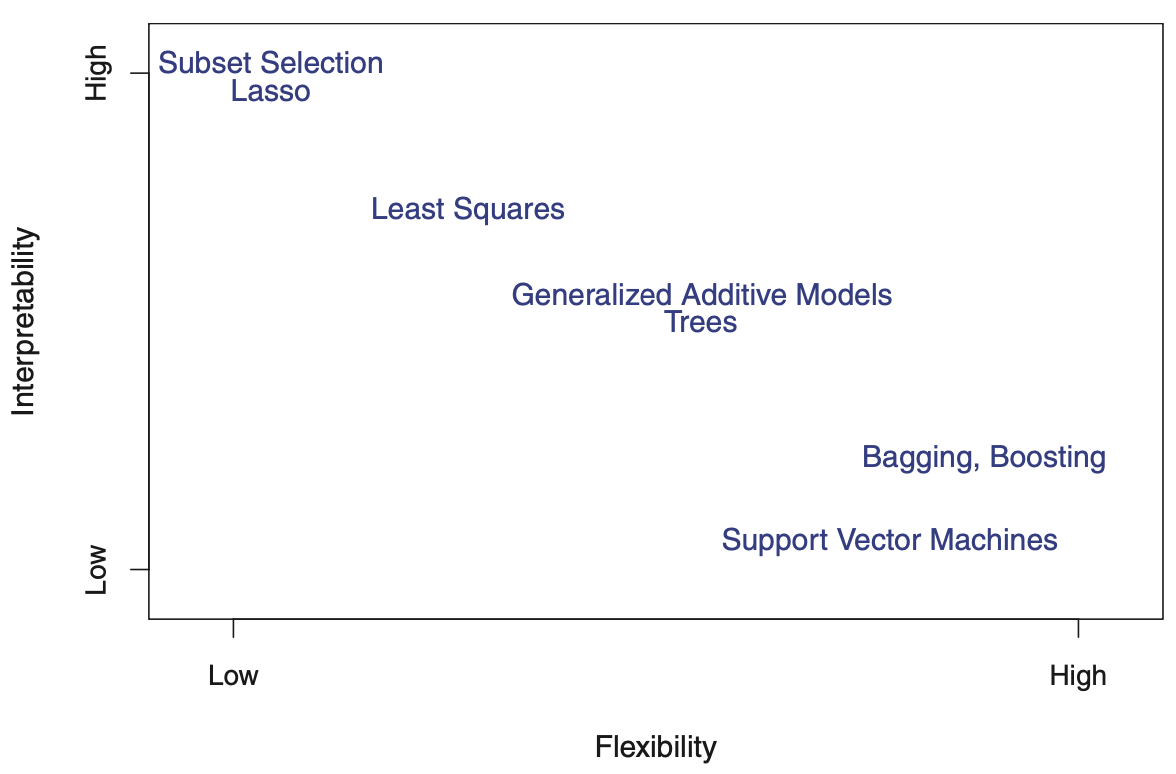
Bildquelle: James et al. (2013): Introduction to Statistical Learning
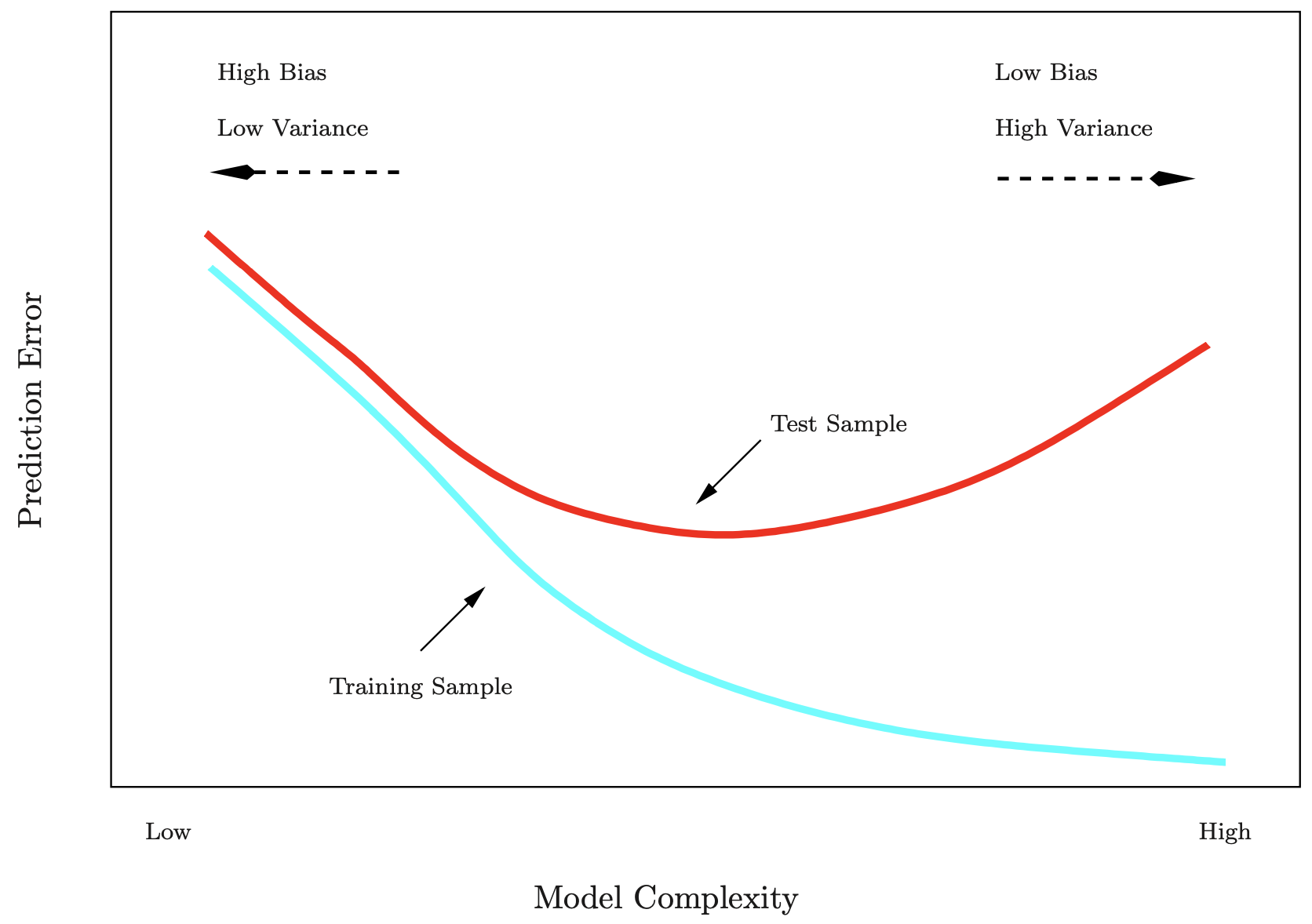
Die Trainingsfehlerrate (\(MSE_{train}\)) unterscheidet sich oft erheblich von der Testfehlerrate (\(MSE_{test}\)). Insbesondere bei sehr flexiblen Modellen kann die Trainingsfehlerrate die Testfehlerrate drastisch unterschätzen.
Bildquelle: James et al. (2013): Introduction to Statistical Learning

Bildquelle: James et al. (2013): Introduction to Statistical Learning
MSE einer einzelnen (schwarz) vs mehrfachen Aufteilung

Bildquelle: James et al. (2013): Introduction to Statistical Learning

Bildquelle: James et al. (2013): Introduction to Statistical Learning
\[CV_{(k)} = \frac{1}{k}\sum_{i=1}^{k}MSE_i\]

Klassifikationsprobleme treten häufig auf, möglicherweise sogar häufiger als Regressionsprobleme.

Bildquelle: http://www.saedsayad.com/logistic_regression.htm
Von Wahrscheinlichkeiten zu den log-transformierten Chancen
| Pi | 0.001 | 0.1 | 0.3 | 0.4 | 0.5 | 0.6 | 0.7 | 0.9 | 0.999 |
|---|---|---|---|---|---|---|---|---|---|
| 1-Pi | 0.999 | 0.9 | 0.7 | 0.6 | 0.5 | 0.4 | 0.3 | 0.1 | 0.001 |
| Oi | 0.001 | 0.11 | 0.43 | 0.67 | 1 | 1.5 | 2.33 | 9 | 999 |
| Ln(Oi) | -6.91 | -2.2 | -0.85 | -0.41 | 0 | 0.41 | 0.85 | 2.20 | 6.91 |
In der logistischen Regression werden die Log Odds als lineare Funktion der erklärenden Variablen modelliert:
\[ln(O_i) = ln(\frac{P_i}{1-P_i}) = g(x_i) \text{ wobei }g(x_i) = \alpha + \beta_1 X_1 + \beta_2 X_2 + ...\beta_p X_p\]
\[O_i = \frac{P_i}{1-P_i} = e^{g(x_i)} \Rightarrow P_i = \frac{e^{g(x_i)}}{1+e^{g(x_i)}}\]
Pi ist immer zwischen 0 und 1

Können wir anhand der Flossenlänge die Artzugehörigkeit (‘Adélie’ oder ‘Gentoo’) bestimmen?







Call:
glm(formula = species ~ flipper_length_mm, family = "binomial",
data = penguins_sub)
Deviance Residuals:
Min 1Q Median 3Q Max
-2.60000 -0.00584 -0.00023 0.01513 1.91868
Coefficients:
Estimate Std. Error z value Pr(>|z|)
(Intercept) -147.0506 37.3471 -3.937 8.24e-05 ***
flipper_length_mm 0.7162 0.1805 3.967 7.27e-05 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
(Dispersion parameter for binomial family taken to be 1)
Null deviance: 364.612 on 264 degrees of freedom
Residual deviance: 20.887 on 263 degrees of freedom
AIC: 24.887
Number of Fisher Scoring iterations: 10Hier interessieren uns jetzt primär nur die Koeffizienten.
Um zu wissen, welche Arten bzw. Faktorstufes R intern als 1 bzw. 0 kodiert, folgenden Code anwenden:
→ Gentoo wird mit 1 kodiert, sprich für diese Art wird die Wahrscheinlichkeit berechnet.
\[E[Y_i]= P_i = \mu_i = \frac{e^{g(x_i)}}{1+e^{g(x_i)}}= \frac{e^{-147.1}*e^{0.716*\text{flipper_length_mm}}}{1+e^{-147.1}*e^{0.716*\text{flipper_length_mm}}}\]
→ Die Wahrscheinlichkeit, dass der Pinguin der Art Gentoo zugehört, steigt von < 0.1% auf > 99% mit zunehmender Flossenlänge im Bereich von 195-215mm.
penguins_sub |>
mutate(
species_num = case_when(species == "Gentoo" ~ 1, .default = 0),
pred = predict(log_reg, type = "response")) |>
ggplot(aes(x = flipper_length_mm, y = pred)) +
geom_point(aes(y = species_num)) +
geom_line() +
geom_point(data = data.frame(fl = fl, pred_fl = pred_fl),
mapping = aes(x = fl, y = pred_fl), colour = "red", size = 2.5) +
geom_hline(yintercept = 0.5, linetype = 2) +
ylim(0,1) +
ylab("Probability") +
theme_light()Wie oft liegt das Modell mit der vorhergesagten Art richtig und wie oft falsch?
| Adelie_obs | Gentoo_obs | |
|---|---|---|
| Adelie_pred | 144 | 1 |
| Gentoo_pred | 2 | 118 |
Wie wäre unsere Modellperformance, wenn wir 50% der Daten als Testdaten zurückhalten?
| Adelie_obs | Gentoo_obs | |
|---|---|---|
| Adelie_pred | 69 | 0 |
| Gentoo_pred | 1 | 62 |
- Die Modellgenauigkeit (‘accuracy’) ist hier sogar noch höher: (69+62)/132*100 = 99.2 %!
![]()


Dann testen Sie doch Ihr Wissen in folgendem Abschlussquiz…
![]()

Bei weiteren Fragen: saskia.otto(at)uni-hamburg.de

Diese Arbeit is lizenziert unter einer Creative Commons Attribution-ShareAlike 4.0 International License mit Ausnahme der entliehenen und mit Quellenangabe versehenen Abbildungen.
Kurswebseite: Data Science 3