

Resampling Techniken
DS3 - Explorative Datenanalyse & Data Mining
Saskia Otto
Universität Hamburg, IMF
Wintersemester 2023/2024
Lernziele
![]()
Am Ende dieser VL- und Übungseinheit werden Sie
- das sog. Bootstrap-Verfahrens auf reale Datensätze anwenden können, um Konfidenzintervalle für Punktschätzungen zu erstellen.
- wissen, wie das Bootstrap-Verfahren funktioniert, insbesondere wie Stichproben MIT Zurücklegen aus einer vorhandenen Stichprobe gezogen werden.
- das Konzepts der Permutation von Beobachtungen kennen und wissen, wie Permutationstests auf verschiedene Arten von Daten anzuwenden sind, um Hypothesen über Populationsparameter zu testen.
- in der Lage sein, die Ergebnisse von Permutationstests zu interpretieren und zu verstehen, wie statistische Signifikanz in diesem Kontext definiert ist.
- Permutationstests mit anderen Hypothesentestverfahren zu vergleichen und die Vor- und Nachteile jeder Methode zu verstehen.
Unser Thema heute

Grafik von Nina Garman (Pixabay)
Schätzverfahren und Hypothesentest
Beide Verfahren der inferenziellen Statistik beruhen auf unterschiedlichen Stichprobenverteilung:
- Schätzverfahren
- Verwendet die Stichprobenverteilung einer Schätzung.
- Sie wird verwendet, um Standardfehler und Konfidenzintervalle zu erhalten.
- Die meisten Methoden gehen davon aus, dass die Stichprobenverteilung annähernd normal ist.
- Hypothesentest
- Verwendet die Null-Stichprobenverteilung (oder Nullverteilung): die Wahrscheinlichkeitsverteilung einer Teststatistik, wenn die Nullhypothese wahr ist.
- Häufig werden die t-, F-, \chi^2- und Normalverteilungen zur Annäherung an die Nullverteilungen verwendet, aus denen die P-Werte berechnet werden.
Schätzverfahren und Hypothesentest
Frage:
Was ist zu tun, wenn die Annahmen der besten verfügbaren Methode verletzt werden und wir nicht auf parametrische Tests und allgemeine lineare Modelle zurückgreifen können?
Antwort: Computerintensive Methoden
Ein Ansatz, bei dem die Leistungsfähigkeit des Computers genutzt wird, um eine Stichprobenverteilung zu erstellen:
- Schätzverfahren: Bootstrapping
- Hypothesentest: Permutationstests
Bootstrapping
Punkt- und Intervallschätzung
Zur Erinnerung
- Auf Basis einer Zufallsprobe wird der Bereich geschätzt, wo der gesuchte Populationsparameter liegen könnte.
- Ausgangspunkt ist immer eine Punktschätzung → dann wird ein (symmetrisches) Intervall bestimmt, das Konfidenzintervall KI
- Konfidenz wird als wiederholte Stichprobe interpretiert.
- Kann für jeden Parameter (z.B. Mittelwert, Varianz, ..) berechnet werden.
- Die Breite des Intervalls hängt ab
- vom Stichprobenumfang, der Varianz der Stichprobe und
- der festgelegten Wahrscheinlichkeit (= Konfidenzniveau) → üblich: 90%, 95%, 99%
KI vom Mittelwert wenn n > 30
KI_{95\%} = 1.96\cdot\sqrt{\frac{\sigma^2}{n}}
KI vom Mittelwert wenn n < 30
KI_{95\%} = t_{(\alpha/2,df)}\cdot\sqrt{\frac{s^2}{n}}
Intervallschätzung | 1
Zur Erinnerung ein Beispiel aus DS2
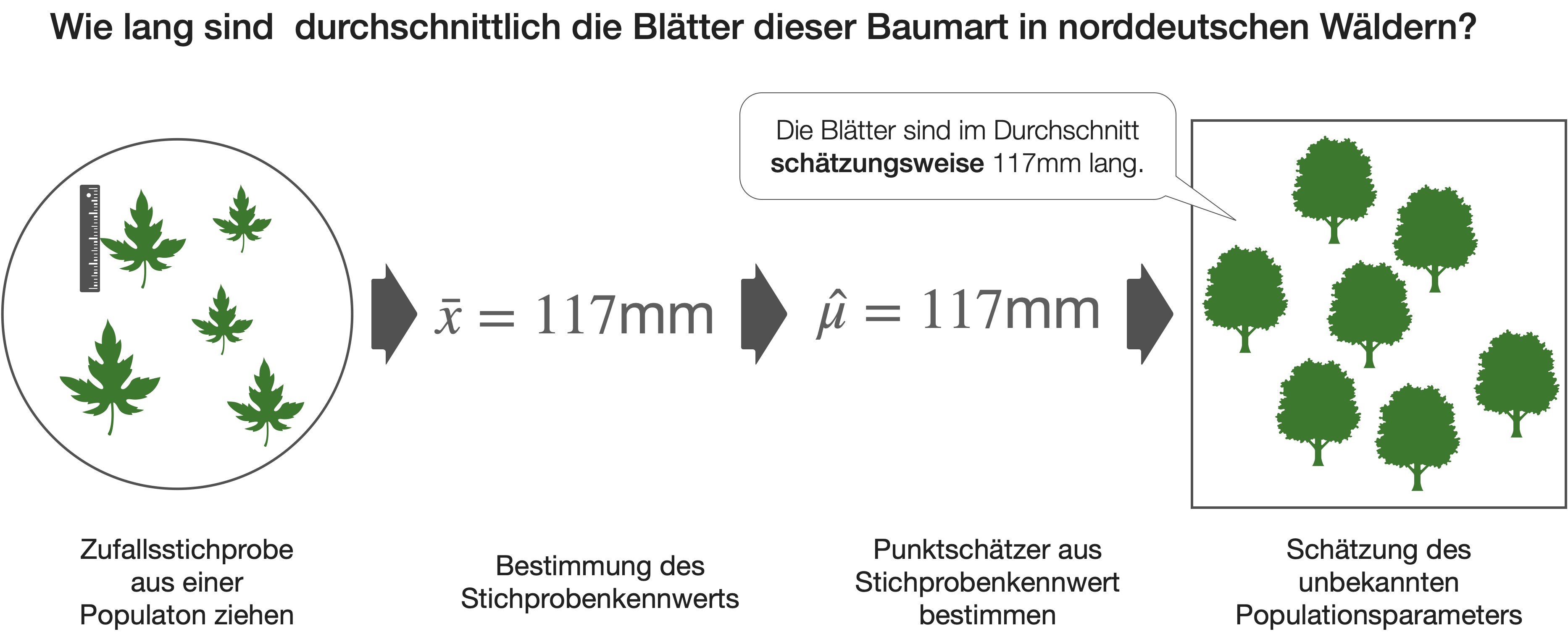
- Wie präzise ist diese Schätzung von 117mm?
- Kann es denn sein, dass der wahre Populationsmittelwert auch 100mm oder 140mm ist und wir einfach Pech mit der Probe hatten?
- In welchem Bereich liegt der wahre Mittelwert höchstwahrscheinlich?
Intervallschätzung | 2
Zur Erinnerung ein Beispiel aus DS2


Intervallschätzung | 3
Parametrisches KI basierend auf der t-Verteilung
- → Wir sind uns zu 95% sicher, dass der wahre Mittelwert im Bereich 111.4 - 112.4 liegt (111.9 ± 0.5).
- Unter der Annahme, dass die Verteilung einer symmetrischen t-Verteilung folgt!
Bootstrapping oder ‘Schnürsenkelmethode’
Nicht-parametrische KIs mittels Bootstrapping berechnen
- Nützliche Methode, wenn Verteilung nicht einer theoretischen Verteilung entspricht (z.B. der z- und t-Verteilung).
- Prinzip:
- Die Verteilung der Stichprobe dient als Grundlage einer ‘Pseudopopulation’.
- Aus dieser ‘Pseudopopulation’ entnehmen wir wiederholt Stichproben und berechnen jeweils die Kenngröße.
- Da der Stichprobenumfang meist limitiert ist, werden Proben mit Rücklegen entnommen (somit unendlich groß).
- Aus der Grundgesamtheit der Kennwerte (z.B. der Mittelwerte) berechnen wir das untere und obere 2.5 % (oder 0.5 %,..) Quantil als unsere Konfidenzgrenze.
- Ergebnisse sind recht zuverlässig und mit Computern schnell umzusetzen.
- Die KI sind nicht unbedingt symmetrisch.
Bootstrapping | Berechnung

→ Wir sind uns nun zu 95% sicher, dass der wahre Mittelwert im Bereich 111.4 - 112.4 liegt.
Bootstrapping | Vergleich


Bootstrapping | ‘boot’ Paket

Statt der manuellen Berechnung können wir die boot() Funktion aus dem gleichnamigen R Paket verwenden:
boot() Funktion im 'boot' Paket
- Die Syntax der Funktion ist recht einfach:
boot(data, statistic, R)datarepräsentiert den Vektor aus dem die Stichproben gezogen werden.statisticsteht für die Funktion, die pro Iteration angewendet werden soll, in unserem Fall für die Berechnung des Mittelwerts.Rdefiniert die Anzahl der Wiederholungsstichproben.
Interpretation | ‘boot’ Paket

Konfidenzintervalle | ‘boot’ Paket

BOOTSTRAP CONFIDENCE INTERVAL CALCULATIONS
Based on 1000 bootstrap replicates
CALL :
boot.ci(boot.out = boot_results)
Intervals :
Level Normal Basic
95% (111.4, 112.4 ) (111.4, 112.3 )
Level Percentile BCa
95% (111.4, 112.4 ) (111.5, 112.5 )
Calculations and Intervals on Original ScaleNormal: parametrisches KI, das auf dem Standardfehler des Mittelwerts und dem Stichprobenumfang basiertPercentile: das sog. Bootstrap-Perzentil-Intervall = Quantil aus den Bootstrap-SchätzungenBCa: angepasstes Bootstrap-Perzentil-Intervall
Schätzung von KI für Regressionsparameter
Wenn Annahmen nicht erfüllt sind
Code
df$res <- residuals(mod_lm)
df$fit <- fitted(mod_lm)
p1 <- ggplot(df, aes(y)) +
geom_histogram(bins = 10, fill = "grey50", colour = "grey10") +
ggtitle("Histogramm von Y")
p2 <- ggplot(df, aes(x, y)) + geom_point() +
geom_smooth(method = "lm", se = TRUE) +
ggtitle("Beziehung Y ~ X")
p3 <- ggplot(df, aes(res)) +
geom_histogram(bins = 10, fill = "grey50", colour = "grey10") +
ggtitle("Histogramm der Residuen")
p4 <- ggplot(df, aes(fit, res)) + geom_point() +
geom_hline(yintercept = 0) +
ggtitle("Residuen vs. fitted")
gridExtra::grid.arrange(p1, p2, p3, p4)
Estimate Std. Error t value Pr(>|t|)
(Intercept) -55.960944 22.550127 -2.481624 1.761662e-02
x 8.135234 1.464779 5.553899 2.329342e-06- Der Steigungsparameter b der Stichprobe als Punktschätzer für die Population beträgt 8.1.
- Gegeben, das die Daten normalverteilt und varianzhomogen sind → können wir uns zu 95 % sicher sein, dass bei einer Wiederholung der Datenerhebung die Regressionssteigung für diese neuen Daten zwischen 5.3 und 11.0 liegen würde.
Schätzung von KI für Regressionsparameter
Bootstrapping
set.seed(32)
it <- 10000
boot_b <- numeric(it)
for (i in 1:it){
n <- nrow(df)
indices <- sample(1:n, replace = T)
y_sample <- df$y[indices]
x_sample <- df$x[indices]
model <- lm(y_sample ~ x_sample)
boot_b[i] <- coef(model)[2]
}
quantile(boot_b,c(0.025,0.975)) 2.5% 97.5%
4.663306 11.757071 → Der Konfidenzbereich liegt nun zwischen 4.7 und 11.8.

Permutationstest

Permutationstests | 1
- Ähnlich wie Bootstrapping, mit dem Unterschied, dass beim Permutationstest Stichproben OHNE Zurücklegen gezogen werden (d.h., wenn ein Wert ausgewählt wurde, kann er nicht erneut ausgewählt werden, so dass kein Wert ein Duplikat sein kann). Dabei werden die Werte einfach neu gemischt.
- Im Falle einer univariaten Statistik (z. B. Mittelwert) ändert sich dadurch nichts.
- Bei zwei oder mehr Variablen bzw. Gruppen ändert sich durch die Umordnung einer Variablen allerdings die Teststatistik, z. B. die Korrelation oder Regression,
Permutationstests | 2
Prinzip der Permutation
Man generiert sich mit Hilfe des Computers seine eigene Zufalls- oder Nullverteilung und prüft dann, ob sich die gefundene Verteilung von dieser Zufallsverteilung unterscheidet.
- Annahme: Die gemessenen Werte sind die Werte, die vorkommen können.
- Frage: Mit welcher Häufigkeit tritt die gefundene Verteilung bei den oben generierten Zufallsverteilungen auf?
- Signifikanz: Die Häufigkeit, mit der die gefundene Verteilung bei den durch zufällige Verteilung generierten Gruppen auftritt, entspricht der Irrtumswahrscheinlichkeit.
Demonstration aus dem Internet
Webseite: https://www.jwilber.me/permutationtest/
Was ist besser?
Nicht-parametrische Tests
- Rang-basierte Tests, wie z. B. der Mann-Whitney-U-Test für zwei Stichproben, stellen Permutationstests dar:
- Daten werden durch Ränge ersetzt und diese permutiert, um eine Nullverteilung zu erzeugen.
- Die genaue Wahrscheinlichkeitsverteilung der U-Statistik ist bekannt.
- Durch die Ersetzung der Daten durch ihre Ränge gehen jedoch Informationen verloren.
Permutationstest
- Statt Daten durch die Ränge zu ersetzen können sie direkt permutiert werden.
- Permutationstests haben bei kleinem Stichprobenumfang eine geringere Teststärke als parametrische Tests, aber sie sind aussagekräftiger als z.B. der Mann-Whitney-U-Test.
- Bei großem Stichprobenumfang haben sie eine ähnliche Teststärke wie parametrische Tests.
Annahmen von Permutationstests
- Zufällige Stichproben.
- Bei Gruppenvergleichen der Mittelwert oder Mediane sollte die Verteilung der Variablen in jeder Population die gleiche Form haben.
- Es gilt jedoch: Permutationstests sind robust gegenüber Abweichungen von der Gleichverteilungsannahme, wenn die Stichprobengröße groß ist (stärker als beim Mann-Whitney-U-Test).
Beispiel 1: Zwei-Stichproben-Vergleich
Schneckenrennen
Unterscheiden sich zwei Schneckenarten in ihrer Laufgeschwindigkeit (in m/Stunde)?

Zwei-Stichproben-Vergleich | Daten
A <- c(4,2,7,9,2,8)
B <- c(7,10,9,5,8,11)
Rennen <- data.frame(
Art = c(rep("A", 6), rep("B", 6)),
Geschw = c(A,B)
)
Rennen |>
group_by(Art) |>
summarise(
Mittelwert = mean(Geschw),
Standardabweichung = sd(Geschw)
)# A tibble: 2 × 3
Art Mittelwert Standardabweichung
<chr> <dbl> <dbl>
1 A 5.33 3.08
2 B 8.33 2.16
Zwei-Stichproben-Vergleich | t-Test
Welch Two Sample t-test
data: Geschw by Art
t = -1.9547, df = 8.9658, p-value = 0.08247
alternative hypothesis: true difference in means between group A and group B is not equal to 0
95 percent confidence interval:
-6.473935 0.473935
sample estimates:
mean in group A mean in group B
5.333333 8.333333
Welch Two Sample t-test
data: Geschw by Art
t = -1.9547, df = 8.9658, p-value = 0.04123
alternative hypothesis: true difference in means between group A and group B is less than 0
95 percent confidence interval:
-Inf -0.1853508
sample estimates:
mean in group A mean in group B
5.333333 8.333333 Zwei-Stichproben-Vergleich | Permutation 1
Permutationstest - einmalig
Differenz beider Mittelwerte als Teststatistik
[1] 3Zwei-Stichproben-Vergleich | Permutation 2
Permutationstest - 10000fach
Zwei-Stichproben-Vergleich | Permutation 2
Permutationstest - 10000fach
set.seed(321)
it <- 10000 # 10000 Iterationen
mystats <- numeric(it)
for (i in 1:it){
# Schleifenkörper: wir kopieren den Code von eben
Rennen_neu <- Rennen
Rennen_neu$Geschw <- sample(Rennen_neu$Geschw)
mystats[i] <- mean(Rennen_neu$Geschw[Rennen_neu$Art == "B"]) -
mean(Rennen_neu$Geschw[Rennen_neu$Art == "A"])
}Zwei-Stichproben-Vergleich | Verteilung
Zwei-Stichproben-Vergleich | Interpretation
- Bei 475 von 10000 Permutationen sind die Differenzwerte zwischen den beiden Mittelwerten gleich 3 oder größer (also als die Teststatistik der Originaldaten).
- Damit ist die Wahrscheinlichkeit, dass eine Differenz von ≥ 3 auftritt = 4.8 %.
- Für die 2-seitige Hypothese vergleichen wir die absoluten Werte!
- D.h., die Wahrscheinlichkeit eine solche Teststatistik wie die beobachtete zu erhalten - wenn die 1- bzw. 2-seitige H0 richtig ist - liegt bei 4.8 bzw. 9.4 %. = die Wahrscheinlichkeit, dass die beobachteten Daten unter der Annahme der H0 auftreten, liegt bei <5 bzw. < 10%.
Beispiel 2: Korrelation
y x
1 0.069360916 0.3926972
2 0.817775199 0.8138806
3 0.942621732 0.3762485
4 0.269381876 0.3808122
5 0.169348123 0.2649184
6 0.033895622 0.4393343
7 0.178785004 0.4576072
8 0.641665366 0.5407075
9 0.022877743 0.6656798
10 0.008324827 0.1126989Korrelation | Permutation 1
Einmalig
y x resample_y
1 0.069360916 0.3926972 0.178785004
2 0.817775199 0.8138806 0.641665366
3 0.942621732 0.3762485 0.269381876
4 0.269381876 0.3808122 0.069360916
5 0.169348123 0.2649184 0.817775199
6 0.033895622 0.4393343 0.942621732
7 0.178785004 0.4576072 0.033895622
8 0.641665366 0.5407075 0.008324827
9 0.022877743 0.6656798 0.169348123
10 0.008324827 0.1126989 0.022877743[1] -0.01818182Korrelation | Permutation 2
Korrelation | Permutation 2
Korrelation | Permutation 3
[1] 5394[1] 0.5394- INTERPRETATION: die Wahrscheinlichkeit, dass die beobachteten Daten unter der Annahme der H0 auftreten, liegt bei 54 %. Wir können daher die H0 nicht ablehnen, es gibt keine signifikante Korrelation.
Korrelationsbeispiel | ‘coin’ Paket
Es gibt das Paket ‘coin’ , das Permutationstests für verschiedene Tests durchführt. Am besten die Vignette anschauen (vignette("coin", package = "coin"):
Asymptotic Spearman Correlation Test
data: y by x
Z = 0.67273, p-value = 0.5011
alternative hypothesis: true rho is not equal to 0Beispiel 3: ANCOVA
Schnabellänge bei Adélie Pinguinen (Insel Dream)

Call:
lm(formula = body_mass_g ~ flipper_length_mm + sex, data = penguin_subs)
Residuals:
Min 1Q Median 3Q Max
-524.73 -186.57 6.13 128.46 716.22
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 696.634 1112.928 0.626 0.5341
flipper_length_mm 14.095 5.918 2.382 0.0209 *
sexmale 643.629 76.015 8.467 2.34e-11 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 267.3 on 52 degrees of freedom
Multiple R-squared: 0.6585, Adjusted R-squared: 0.6454
F-statistic: 50.13 on 2 and 52 DF, p-value: 7.387e-13ANCOVA | Permutation 1

Wir extrahieren zuerst die t- und F-Statistiken aus der summary() bzw. drop1() Funktion.
ANCOVA | Permutation 2

ANCOVA | Permutation 2

Wir schreiben die Schleife
set.seed(123)
it <- 10000 # 10000 Iterationen
mystats_flipper <- numeric(it)
mystats_sex <- numeric(it)
for (i in 1:it){
# Schleifenkörper
df <- penguin_subs
df$body_mass_g_shuffled <- sample(df$body_mass_g)
mod <- lm(body_mass_g_shuffled ~ flipper_length_mm + sex, df)
mystats_flipper[i] <- summary(mod)$coefficients[2, "t value"]
mystats_sex[i] <- drop1(mod, test = "F")$`F value`[3]
}ANCOVA | Permutation 3

Verteilung der Teststatistiken
INTERPRETATION: die Wahrscheinlichkeit, dass die beobachteten Daten unter der Annahme der H0 auftreten, liegt bei beiden Kovariaten unter 5 %. Wir können daher heweils die H0 ablehnen, es gibt einen signifikanten Effekt beider Variablen.
Fragen..??

Total konfus?

Buchkapitel zum Nachlesen
- The R Book von M.J. Crawley:
- Kapitel 8.12 Bootstrap
- Wikipedia zu Bootstrap (https://en.wikipedia.org/wiki/Bootstrapping_(statistics))
- Dokumentation des ‘coin’ Pakets (https://cran.r-project.org/web/packages/coin/vignettes/coin.pdf)
Übungsaufgabe
 r
r
Übung
![]()
- Aufgaben im Übungsskript DS3_06_Uebungen_Resampling.Rmd mit abschließendem Quiz in Moodle (zuhause VOR der Übung)
- Gegebenenfalls Anwendung der Resampling-Techniken in Ihrer Fallstudie.
- WÄHREND der Übungsstunde: Besprechung der Lösung und Fragenrunde
R Notebook und Datensätze sind im Moodlekurs (Woche 6) zu finden.
Total gelangweilt?

Dann testen Sie doch Ihr Wissen in folgendem Abschlussquiz…
Abschlussquiz
![]()

Bei weiteren Fragen: saskia.otto(at)uni-hamburg.de

Diese Arbeit is lizenziert unter einer Creative Commons Attribution-ShareAlike 4.0 International License mit Ausnahme der entliehenen und mit Quellenangabe versehenen Abbildungen.
Kurswebseite: Data Science 3




