'data.frame': 153 obs. of 6 variables:
$ Ozone : int 41 36 12 18 NA 28 23 19 8 NA ...
$ Solar.R: int 190 118 149 313 NA NA 299 99 19 194 ...
$ Wind : num 7.4 8 12.6 11.5 14.3 14.9 8.6 13.8 20.1 8.6 ...
$ Temp : int 67 72 74 62 56 66 65 59 61 69 ...
$ Month : int 5 5 5 5 5 5 5 5 5 5 ...
$ Day : int 1 2 3 4 5 6 7 8 9 10 ...
Multiple lineare Regression und Modellselektion
DS3 - Explorative Datenanalyse & Data Mining
Saskia Otto
Universität Hamburg, IMF
Wintersemester 2023/2024
Lernziele
![]()
Am Ende dieser VL- und Übungseinheit werden Sie
- die Unterschiede zwischen einer bivariaten und einer multiplen Regression kennen.
- wissen, warum und wie auf (Multi-)Kollinearität vorab geprüft werden muss.
- mehrere Kriterien für die Modellauswahl kennen und im Rahmen einer ‘backward selection’ anwenden können.
- wissen, was mit Ockhams Rasiermesser gemeint ist.
Regression mit
≥ 2 Kovariaten

Multiple Regression | Ziel
- Identifizierung des Einflusses mehrerer unabhängiger Kovariaten X_p auf die Antwortvariable unter Berücksichtigung der Beziehungen zwischen den Kovariaten.
- Vorhersage von Y-Werten aus den Werten mehrerer X-Variablen.
- Bestimmung der einflussreichsten Parameter aus mehreren unabhängigen Variablen → empirische Variablenauswahl.
Multiple Regression | Gleichung
Die Grundsätze einer einfachen, bivariaten Regressionsanalyse lassen sich auf zwei oder mehr kontinuierliche Variablen ausweiten:

Faustregel: nicht mehr als 10 erklärende Variablen
Multiple Regression | Neu
- Zusätzliche Annahmen / Datenexplorationen
- Schätzung der Parameter (Achsenabschnitt \alpha & partielle Regressionskoeffizienten \beta_p)
- Signifikanztest
- (Multiples) R^2 vs. korrigiertes R^2
- Modellauswahl
Zusätzliche Annahmen
- Wie bei der einfachen (bivariaten) Regression
- Normalität
- Homogenität
- Unabhängigkeit
- Linearität
- NEU
- Anzahl der Kovariaten < Anzahl der Beobachtungen (p < n)
- Keine lineare Beziehung zwischen den Kovariaten (keine Kollinearität / Multikollinearität)
Problem der Kollinearität
- Kleine Änderungen in den Daten → extreme Änderungen in den Regressionskoeffizienten
- Schätzungen zwischen verschiedenen Stichproben derselben Population können stark voneinander abweichen → hohe Standardfehler der geschätzten Parameter
- Schätzungen von \beta’s und Signifikanztests unzuverlässig.
- Shiny Demo
Prüfen auf Kollinearität

- Paarweise Korrelation und Streudiagramme →
cor()undpairs() - Berechnung des VIF-Werts für jede Kovariate →
car::vif()
VIF-Wert
VIF_i = \frac{1}{TOL_i} = \frac{1}{1-R_i^2}
- R^2 hier aus Modell, welches alle Kovariaten enthält außer der Kovariate, für die der VIF-Wert berechnet wird.
- Faustregel: VIF > 3 ist problematisch (einige sagen VIF>10, hängt auch von der Anzahl an Kovariaten ab)
- Wenn Kovariaten einen VIF > 3 haben,
- → die Kovariate mit dem höchsten VIF entfernen (oder nach biologischem Wissen/Interesse entscheiden)
- → die VIFs für die verbleibenden Kovariaten neu berechnen
- → fortfahren, bis alle Kovariaten VIFs < 3 haben.
Weitere Datenexploration
- Unabhängigkeit (posteriori)
- Visualisieren Sie im Streudiagramm die Residuen gegen die einzelnen erklärenden Variablen (auch diejenigen, die aus dem endgültigen Modell ausgeschlossen wurden) und suchen Sie nach einem Muster.
- Interaktionen (a priori)
- Darstellung von Y gegen X_1, X_2,… in Abhängigkeit von einer anderen Kovariate
- Nützliche Funktion
coplot()
Parameterschätzung
- Achsenabschnitt (intercept) \alpha
- Wert von Y, wenn alle X-Werte gleich Null sind → weniger wichtig
- Partielle Regressionskoeffizienten (slopes) \beta_p (\beta_1, \beta_2,.. )
- Effekt einer Erhöhung von X_p um 1 Einheit, wobei alle anderen Kovariaten oder Prädiktoren (X) konstant bleiben
- Regressionskoeffizienten der Stichprobe (a, b_p) werden mit OLS Methode geschätzt.
- \hat{y}_i = a + b_1 x_{1,i} + b_2 x_{2,i} + ...
\hat{y}_i = \text{vorhergesagter Y-Wert für jede Kombination von X-Werten}
Signifikanztests | Partielle Koeffizienten
- H_0:\beta_p = 0
- Getrennte t-Tests für jeden partiellen Regressionskoeffizienten im Modell
- Partielle t-Tests verwenden: t_p = \frac{b_p}{s.e.(b_p)}
- Vergleich mit t-Verteilung mit N-2 Freiheitsgraden
- H_0 verwerfen, wenn p < 0.05
Anpassung des Bestimmtheitsmaß


Lösung: korrigiertes Bestimmtheitsmaß

Modellauswahl

Ausgangssituation
- Wir wollen das optimale Modell finden, das die Parameter identifiziert, die die gesammelten Daten am besten erklären.
- Aber welches von allen möglichen Modellen passt am besten zu den Daten?
- Viele mögliche Modelle
- für p Prädiktoren, # mögliche Modelle = 2^p
- → 6 Prädiktoren, 64 mögliche Modelle
- → 10 Prädiktoren, 1024 mögliche Modelle
- Effiziente Suchverfahren erforderlich
- Verschiedene Ansätze und unterschiedliche Kriterien für die Auswahl des “besten” Modells
- Sparsamkeitsprinzip (principle of parsimony)
Sparsamkeitsprinzip
Oder Ockhams Rasiermesser
Das Prinzip der Parsimonie wird dem englischen Philosophen William von Ockham aus dem frühen 14. Jahrhundert zugeschrieben, der darauf bestand, dass bei einer Reihe gleichwertiger Erklärungen für ein bestimmtes Phänomen die einfachste Erklärung die richtige ist. Sie wird Ockhams Rasiermesser genannt, weil er seine Erklärungen auf das absolute Minimum ‘rasierte’.

Video – What is Ockham’s Razor?
Kriterien der Modellauswahl
- Hypothesentests (z.B. der F-Test)
- Korrigiertes R^2 → je höher desto besser → absolutes Maß
- Akaike’s Informationskriterium (AIC) → je niedriger desto besser → relatives Maß
- Faustregel: Wenn der Unterschied zwischen 2 Modellen im AIC ≤ 2 beträgt, sollte das einfachere Modell gewählt werden.
- Berücksichtigt das Maß der Anpassungsgüte und fügt einen so genannten Strafterm für die Anzahl der unabhängigen Parameter im Modell hinzu.

- Bayesian Informationskriterium (BIC)
- Gibt der Anzahl an Kovariaten mehr Gewicht
- Empfehlenswert, wenn p > 7
‘All subsets selection’
- Vergleich ausgewählter Teilmengen von Modellen und Wahl des optimalen Modells mittels AIC oder BIC.
- Kann zu einer großen Anzahl von zu vergleichenden Modellen führen.
- Viele Iterationen und zeitaufwändig, wenn p > 5.
- Die Anzahl kann jedoch reduziert werden, indem a priori einige Teilmengen auf der Grundlage ökologischer Kenntnisse ausgewählt werden.
‘Stepwise selection’
- Untersuchung einer Teilmenge möglicher Modelle auf der Grundlage eines bestimmten Kriteriums.
- Sog. ‘forward selection’
- Sog. ‘backward selection’:
- Startet mit allen Prädiktoren (volles Modell) und entfernt eine Variable bzw. einen Term nach dem anderen bis R_{adj}^2 sinkt bzw. der AIC steigt.
- ’stepwise selection’:
- kombiniert vorwärts und rückwärts gerichtete Auswahl
‘Backward selection’ | 1
Wir starten mit allen Variablen und Interaktionen
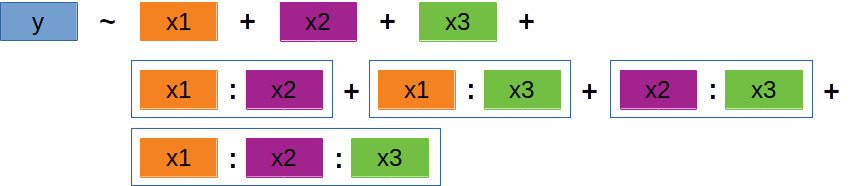
→ Höchstwahrscheinlich viel zu viele Variablen und Interaktionen im Modell.
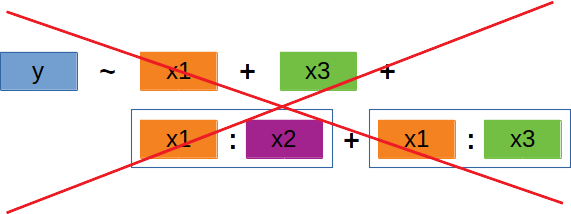
‘Backward selection’ | 2
Schrittweises Entfernen
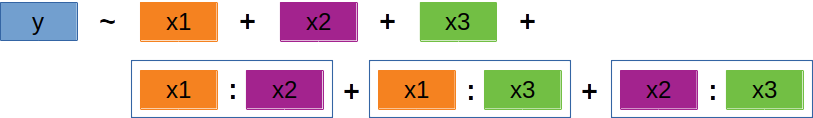

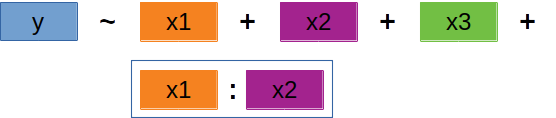

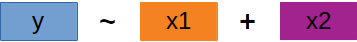
Modell sollte möglichst simpel sein → erst die komplexeren, dann die weniger komplexen Terme entfernen.
‘Backward selection’ | 3
Schrittweises Entfernen
- Also erst Entfernen komplexer Interaktionsterme (z.B. 4er- vor 3er-Interaktion).
- Einzelne Variablen werden (meistens) erst ganz zum Schluss entfernt.
- Solange eine Variable in einer Interaktion auftritt, darf die Variable selbst nicht entfernt werden.
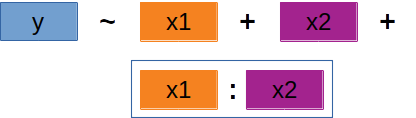
‘Backward selection’ | 4
Schrittweises Entfernen
- Es ist durchaus möglich, einzelne Variablen zu entfernen, bevor eine Interaktion entfernt wird → solange diese Variablen nicht Teil der Interaktion sind!
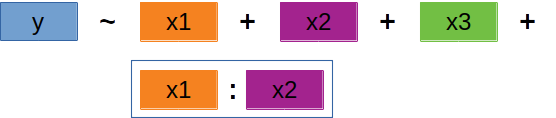

Modellvergleiche in R
- 2 verschachtelte Modelle können mit einer F-Statistik vergleichen werden.
- →
anova(model1, model2)oderdrop1(model, test = "F") - 2 Modelle werden als verschachtelt bezeichnet, wenn die erklärenden Variablen in einem Modell (= ‘reduziertes Modell’) eine Teilmenge derjenigen im anderen Modell (= ‘volles Modell’) sind.
- →
- AIC Vergleich →
AIC(model1, model2, model3,..)- Vergleich auch von Modellen mit völlig unterschiedlichen erklärenden Variablen möglich
- ABER: Y muss immer gleich sein (keine unterschiedliche Transformation und auch nicht weniger/mehr Werte durch NAs).
- Wenn die Modelle den gleichen/ähnlichen Wert haben (AIC-Unterschied < 2) → wählt das einfachere Modell.
Beispiel in R

Beispiel in R
‘airquality’-Datensatz
- Enthält Luftqualitätsmessungen in New York zwischen Mai und September 1973.
- Fragestellung: Welchen Effekt haben die Sonneneinstrahlung (
Solar.R) und die Wind- (Wind) und Temperaturbedingungen (Temp) auf die Ozonwerte (Ozone)?
Datenexploration | Verteilung und NAs
Die Ozonewerte sind stark rechtsschief und eine erste Modellselektion zeigte, dass Ozon transformiert werden muss (4te-Wurzel).

Datenexploration | Beziehungen
![]()
Datenexploration | Kolliniarität
Die vif() Funktion aus dem 'car' Paket
Solar.R Wind Temp
1.095253 1.329070 1.431367 - Keine Kovariate zeigt einen VIF-Wert > 3.
- Es können daher alle im Modell bleiben.
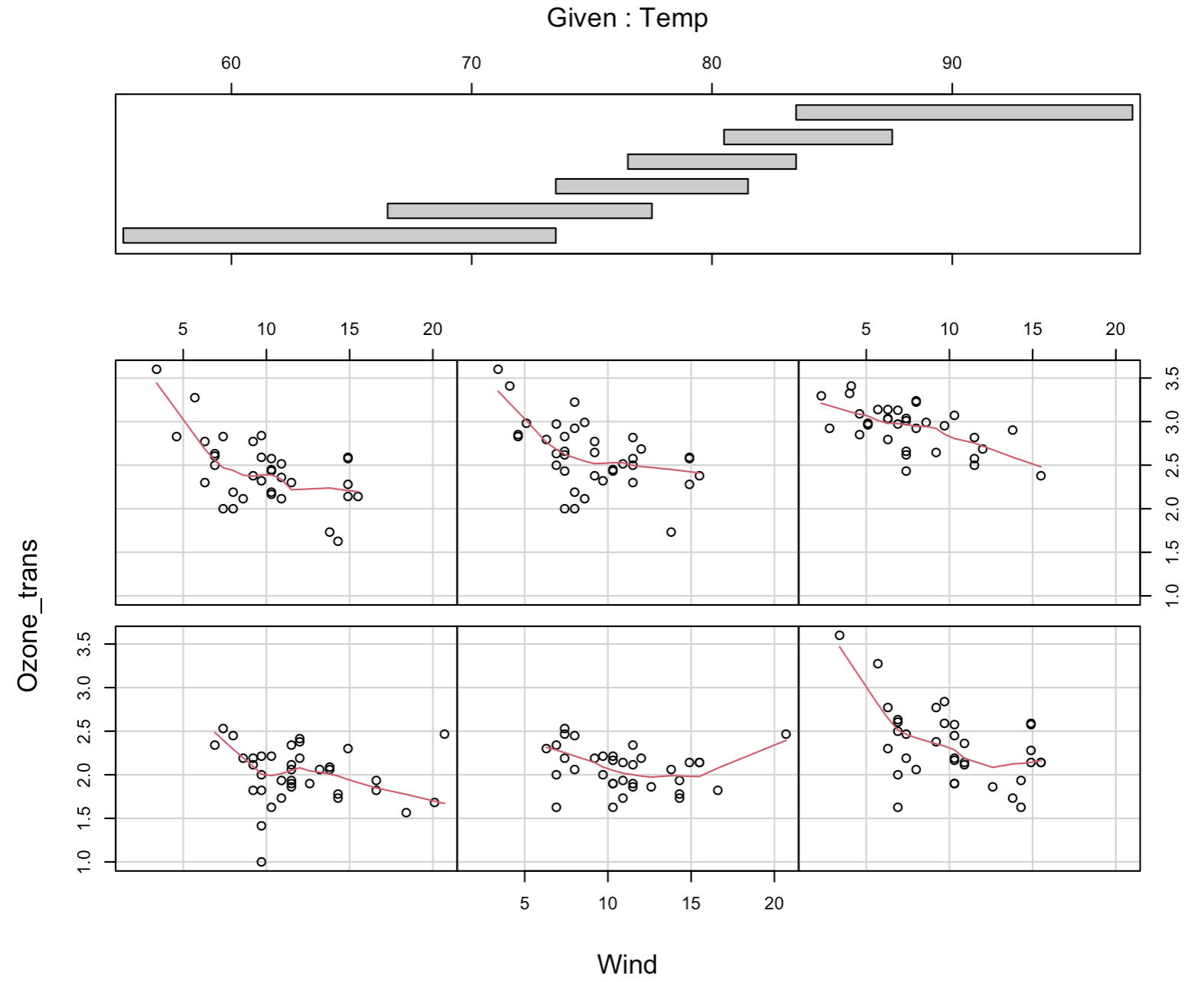
Datenexploration | Interaktionen
![]()
Volles Modell | Validierung

Volles Modell | Numerischer Output
Call:
lm(formula = Ozone_trans ~ Solar.R * Wind * Temp, data = airquality)
Residuals:
Min 1Q Median 3Q Max
-0.64623 -0.19417 -0.04487 0.16945 0.73236
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) -1.664e+00 1.549e+00 -1.074 0.2852
Solar.R 6.045e-03 8.431e-03 0.717 0.4750
Wind 1.983e-01 1.275e-01 1.555 0.1231
Temp 5.322e-02 2.118e-02 2.513 0.0135 *
Solar.R:Wind -7.011e-04 7.438e-04 -0.943 0.3481
Solar.R:Temp -5.464e-05 1.118e-04 -0.489 0.6260
Wind:Temp -3.078e-03 1.801e-03 -1.709 0.0905 .
Solar.R:Wind:Temp 8.789e-06 1.005e-05 0.874 0.3839
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 0.2755 on 103 degrees of freedom
Multiple R-squared: 0.7139, Adjusted R-squared: 0.6945
F-statistic: 36.72 on 7 and 103 DF, p-value: < 2.2e-16Modellauswahl
Automatische ‘backward selection’ mit step()
![]()
Start: AIC=-278.51
Ozone_trans ~ Solar.R * Wind * Temp
Df Sum of Sq RSS AIC
- Solar.R:Wind:Temp 1 0.058039 7.875 -279.69
<none> 7.817 -278.51
Step: AIC=-279.69
Ozone_trans ~ Solar.R + Wind + Temp + Solar.R:Wind + Solar.R:Temp +
Wind:Temp
Df Sum of Sq RSS AIC
- Solar.R:Wind 1 0.025836 7.9008 -281.32
- Solar.R:Temp 1 0.089427 7.9644 -280.44
<none> 7.8750 -279.69
- Wind:Temp 1 0.301181 8.1762 -277.52
Step: AIC=-281.32
Ozone_trans ~ Solar.R + Wind + Temp + Solar.R:Temp + Wind:Temp
Df Sum of Sq RSS AIC
- Solar.R:Temp 1 0.12240 8.0232 -281.62
<none> 7.9008 -281.32
- Wind:Temp 1 0.44304 8.3439 -277.27
Step: AIC=-281.62
Ozone_trans ~ Solar.R + Wind + Temp + Wind:Temp
Df Sum of Sq RSS AIC
<none> 8.0232 -281.62
- Wind:Temp 1 0.67153 8.6948 -274.70
- Solar.R 1 1.50797 9.5312 -264.50
Call:
lm(formula = Ozone_trans ~ Solar.R + Wind + Temp + Wind:Temp,
data = airquality)
Coefficients:
(Intercept) Solar.R Wind Temp Wind:Temp
-1.364170 0.001347 0.129726 0.050008 -0.002197 ‘Optimales’ Modell | Validierung 1

‘Optimales’ Modell | Validierung 2
Zusätzliche Plots manuell erstellt:
Code
res <- residuals(mod_final)
fit <- fitted(mod_final)
par(mfrow = c(2,3))
hist(res)
plot(airquality$Ozone_trans ~ fit, main = "Vorhersage-Performance"); abline(0,1)
# Unabhaengigkeitscheck: Residuen vs. jede erklaerende Var.
plot(0, 0, type = "n", axes = FALSE, xlab="", ylab="")
plot(res ~ airquality$Solar.R); abline(0,0)
plot(res ~ airquality$Wind); abline(0,0)
plot(res ~ airquality$Temp); abline(0,0)
‘Optimales’ Modell | Numerischer Output
Call:
lm(formula = Ozone_trans ~ Solar.R + Wind + Temp + Wind:Temp,
data = airquality)
Residuals:
Min 1Q Median 3Q Max
-0.6036 -0.2040 -0.0415 0.1940 0.7572
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) -1.3641697 0.6571265 -2.076 0.04032 *
Solar.R 0.0013472 0.0003018 4.463 2.02e-05 ***
Wind 0.1297264 0.0579676 2.238 0.02732 *
Temp 0.0500078 0.0080260 6.231 9.56e-09 ***
Wind:Temp -0.0021973 0.0007377 -2.979 0.00359 **
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 0.2751 on 106 degrees of freedom
Multiple R-squared: 0.7064, Adjusted R-squared: 0.6953
F-statistic: 63.75 on 4 and 106 DF, p-value: < 2.2e-16Modell ohne Interaktion | 1
![]()
Wie wäre die Modellperformance ohne Interaktionsterm?
Anpassen eines Modells mit update()
df AIC
mod_final 6 35.38642
mod_no_interaction 5 42.30848
Call:
lm(formula = Ozone_trans ~ Solar.R + Wind + Temp, data = airquality)
Residuals:
Min 1Q Median 3Q Max
-0.65423 -0.20412 -0.00332 0.17832 0.77052
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 0.3780794 0.3102812 1.219 0.226
Solar.R 0.0012877 0.0003121 4.127 7.31e-05 ***
Wind -0.0410683 0.0088073 -4.663 9.04e-06 ***
Temp 0.0282069 0.0034121 8.267 4.07e-13 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 0.2851 on 107 degrees of freedom
Multiple R-squared: 0.6818, Adjusted R-squared: 0.6729
F-statistic: 76.42 on 3 and 107 DF, p-value: < 2.2e-16Modell ohne Interaktion | 2
![]()
OHNE Interaktion

MIT Interaktion

- Die Residuen sehen im Modell mit Interaktionsterm etwas besser aus.
- Da auch der AIC deutlich besser hier ist, behalten wir den Interaktionsterm im finalen Modell.
Modellvisualisierung
Solar.R Effekt
Kombinierter Effekt von Wind und Temp
Code
library(plotly)
df <- data_grid(airquality,
Wind = seq_range(Wind, 50),
Temp = seq_range(Temp, 50),
.model = mod_final) |>
add_predictions(model = mod_final)
df_wide <- df |>
pivot_wider(names_from = Wind,
values_from = pred) |>
select(-c(1:2)) |>
as.matrix()
p1 <- plot_ly(airquality,
x = ~ Wind,
y = ~ Temp,
z = ~ Ozone_trans,
type = "scatter3d")
p2 <- add_trace(p = p1,
z = df_wide,
x = seq_range(airquality$Wind, 50),
y = seq_range(airquality$Temp, 50),
type = "surface")
p2Fragen..??

Total konfus?

Buchkapitel zum Nachlesen
- The R Book von M.J. Crawley:
- Kapitel 10.13 Multiple Regression
- Experimental Design and Data Analysis for Biologists von G.P. Quinn & M.J. Keough:
- Kapitel 6.1 Multiple linear regression analysis
Übungsaufgabe
Übung
![]()
- Zuhause (VOR der Übung): Aufgabe 1 des Übungsskripts DS3_04_Uebungen_MultipleRegression.Rmd:
- Lesen des Appendix A - Required Pre-knowledge: A Linear Regression and Additive Modelling Example aus Zuur et al. (2009): Mixed Effects Models and Extensions in Ecology with R
- Ausführen und Kennenlernen des dazugehörigen Codes (s. Notebook)
- WÄHREND der Übungsstunde: Aufgabe 2 des Übungsskripts
- Durchführung einer Modellauswahl (wie in Aufgabe 1) nur ohne Faktor ‘fGraze’.
- Beantwortung des Moodle-Quiz zur Aufgabe 1
R Notebook, Datensatz und Anhangstext sind im Moodlekurs (Woche 4) zu finden.
Total gelangweilt?

Dann testen Sie doch Ihr Wissen in folgendem Abschlussquiz…
Abschlussquiz
![]()

Bei weiteren Fragen: saskia.otto(at)uni-hamburg.de

Diese Arbeit is lizenziert unter einer Creative Commons Attribution-ShareAlike 4.0 International License mit Ausnahme der entliehenen und mit Quellenangabe versehenen Abbildungen.
Kurswebseite: Data Science 3




