'data.frame': 80 obs. of 4 variables:
$ Treatment: int 100 100 100 100 100 100 100 100 100 100 ...
$ Patch : int 1 1 1 1 1 2 2 2 2 2 ...
$ Quadrat : int 1 2 3 4 5 1 2 3 4 5 ...
$ Algae : int 0 0 0 6 2 0 0 0 0 0 ...
2-faktorielle Varianzanalyse: verschachteltes Design
DS3 - Explorative Datenanalyse & Data Mining
Saskia Otto
Universität Hamburg, IMF
Wintersemester 2023/2024
Lernziele
![]()
Am Ende dieser VL- und Übungseinheit werden Sie
- wissen, wie pseudoreplizierte Daten und Daten von verschachtelten bzw. hierarchischen Designs mithilfe einer Varianzanalyse analysiert werden können.
- eine alternative, bessere Variante zur Analyse der Fallstudiedaten (aus DS2) durchführen können..
- wissen, wie feste und zufällige Faktoren in die Berechnung der F-Werte einfließen
- 3 Varianten zur Berechnung einer mehrfaktoriellen, verschachtelten ANOVA kennen.
2-faktorielle Varianzanalyse mit verschachtelten Faktoren
-Theorie

Verschachtelte (hierarchische) Designs
![]()
- In der Ökologie wird Pseudoreplikation oft verursacht durch fehlende Replikation auf der richtigen Ebene des experimentellen Designs.
- Subsampling kann hier hilfreich sein.
- Häufig: ‘nested ANOVA’ mit zwei Faktoren
- Faktor A mit p Gruppen oder Stufen → fest oder zufällig, aber normalerweise fest
- Faktor B mit q Gruppen oder Ebenen innerhalb jeder Ebene von A (die Unterprobe) → in der Regel zufällig
Einfluss der Seeigelbeweidung im Korallenriff
![]()
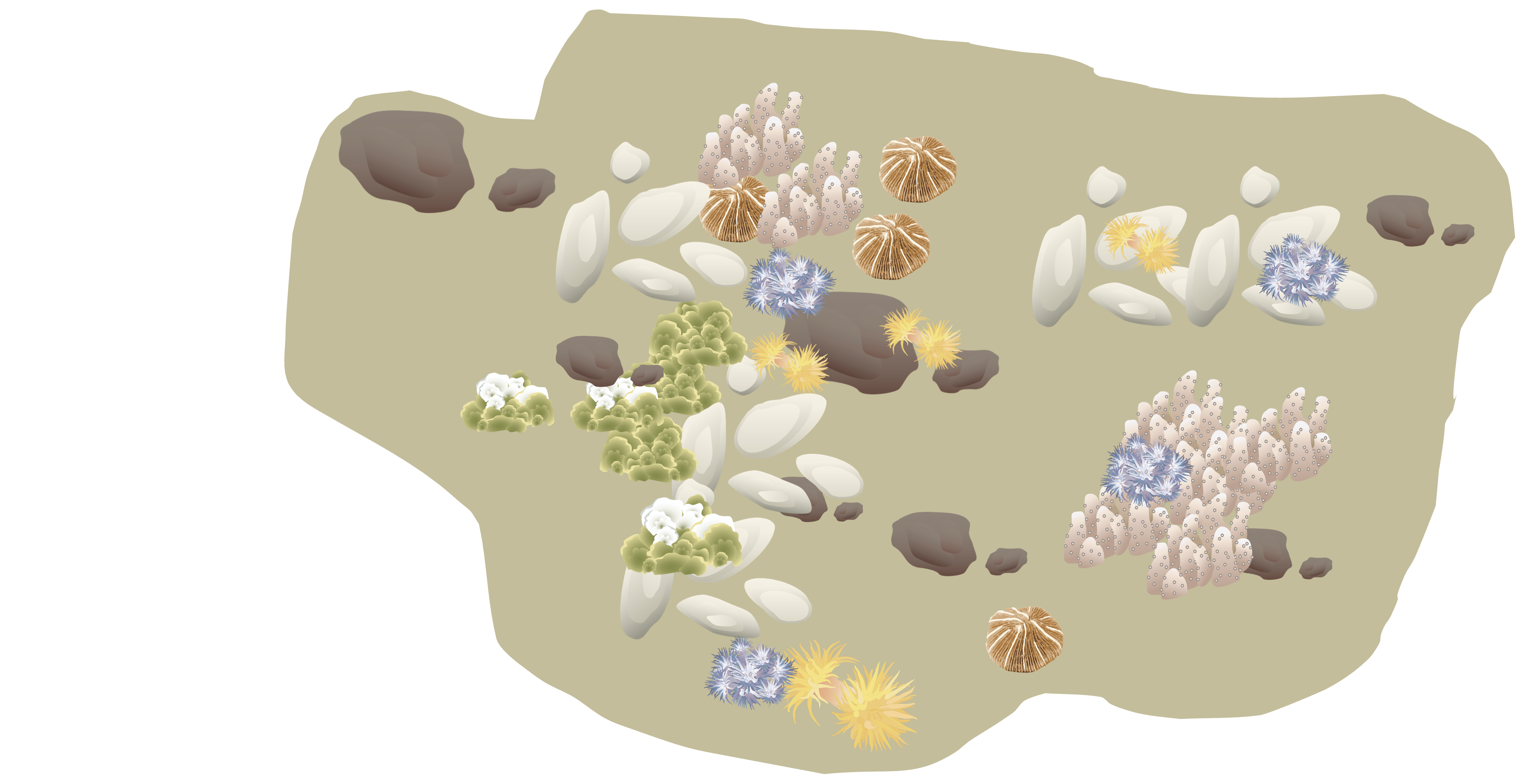
Beispiel ist aus Kapitel 9.1 in Quinn & Keough (2002): Experimental Design and Data Analysis for Biologists
- Andrew & Underwood (1993) manipulierten die Dichte von Seeigeln in der flachen subtidalen Region eines Standorts in der Nähe von Sydney, um die Auswirkungen auf den prozentualen Bewuchs mit Fadenalgen zu testen.
- Es gab vier Seeigelbehandlungen: keine Seeigel, 33% der ursprünglichen Dichte, 66% der ursprünglichen Dichte und 100% der ursprünglichen Dichte.
- Die Behandlungen wurden an vier verschiedenen Stellen (3-4 m^2 Flächen) pro Behandlung wiederholt, und der prozentuale Bewuchs mit Fadenalgen (Antwortvariable) wurde in fünf zufälligen Quadraten pro Stelle gemessen.
- Es handelt sich somit um ein verschachteltes Design mit Behandlung (fester Faktor), Stellen (im Engl. ‘patches’) innerhalb der Behandlung (zufälliger Faktor) und Quadraten als Replikate.
Seeigelbeweidung im Korallenriff
Das Design
![]()
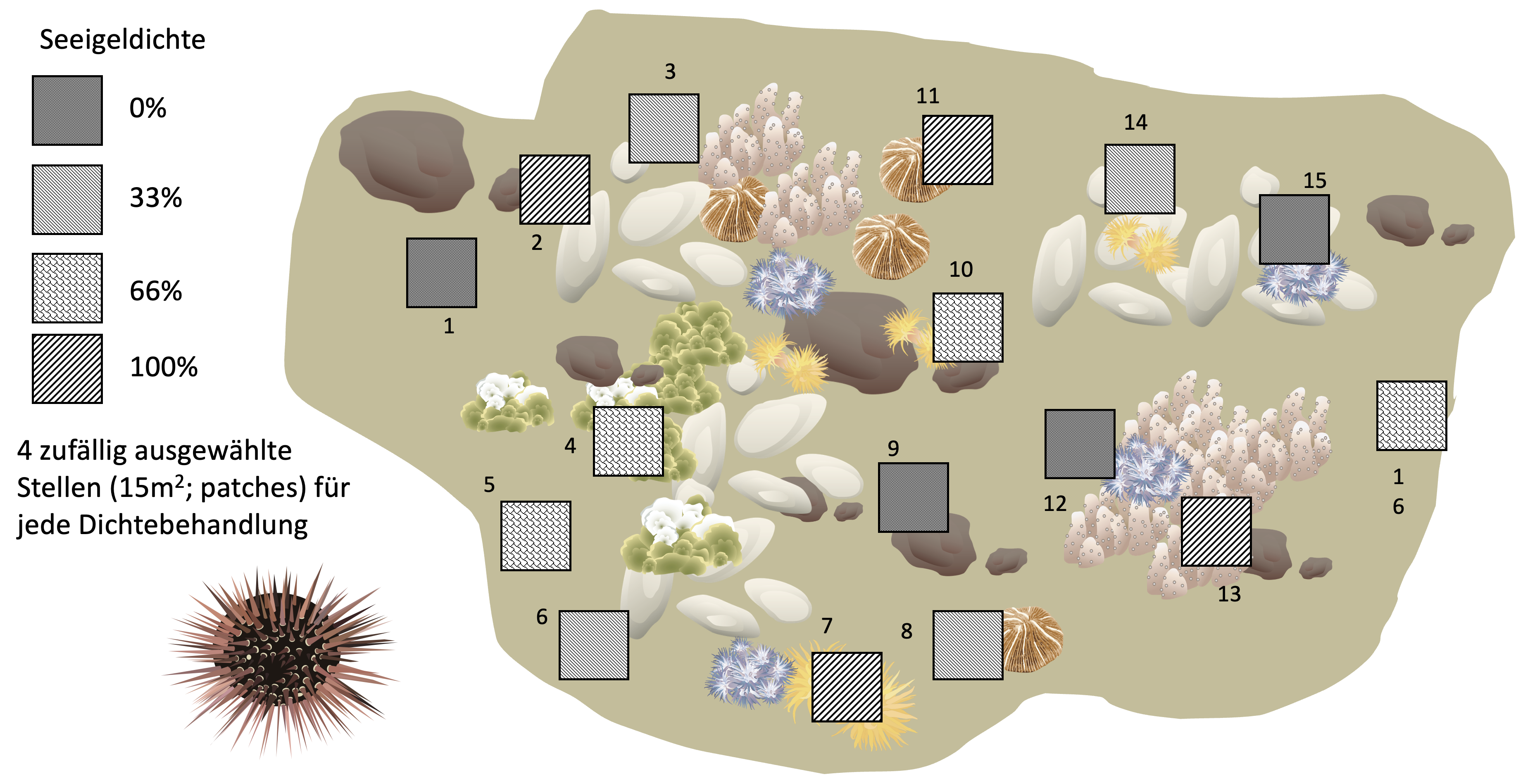
- Jeder Patch (15 m^2) ist zu groß, um ihn zu erfassen ➜ 5 Quadrate (3-4 m^2) als Unterprobe
- Patches werden zu Faktor B innerhalb von Faktor A
- Rest: 5 Wiederholungsquadrate in jedem Patch innerhalb jeder Dichtestufe
Lineares ANOVA Model
![]()
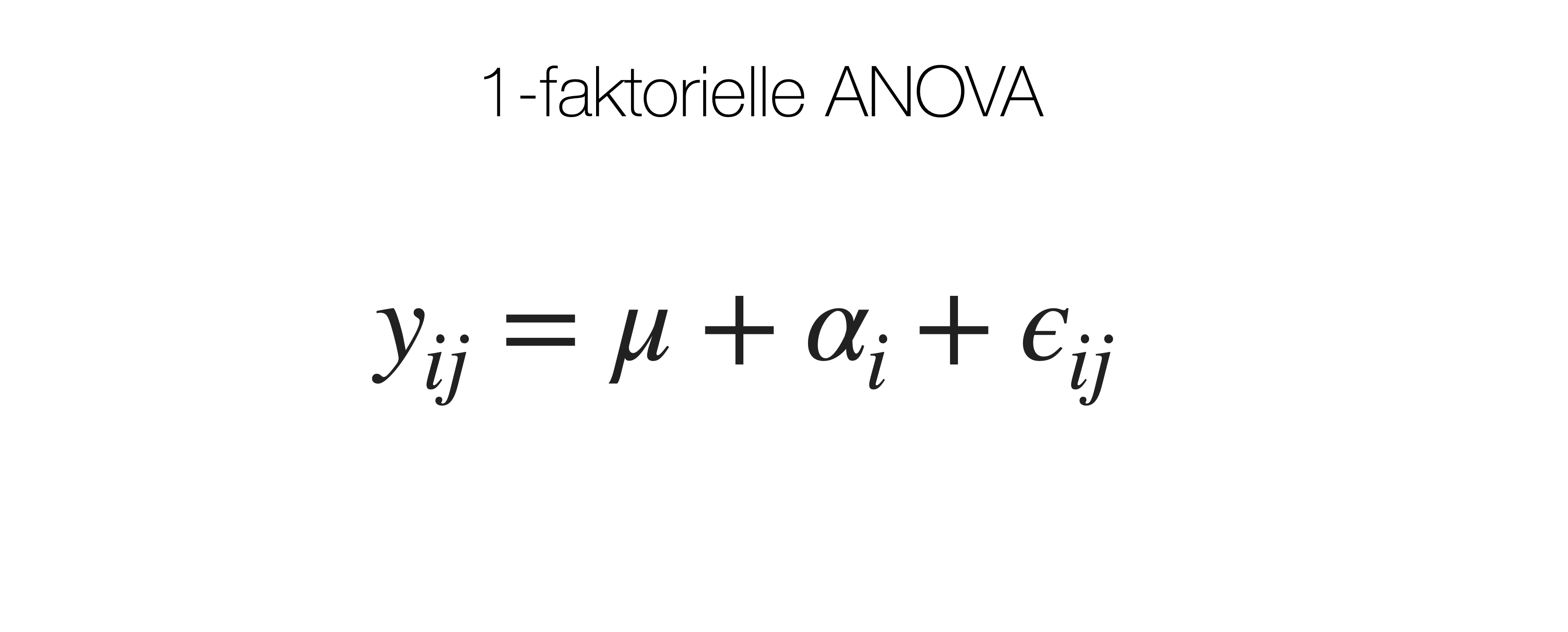
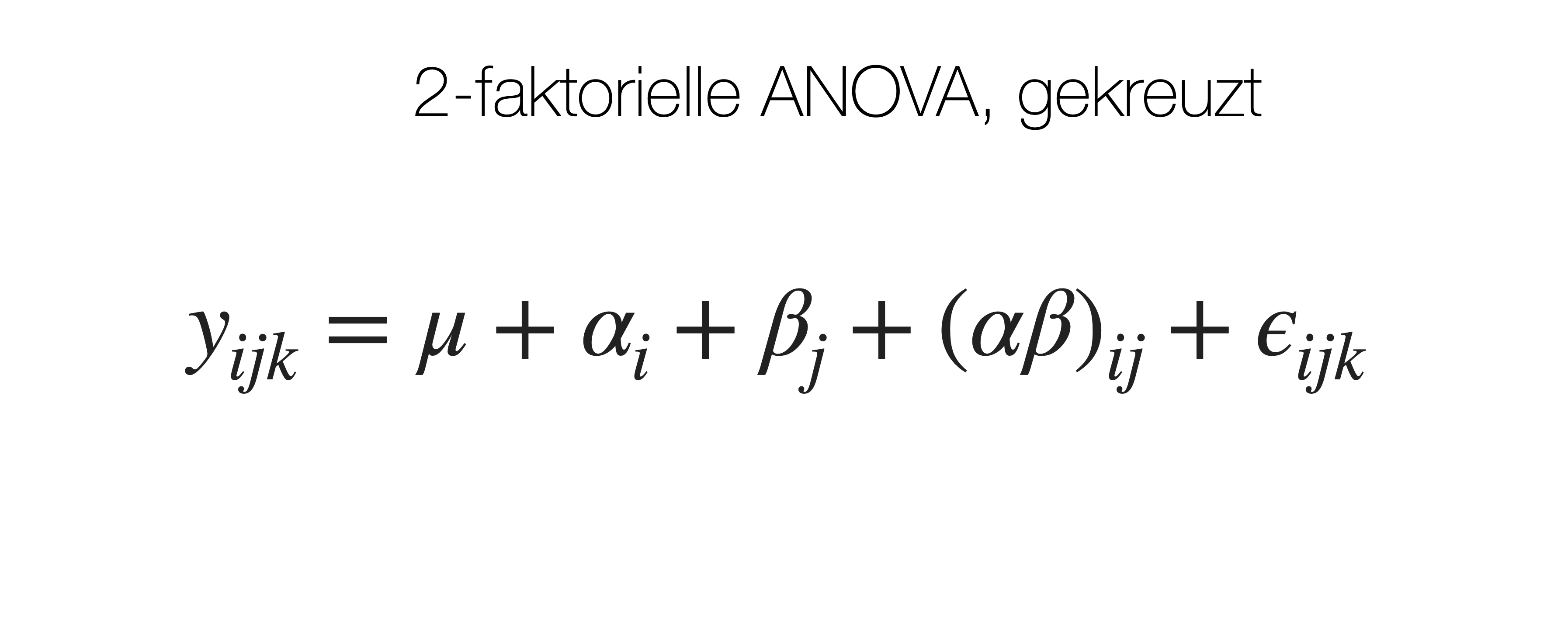


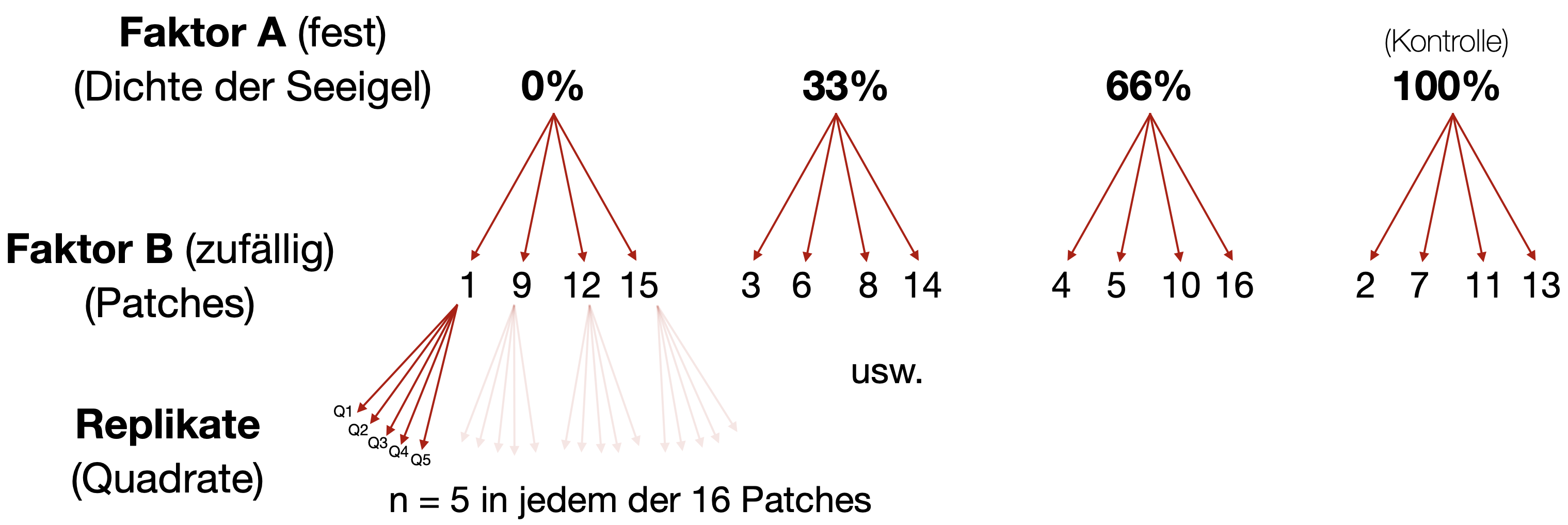
In unserem Beispiel ist
- i = Dichtestufe 1,..,p (p=4 hier) (Faktor A)
- j = Patches 1,..,q (q=4 hier) in jeder Gruppe von p (Faktor B)
- k = Replikate 1,..,n (n=5 hier)
\text{(% Bewuchs Fadenalgen)} = \mu + \text{(Seeigeldichte)}_i + \text{(Patch innerhalb der Seeigeldichte)}_{j(i)} + \epsilon_{ijk}
Hypothesen
Wir wollen nun 2 Hypothesen testen
Faktor A - H0_A: \alpha_1 = \alpha_2 = .. \alpha_i = 0
- → d.h. es gibt keinen Unterschied in der durchschnittlichen Menge an Fadenalgen zwischen den vier Seeigeldichte-Behandlungen
Faktor B - H0_B: \sigma_{\beta}^2 = 0
- → d.h. es gibt keine signifikante Variabilität zwischen den Patches innerhalb jeder Dichtestufe
Varianzzerlegung


ANOVA Tabelle

Berechnung der F-Werte

Interpretation der Ergebnisse
![]()

\Rightarrow Die H0_A kann nicht abgelehnt werden: es gibt keine signifikanten Unterschiede in der Menge der Fadenalgen zwischen den verschiedenen Dichtestufen.
\Rightarrow Die H0_B kann abgelehnt werden: es gibt sign. Unterschiede zwischen den Patches innerhalb jeder Dichtestufe.
2-faktorielle Varianzanalyse mit verschachtelten Faktoren
-Durchführung in R

Datentyp

![]()
Beide Faktoren müssen in R auch als Faktor definiert sein.
Codierung

![]()
Der verschachtelte Faktor (hier ‘Patch’) muss für jede Stufe eine einzigartige ID haben!
# A tibble: 16 × 3
# Groups: Treatment, Patch [16]
Treatment Patch n
<fct> <fct> <int>
1 0 1 5
2 0 2 5
3 0 3 5
4 0 4 5
5 33 1 5
6 33 2 5
7 33 3 5
8 33 4 5
9 66 1 5
10 66 2 5
11 66 3 5
12 66 4 5
13 100 1 5
14 100 2 5
15 100 3 5
16 100 4 5# A tibble: 16 × 3
# Groups: Treatment, Patch_newIDs [16]
Treatment Patch_newIDs n
<fct> <fct> <int>
1 0 0:1 5
2 0 0:2 5
3 0 0:3 5
4 0 0:4 5
5 33 33:1 5
6 33 33:2 5
7 33 33:3 5
8 33 33:4 5
9 66 66:1 5
10 66 66:2 5
11 66 66:3 5
12 66 66:4 5
13 100 100:1 5
14 100 100:2 5
15 100 100:3 5
16 100 100:4 5Exploration

![]()
Durchführung der ANOVA in R

Verschiedene Möglichkeiten
Variante 1
aov() Funktion mit Angabe des verschachtelten (festen) Faktors in der Formel:
aov(y ~ A/B/C)
wobei A/B/C das gleiche ist wie
A + B %in% A + C %in% B %in% A- Antwortvariable y
- Fester Faktor A
- Der zweite Faktor B ist eingebettet (‘nested’) in A → für R fest
- Der dritte Faktor C ist eingebettet (‘nested’) in Faktor B, eingebettet in A → für R fest
Variante 2
aov() Funktion mit Angabe des zufälligen (verschachtelten) Faktors in der Formel:
aov(y ~ A + Error(B))
- fester Faktor A
- Faktor B wird hier als zufälliger Faktor definiert → bei eindeutigen IDs erkennt R die Verschachtelung automatisch.
Variante 3
Als lineares gemischtes Effektmodell (LME) mit dem R Paket ‘nlme’ (oder ‘lme4’):
nlme:lme(fixed = y ~ a, random = ~1 | b)
fixed:
- Hier werden die festen, unverschachtelten Faktoren als feste Komponente des Modells definiert. Die Formelschreibweise ist hier wie bekannt.
random:
- Hier werden die zufälligen, verschachtelten Faktoren definiert.
- Der Name der Antwortvariable wird in der Zufallskomponente vor dem Tildezeichen weggelassen.
- Nach dem Achsenabschnitt folgt der vertikale Balken |, der als “bei folgender räumlicher Anordnung der Zufallsvariablen” zu lesen ist.
- In diesem Beispiel ist Faktor C eingebettet in Faktor B und dieser wiederum in A.
Variante 1

![]()
Df Sum Sq Mean Sq F value Pr(>F)
Treatment 3 14429 4810 16.108 6.58e-08 ***
Treatment:Patch_newIDs 12 21242 1770 5.928 8.32e-07 ***
Residuals 64 19110 299
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1Vor- und Nachteile
- Wir erhalten die vollständige Tabelle bei einem 2-faktoriellen, verschachtelte Design.
- Aber: da der verschachtelte Faktor als fester Faktor in R interpretiert wird, ist die Berechnung des F-Werts inkorrekt (anstelle der MS_{B(A)} sind im Nenner die MS_{Residuen}) und muss manuell berechnet werden.
- Wir können die typischen Diagnostikplots erstellen.
Variante 1 | Korrektur

![]()
Manuelle Berechnung des F- und p-Werts für Faktor A (Treatment = Seeigeldichte):
Variante 1 | Modelldiagnostik

![]()
Hinweis
- Es gibt klare Anzeichen, dass die Annahme der Varianzhomogenität nicht gegeben ist.
- Eine Arcus-Sinus- und auch Log-Transformation auf die Prozentdaten haben keine Verbesserung gebracht.
- Zur Vergleichbarkeit mit dem Originalartikel Andrew & Underwood (1993) und dem Buchkapitel 9.1 in Quinn & Keough (2002) wurde die Demonstration hier mit den untransformierten Daten durchgeführt.
Variante 2

![]()
Error: Patch_newIDs
Df Sum Sq Mean Sq F value Pr(>F)
Treatment 3 14429 4810 2.717 0.0913 .
Residuals 12 21242 1770
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Error: Within
Df Sum Sq Mean Sq F value Pr(>F)
Residuals 64 19110 298.6 Vor- und Nachteile
- F- und p-Wert für
Treatment(Seeigeldichte) sind nun richtig berechnet. - Wir erhalten aber keinen F- und p-Wert für unseren verschachtelten Faktor.
- Wir können keine Modelldiagnostikplots mit
ploterstellen oder Residuen mitresiduals()direkt ausgeben.
Variante 3 | anova() Output

![]()
library(nlme)
mod_lme <- lme(fixed = Algae ~ Treatment,
random = ~ 1|Patch_newIDs, data = sea_urchins)
anova(mod_lme) numDF denDF F-value p-value
(Intercept) 1 64 18.555081 0.0001
Treatment 3 12 2.717102 0.0913Vor- und Nachteile
- F- und p-Wert für
Treatment(Seeigeldichte) sind richtig berechnet. - Aber: wir erhalten keine typische ANOVA-Tabelle mit Angabe der Freiheitsgrade und MS sowie der p-Werte der verschachtelten Faktoren.
- Dafür können wir einige andere Informationen extrahieren …
Variante 3 | summary() Output

![]()
Linear mixed-effects model fit by REML
Data: sea_urchins
AIC BIC logLik
694.1502 708.1346 -341.0751
Random effects:
Formula: ~1 | Patch_newIDs
(Intercept) Residual
StdDev: 17.15554 17.28005
Fixed effects: Algae ~ Treatment
Value Std.Error DF t-value p-value
(Intercept) 39.20 9.407876 64 4.166721 0.0001
Treatment33 -20.20 13.304746 12 -1.518255 0.1548
Treatment66 -17.65 13.304746 12 -1.326594 0.2093
Treatment100 -37.90 13.304746 12 -2.848607 0.0147
Correlation:
(Intr) Trtm33 Trtm66
Treatment33 -0.707
Treatment66 -0.707 0.500
Treatment100 -0.707 0.500 0.500
Standardized Within-Group Residuals:
Min Q1 Med Q3 Max
-1.9807559 -0.3105567 -0.1092864 0.2831185 2.5909912
Number of Observations: 80
Number of Groups: 16 Variante 3 | Anzeige der Effektgrößen

![]()
(Intercept) Treatment33 Treatment66 Treatment100
39.20 -20.20 -17.65 -37.90 (Intercept)
0:1 -4.1565747
0:2 18.9539808
0:3 -30.7586531
0:4 15.9612470
33:1 -13.6335652
33:2 -15.7949840
33:3 15.4624581
33:4 13.9660911
66:1 5.6945074
66:2 12.6775530
66:3 -17.0835222
66:4 -1.2885382
100:1 0.2493945
100:2 -1.0807094
100:3 -0.2493945
100:4 1.0807094Variante 3 | Varianzkomponenten

![]()
Berechnung der prozentualen Varianzkomponenten in der zufälligen Komponente:
Patch_newIDs = pdLogChol(1)
Variance StdDev
(Intercept) 294.3125 17.15554
Residual 298.6000 17.28005In der intercept ist der Faktor Patch_newIDs versteckt.
- → Von den zufälligen Effekten entfällt 49.7% der Varianz der Fadenalgenbedeckung auf die unterschiedlichen Patches innerhalb der Dichtestufen.
Variante 3 | Modelldiagnostik

![]()
![]()
Variante 4 - Die ‘Summary Measure
Analysis’

![]()
Zum Vergleich
Df Sum Sq Mean Sq F value Pr(>F)
Treatment 3 2886 961.9 2.717 0.0913 .
Residuals 12 4248 354.0
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 12-faktorielle Varianzanalyse mit verschachtelten Faktoren
-Beispiel DS2 Fallstudie

Datenaufbereitung
![]()
'data.frame': 450 obs. of 15 variables:
$ Gruppenname : chr "A-Team" "A-Team" "A-Team" "A-Team" ...
$ Datum : chr "17.4.2023" "17.4.2023" "17.4.2023" "17.4.2023" ...
$ Uhrzeit : chr "14:00" "14:00" "14:00" "14:00" ...
$ Breitengrad : num 53.6 53.6 53.6 53.6 53.6 ...
$ Laengengrad : num 9.97 9.97 9.97 9.97 9.97 ...
$ Standorttyp : chr "sonnig" "sonnig" "sonnig" "sonnig" ...
$ Art : chr "Iris virginica" "Iris virginica" "Iris virginica" "Iris virginica" ...
$ Gattung : chr "Iris" "Iris" "Iris" "Iris" ...
$ Familie : chr "Iridaceae" "Iridaceae" "Iridaceae" "Iridaceae" ...
$ Ordnung : chr "Asparagales" "Asparagales" "Asparagales" "Asparagales" ...
$ Pflanzen_ID : int 1 1 1 1 1 1 1 1 1 1 ...
$ Blueten_ID : int 1 1 1 2 2 2 3 3 3 4 ...
$ Kronblatt_ID: int 1 2 3 4 5 6 7 8 9 10 ...
$ Blattbreite : num 2.15 2.15 2.03 1.77 2 ...
$ Blattlaenge : num 5.6 5.7 5.6 5.4 5.5 5.5 6 5.8 6 5.6 ...# ID-Spalten als Faktor definieren
demo$Pflanzen_ID_f <- factor(demo$Pflanzen_ID)
demo$Blueten_ID_f <- factor(demo$Blueten_ID)
demo$Kronblatt_ID_f <- factor(demo$Kronblatt_ID)
# Gruppenname auch als Faktor abspeichern (fuer Modelle)
demo$Gruppenname_f <- factor(demo$Gruppenname,
levels = c("A-Team", "B Group", "CClub"),
labels = c("A", "B", "C"))
# Erstellen von einzigartigen IDs und Speicherung der Faktoren als Einzelvektoren
gruppe <- demo$Gruppenname_f
pflanze <- demo$Gruppenname_f:demo$Pflanzen_ID_f
bluete <- demo$Gruppenname_f:demo$Pflanzen_ID_f:demo$Blueten_ID_f
# Eine Datenexploration hat gezeigt, dass die Blattlaenge
# log-transformiert werden muss
y <- log(demo$Blattlaenge)Variante 1: aov()
Bei diesem Datensatz dauert Variante 1 sehr (!!!) lange.
Variante 1: aov() | Korrektur
Manuelle Berechnung des F- und p-Werts für Faktor A (gruppe):
Variante 3: nlme::lme()
Variante 3: nlme::lme() | Effektgrößen
(Intercept) gruppeB gruppeC
1.6839400 -1.2885207 -0.2374901 Level: pflanze
(Intercept)
A:1 0.026049304
A:2 0.104378797
A:3 0.089879458
A:4 -0.020456987
A:5 0.134588902
A:6 -0.065285951
A:7 -0.040002404
A:8 -0.043275682
A:9 -0.085227928
A:10 -0.100647508
B:1 0.180113993
B:2 -0.085323417
B:3 -0.083100345
B:4 -0.025494836
B:5 -0.057011711
B:6 0.035337550
B:7 0.106376960
B:8 0.024575963
B:9 -0.025535840
B:10 -0.069938317
C:1 -0.116483168
C:2 0.045447014
C:3 0.086482600
C:4 -0.147459911
C:5 -0.058896042
C:6 -0.036848622
C:7 0.055178502
C:8 0.009277929
C:9 0.094217799
C:10 0.069083899
Level: bluete %in% pflanze
(Intercept)
A:1/A:1:1 0.0155010746
A:1/A:1:2 -0.0094261918
A:1/A:1:3 0.0584861363
A:1/A:1:4 0.0002688297
A:1/A:1:5 -0.0568013465
A:2/A:2:1 -0.0498614009
A:2/A:2:2 0.0071208528
A:2/A:2:3 0.0341449838
A:2/A:2:4 0.0473656393
A:2/A:2:5 -0.0066001027
A:3/A:3:1 0.0855442808
A:3/A:3:2 0.0280941420
A:3/A:3:3 -0.0683333312
A:3/A:3:4 -0.0277666270
A:3/A:3:5 0.0101627522
A:4/A:4:1 -0.0291775359
A:4/A:4:2 -0.0341148930
A:4/A:4:3 0.0491140579
A:4/A:4:4 0.0589954379
A:4/A:4:5 -0.0511219939
A:5/A:5:1 0.0137078987
A:5/A:5:2 0.0355680547
A:5/A:5:3 0.0004261852
A:5/A:5:4 0.0094231064
A:5/A:5:5 -0.0176443947
A:6/A:6:1 0.0249022444
A:6/A:6:2 -0.0956396202
A:6/A:6:3 -0.0301203959
A:6/A:6:4 0.0456890206
A:6/A:6:5 0.0350473537
A:7/A:7:1 -0.0126235506
A:7/A:7:2 0.0247049991
A:7/A:7:3 0.0401306374
A:7/A:7:4 -0.0572894385
A:7/A:7:5 -0.0072515521
A:8/A:8:1 -0.0315053310
A:8/A:8:2 -0.0260269494
A:8/A:8:3 0.0326913173
A:8/A:8:4 0.0010541610
A:8/A:8:5 0.0104490596
A:9/A:9:1 0.0252306637
A:9/A:9:2 -0.0250983175
A:9/A:9:3 0.0307090453
A:9/A:9:4 -0.0378350797
A:9/A:9:5 -0.0192739080
A:10/A:10:1 -0.0014787929
A:10/A:10:2 0.0430807119
A:10/A:10:3 0.0378110435
A:10/A:10:4 -0.0430931473
A:10/A:10:5 -0.0673397891
B:1/B:1:1 0.0093144799
B:1/B:1:2 0.0692328716
B:1/B:1:3 -0.0214560063
B:1/B:1:4 0.0692328716
B:1/B:1:5 -0.0708123473
B:2/B:2:1 -0.1079738859
B:2/B:2:2 0.0013890781
B:2/B:2:3 0.0218909914
B:2/B:2:4 0.0791514656
B:2/B:2:5 -0.0207546753
B:3/B:3:1 -0.0209578685
B:3/B:3:2 0.0186308696
B:3/B:3:3 -0.0209578685
B:3/B:3:4 0.0186308696
B:3/B:3:5 -0.0209578685
B:4/B:4:1 -0.0482655143
B:4/B:4:2 -0.0086767762
B:4/B:4:3 0.0104100486
B:4/B:4:4 0.0091777560
B:4/B:4:5 0.0294968733
B:5/B:5:1 -0.0426100696
B:5/B:5:2 -0.0016062428
B:5/B:5:3 0.0913632956
B:5/B:5:4 -0.0426100696
B:5/B:5:5 -0.0221081562
B:6/B:6:1 -0.0782512985
B:6/B:6:2 -0.0222231169
B:6/B:6:3 -0.0031362922
B:6/B:6:4 0.0325727721
B:6/B:6:5 0.0819291130
B:7/B:7:1 -0.0096144671
B:7/B:7:2 0.0397418739
B:7/B:7:3 0.0071572878
B:7/B:7:4 -0.0442407541
B:7/B:7:5 0.0397418739
B:8/B:8:1 -0.0502329202
B:8/B:8:2 0.0582760806
B:8/B:8:3 0.0057952614
B:8/B:8:4 -0.0120592707
B:8/B:8:5 0.0057952614
B:9/B:9:1 0.0104440802
B:9/B:9:2 0.0295309049
B:9/B:9:3 -0.0496465713
B:9/B:9:4 -0.0277295692
B:9/B:9:5 0.0295309049
B:10/B:10:1 -0.0523835763
B:10/B:10:2 -0.0318816629
B:10/B:10:3 -0.0318816629
B:10/B:10:4 0.0472958133
B:10/B:10:5 0.0472958133
C:1/C:1:1 -0.0413476957
C:1/C:1:2 0.0660640078
C:1/C:1:3 -0.0181571146
C:1/C:1:4 0.0390524005
C:1/C:1:5 -0.0815121869
C:2/C:2:1 0.0099725693
C:2/C:2:2 -0.0149021073
C:2/C:2:3 -0.0608058907
C:2/C:2:4 0.0517786323
C:2/C:2:5 0.0279637521
C:3/C:3:1 -0.0244951776
C:3/C:3:2 -0.0244951776
C:3/C:3:3 0.0235456522
C:3/C:3:4 0.0518561457
C:3/C:3:5 0.0002428484
C:4/C:4:1 0.0001742116
C:4/C:4:2 -0.0156386196
C:4/C:4:3 -0.0633830158
C:4/C:4:4 0.0717656287
C:4/C:4:5 -0.0383659528
C:5/C:5:1 -0.0364299132
C:5/C:5:2 -0.0364299132
C:5/C:5:3 0.0523177500
C:5/C:5:4 0.0388199887
C:5/C:5:5 -0.0364299132
C:6/C:6:1 -0.0125122732
C:6/C:6:2 -0.0198540083
C:6/C:6:3 -0.0403559217
C:6/C:6:4 0.0275026373
C:6/C:6:5 0.0338626698
C:7/C:7:1 -0.0104016029
C:7/C:7:2 0.0497717806
C:7/C:7:3 0.0139260657
C:7/C:7:4 0.0190947525
C:7/C:7:5 -0.0553847557
C:8/C:8:1 0.0872453527
C:8/C:8:2 0.0022466149
C:8/C:8:3 -0.0307874420
C:8/C:8:4 -0.0372971359
C:8/C:8:5 -0.0185478942
C:9/C:9:1 -0.0368646633
C:9/C:9:2 0.0230755167
C:9/C:9:3 0.0625541178
C:9/C:9:4 0.0230755167
C:9/C:9:5 -0.0428021767
C:10/C:10:1 -0.0285949830
C:10/C:10:2 0.0324066193
C:10/C:10:3 0.0496396485
C:10/C:10:4 -0.0034277107
C:10/C:10:5 -0.0287316337Variante 3: nlme::lme() | Varianzkomp.
Berechnung der prozentualen Varianzkomponenten in der zufälligen Komponente:
Variance StdDev
pflanze = pdLogChol(1)
(Intercept) 0.007918459 0.08898572
bluete = pdLogChol(1)
(Intercept) 0.002440501 0.04940143
Residual 0.001500141 0.03873166[1] 66.77115 20.57914 12.64970- → Von den zufälligen Effekten entfällt 67% der Varianz der Blattlängen auf die unterschiedlichen Pflanzen (innerhalb der Gruppen), 20% auf die Blüten (innerhalb der Pflanzen) und 13% auf die einzelnen Kronblätter innerhalb einer Blüte.
Zusammenfassung
![]()
- Bei verschachtelten (hierarchischen) Designs und Pseudoreplikation sind die verschachtelten Faktoren meist zufällige Faktoren.
- Als Denominator beim F-Quotient des Hauptterms (die Behandlungsstufe meist) gehen die MS des verschachtelten Faktors ein, wenn dieser zufällig ist!
- Daher ist es wichtig in R, den verschachtelten Faktor eindeutig als zufällig zu definieren.
- Alle 3 vorgestellten Varianten haben ihre Vor- und Nachteile.
- Variante 1:
- ⨁ Wir erhalten die vollständige Tabelle bei einem 2-faktoriellen, verschachtelte Design und können die typischen Diagnostikplots erstellen.
- ⊖ F- und p-Wert vom Hauptfaktor müssen manuell berechnet werden.
- Variante 2:
- ⨁ F- und p-Wert für den Hauptfaktor werden richtig berechnet.
- ⊖ Wir erhalten keinen F- und p-Wert für unseren verschachtelten Faktor und können keine Modelldiagnostikplots mit
plot()erstellen oder Residuen mitresiduals()direkt ausgeben.
- Variante 3:
- ⨁ F- und p-Wert für den Hauptfaktor werden richtig berechnet und wir können einige Informationen extrahieren wie Effektgrößen und Anteile der Varianzkomponenten.
- ⊖ Aber: wir erhalten keine typische ANOVA-Tabelle mit Angabe der Freiheitsgrade und MS sowie der p-Werte der verschachtelten Faktoren.
Fragen..??

Total konfus?

Buchkapitel zum Nachlesen
- The R Book von M.J. Crawley:
- Kapitel 11.3 Pseudoreplication: Nested designs and split plots
- Kapitel 19.5 Hierarchical sampling and variance components analysis
- Experimental Design and Data Analysis for Biologists von G.P. Quinn & M.J. Keough:
- Kapitel 9.1 Nested (hierarchical) designs
Übungsaufgabe
Übung
![]()
- Zuhause (VOR der Übung):
- Durchführung einer 2-faktoriellen, verschachtelten ANOVA für eine Umweltverträglichkeitsstudie (curdies.csv Datensatz).
- WÄHREND der Übungsstunde:
- Durchführung einer 3-faktoriellen, verschachtelten ANOVA auf die Fallstudiendaten aus DS2 (siehe Frage 2 der Fallstudie).
Das R Notebook und der Datensatz für 1. sind im Moodlekurs (Woche 2) zu finden. Das Skript dieser VL ist auch auf Moodle.
Für Aufgabe 2 die Fallstudiendaten aus DS2 mitbringen!
Total gelangweilt?

Dann testen Sie doch Ihr Wissen in folgendem Abschlussquiz…
Abschlussquiz
![]()

Bei weiteren Fragen: saskia.otto(at)uni-hamburg.de

Diese Arbeit is lizenziert unter einer Creative Commons Attribution-ShareAlike 4.0 International License mit Ausnahme der entliehenen und mit Quellenangabe versehenen Abbildungen.
Kurswebseite: Data Science 3




