Df Sum Sq Mean Sq F value Pr(>F)
dosis_fac 1 6834 6834 167.1 9.38e-12 ***
Residuals 22 900 41
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Moduleinführung: was ist EDA, DM, ML und SL?
DS3 - Vom experimentellen Design zur
explorativen Datenanalyse & Data Mining
Saskia Otto & Monika Eberhard
Universität Hamburg, IMF
Wintersemester 2025/2026
Modulübersicht

Lernziele des Moduls
![]()
Am Ende des Semesters werden Sie …
- grundlegende Kenntnisse zu verschiedenen Methoden des Lernens aus Daten erworben haben,
- ein Verständnis für statistische Modelle entwickelt haben und Methoden wie die Kovarianzanalyse, multiple Regression, lineare gemischte Modelle sowie Resampling-Techniken auf Daten anwenden können,
- die Prinzipien des überwachten und unüberwachten Lernens kennen und bei letzterem zwei grundlegende Methoden der multivariaten Analyse anwenden und interpretieren können.
- die Grundlagen von Open Science und die Anwendung von R Markdown bzw. Quarto zur Dokumentation und Kommunikation wissenschaftlicher Arbeiten beherrschen,
- in der Lage sein, die erlernten Modellierungsverfahren und Analysetechniken auf die Fallstudie aus dem vorherigen Data Science Modul 2 anzuwenden.
Modulstruktur: Theorie & Praxis
Vorlesung
- Wöchentliche VL (75min)
- keine Anwesenheitspflicht
- VL-Aufzeichnungen auf Moodle
Abschließende Klausur (Note)
- 90min, inkl. praktischem Übungsteil
- 1. Termin am 4.2.2026, 10 Uhr (IZS)
- 2. Termin am 16.3.2026, 10 Uhr (IZS)
- ansonsten in jedem folgenden Wintersemester
Übungen
- 7 Übungstage (90min)
- Anwesenheitspflicht!
- Fehlzeiten max. 15% (1 Übungstag)
- Leistungsnachweis: Moodle-Quizze zu den
- Vorbereitungsaufgaben (@home)
- Übungsaufgaben
Das Modul wird als vollständig abgeschlossen betrachtet, sobald beide Komponenten erfolgreich absolviert wurden.
Vorlesungsthemen
| VL | Datum | Thema | Übung |
|---|---|---|---|
| 1 | 14.11.2025 | Moduleinführung und Wiederholung DS2 | → Übung 1 |
| 2 | 21.11.2025 | 2-faktorielle, gekreuzte Varianzanalyse (ANOVA) | → Übung 2 |
| 3 | 28.11.2025 | Kovarianzanalyse (ANCOVA) | → Übung 3 |
| 4 | 05.12.2025 | Lineare Gemischte Modelle und verschachtelte ANOVA | → Übung 4 |
| 5 | 12.12.2025 | Multiple lineare Regression | |
| 6 | 19.12.2025 | VIDEO: Open Science und R Markdown | |
| - | Weihnachtsferien | ||
| 7 | 09.01.2026 | Resampling-Techniken | → Übung 5 |
| 8 | 16.01.2026 | Unsupervised Learning 1: Clusteranalyse | → Übung 6 |
| 9 | 24.01.2026 | Unsupervised Learning 2: PCA | → Übung 7 |
| 10 | 30.01.2026 | Wiederholung |
Übungsgruppen
| Gruppe | Tag & Zeit | Raum | Dozent*in | Termine |
|---|---|---|---|---|
| A | Mo, 13:30–15:00 | IZS, 115 | Dr. Kim Rohlfing | 17.11./24.11./1.12./8.12./12.1./19.1./26.1. |
| B | Mo, 13:00–14:30 | IPM, E.004 | Dr. Dragan Matevski | 17.11./24.11./1.12./8.12./12.1./19.1./26.1. |
| C | Mi, 10:30–12:00 | IPM, gr. HS | Dr. Dragan Matevski | 19.11./26.11./3.12./10.12./14.1./21.1./28.1. |
| D | Mi, 10:30–12:00 | IZS, 115 | Prof. Dr. Kathrin Otte | 19.11./26.11./3.12./10.12./14.1./21.1./28.1. |
| E | Fr, 13:15–14:45 | IZS, Kosswig-Saal | Dr. Monika Eberhard | 21.11./28.11./5.12./12.12./16.1./23.1./30.1. |
| F | Fr, 13:15–14:45 | IZS, 116 | Dr. Kim Rohlfing | 21.11./28.11./5.12./12.12./16.1./23.1./30.1. |
| BMARSYS | Mo, 14:00–15:30 | IMF, GES, 107 | Dr. Saskia Otto | 17.11./24.11./1.12./8.12./12.1./19.1./26.1. |
Materialien & Tools
- VL-Folien im HTML-Format
- Handbuch
- Allgemeine Modulinformationen
- Aufgabenstellung Übungen
- R Studio-Projektordner → ZIP-Datei zum Download
- R Notebooks
- Daten
- Moodle:
- VL: PDF-Folien, Aufzeichnungen
- Quizze begleitend zu den Übungen mit Lösungen
- Klausur, Klausurvorbereitendes Quiz
- Kommunikationsplattform
- RStudio Server



Literatur
Deutsch
- Biostatistik - Praktische Einführung in Konzepte und Methoden von F. Bärlocher, 1999, Thieme Verlag, 206 S.
- Parametrische Statistik - Verteilungen, maximum likelihood und GLM in R von C. Dormann, 2017, Springer Spektrum, 363 S.
Englisch
- Experimental Design and Data Analysis for Biologists von G.P. Quinn & M.J. Keough, 2002, Cambridge, UK, 553 S.
- The R Book von M.J. Crawley, 2013, Wiley, 945 S. \rightarrow sehr umfangreiches Buch (fast 1000 Seiten!)
- Mixed Effects Models and Extensions in Ecology with R von A.F. Zuur, E.N. Ieno, N.J. Walker, A.S. Saveliev und G.M. Smith, 2009, Springer Science+Business Media, LLC, NewYork, U.S.A., 574 S. (https://highstat.com)
- An Introduction to Statistical Learning with Applications in R von G. James, D. Witten, T. Hastie und R. Tibshirani, 2013, Springer Science+Business Media, 426 S.
- Numerical Ecology with R von D. Borcard, F. Gillet & P. Legendre, 2011, Springer, 306 S. → für multivariate Statistik
Kommunikationswege
- Stellen Sie so weit wie möglich alle Fragen während der Übungsstunden und nach der Vorlesung.
- Außerhalb dessen läuft die Kommunikation über Moodle:
- Spezifische Nachrichten bitte direkt an die Übungsgruppenleiter*innen senden (in Moodle).
- Allgemeine Fragen können im Moodle FAQ Forum gestellt werden, die wir oder auch Sie alle beantworten können.
Sonstiges
- Bitte um Fehlerhinweise und Verbesserungsvorschläge
- Kritischer Umgang mit dem Themengebiet
- Kritik, Fragen, Anmerkungen
Weitere Fragen?
Lernen von Daten
Image by starline on Freepik
Entwicklung der Statistik | 1
- Die Entwicklung der Statistik ist eng gekoppelt an Fragestellungen und Herausforderungen in der Wissenschaft und Industrie.
- In den Anfängen stammten diese Probleme häufig aus landwirtschaftlichen und industriellen Experimenten, waren von relativ geringem Umfang und meist Hypothesen-getrieben
- → Student’s t-Verteilung und -Test von William Sealey Gosset bei seiner Forschung für die Guinness Brauerei entwickelt.
- In den 1960ern wurde die “Explorative Datenanalyse (EDA)” maßgeblich von dem Statistiker John W. Tukey entwickelt.
- Mit dem Aufkommen von Computern und dem Informationszeitalter sind die statistischen Probleme sowohl vom Umfang als auch von der Komplexität her explodiert.
Entwicklung der Statistik | 2
- Herausforderungen in den Bereichen Datenspeicherung, -organisation und -suche haben zum neuen Gebiet des “Data Mining” geführt.
- Statistische und rechnerische Probleme in Biologie und Medizin haben die “Bioinformatik” hervorgebracht.
- In vielen Bereichen werden riesige Datenmengen generiert, und die Aufgabe des Statistikers besteht darin, aus all diesen Daten einen Sinn zu machen: wichtige Muster und Trends zu extrahieren und zu verstehen, “was die Daten sagen” → Lernen aus Daten.
EDA vs. Data Mining | 1
Die Explorative Datenanalyse (EDA) und Data Mining sind zwei verschiedene, jedoch miteinander verbundene Konzepte im Bereich der Datenanalyse:
Explorative Datenanalyse (EDA)
- ZWECK:
- Daten erkunden, Muster identifizieren, Hypothesen generieren und einen ersten Einblick in die Daten erhalten.
- METHODEN UND TECHNIKEN:
- Umfasst die deskriptive Statistik, Datenvisualisierung und einfache statistische Tests.
- STADIUM IM ANALYSEPROZESS:
- Oft der erste Schritt in der Datenanalyse, um die Daten besser zu verstehen und Hypothesen für weitere Analysen zu generieren.
- ZIELVARIABLEN:
- Weniger darauf ausgerichtet, vorherzusagen oder spezifische Zielvariablen zu modellieren.
EDA vs. Data Mining | 2
Die Explorative Datenanalyse (EDA) und Data Mining sind zwei verschiedene, jedoch miteinander verbundene Konzepte im Bereich der Datenanalyse:
Data Mining
- ZWECK:
- Verborgene Muster und Zusammenhänge in großen Datenmengen entdecken, um Vorhersagen zu treffen und Muster zu klassifizieren.
- METHODEN UND TECHNIKEN:
- Fortgeschrittenere statistische und maschinelle Lernmethoden wie Clusteranalyse, Klassifikation, Regression, Assoziationsregeln.
- STADIUM IM ANALYSEPROZESS:
- Tritt oft in späteren Stadien der Analyse auf, nachdem die EDA durchgeführt wurde.
- ZIELVARIABLEN:
- Fokus auf der Modellierung von Zielvariablen.
EDA Demonstration













Lernen aus den Daten
→ Die Wissenschaft des Lernens spielt eine Schlüsselrolle in den Bereichen Statistik, Data Mining und künstliche Intelligenz und überschneidet sich mit Bereichen der Ingenieurwissenschaften und anderen Disziplinen.
Was macht ML aus?
ML | Typisches Anwendungsbeispiel
Bilderkennung

ML | Weitere Anwendungen
- Ermittlung der Risikofaktoren für Prostatakrebs auf der Grundlage klinischer und demografischer Variablen.
- Identifizierung von Mustern in Genexpressionsdaten, um Gene zu identifizieren, die in verschiedenen biologischen Zuständen unterschiedlich reguliert sind → Früherkennung von Krebserkrankungen oder anderen genetisch bedingten Krankheiten.
- Analyse von DNA-Sequenzen, um phylogenetische Beziehungen zwischen verschiedenen Arten zu rekonstruieren.
- Analyse komplexe Metagenomik-Daten zur Erforschung der Rolle von Mikroorganismen in verschiedenen Umgebungen, vom Darm bis zu aquatischen Ökosystemen.
- Analyse von ökologische Daten und Entwicklung von Modellen, die uns helfen, die Verbreitung von Arten vorherzusagen, Ökosysteme zu schützen oder invasive Arten zu kontrollieren.
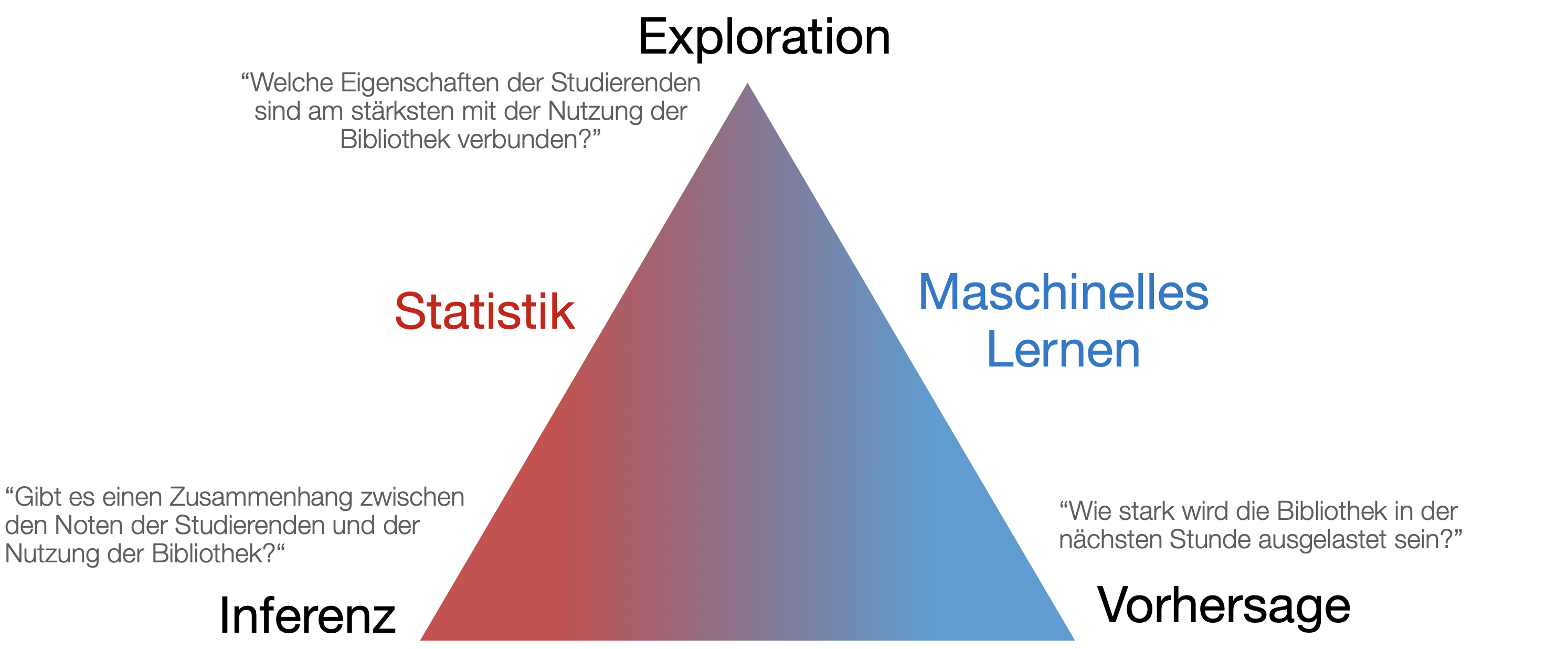
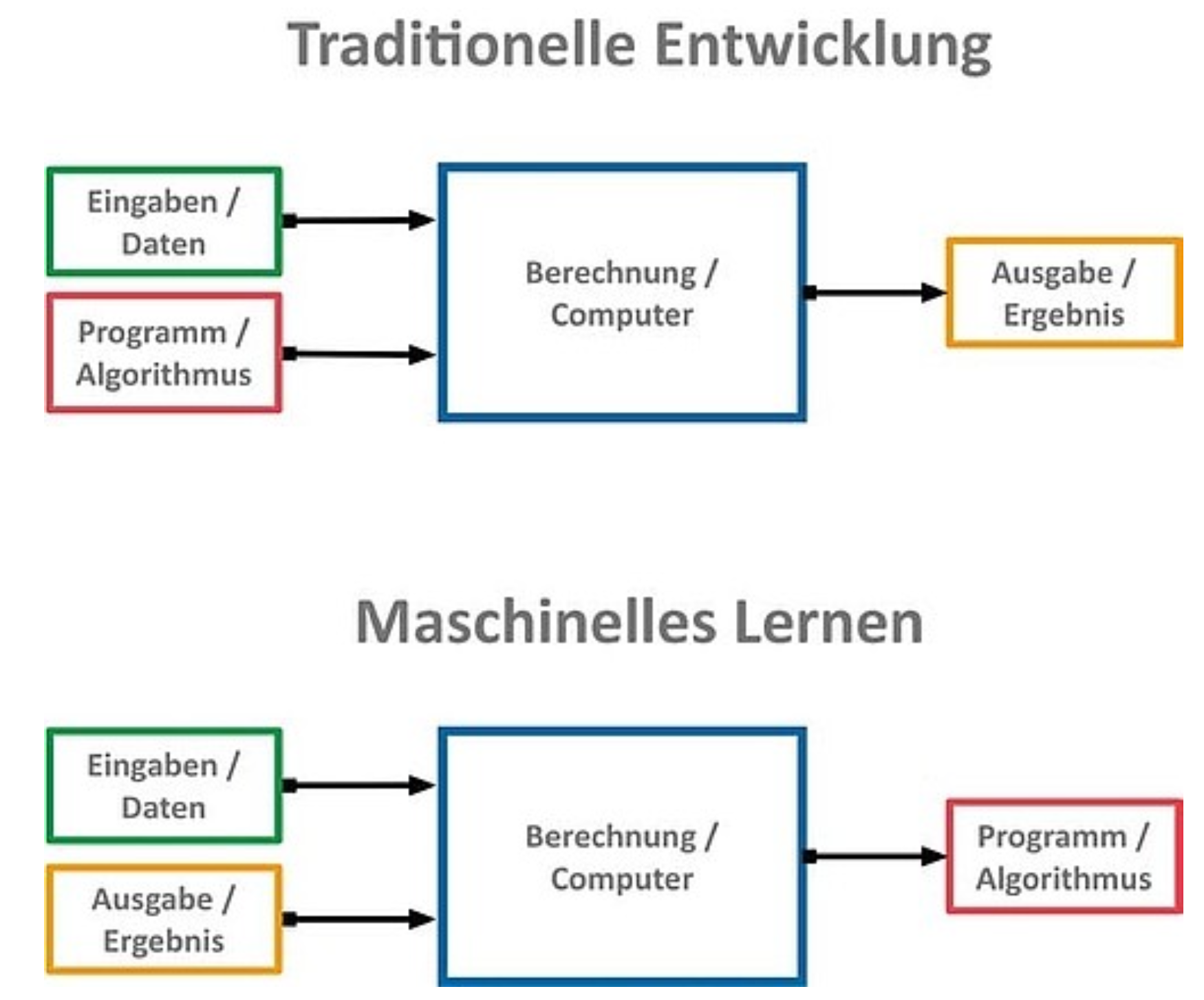
Was ist statistisches Lernen?
![]()
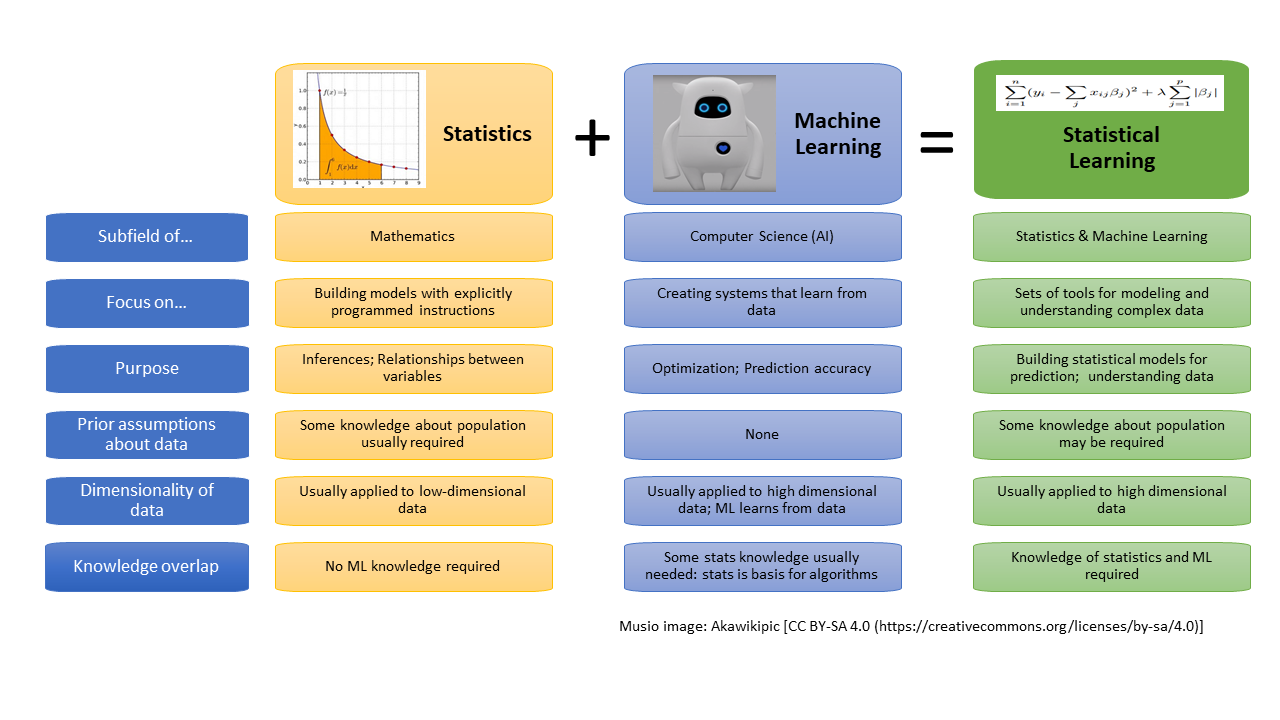
- Maschinelles Lernen ist ein Teilgebiet der Künstlichen Intelligenz.
- Statistisches Lernen entstand als Teilgebiet der Statistik.
- Es gibt viele Überschneidungen - beide Bereiche konzentrieren sich auf überwachte und nicht überwachte Probleme:
- Beim ML liegt der Schwerpunkt stärker auf groß angelegten Anwendungen und der Vorhersagegenauigkeit.
- Beim SL liegt der Schwerpunkt auf Modellen und deren Interpretierbarkeit sowie auf Präzision und Unsicherheit.
- Unterscheidung verschwimmt immer mehr.
‘Supervised’ vs. ‘Unsupervised’ Learning
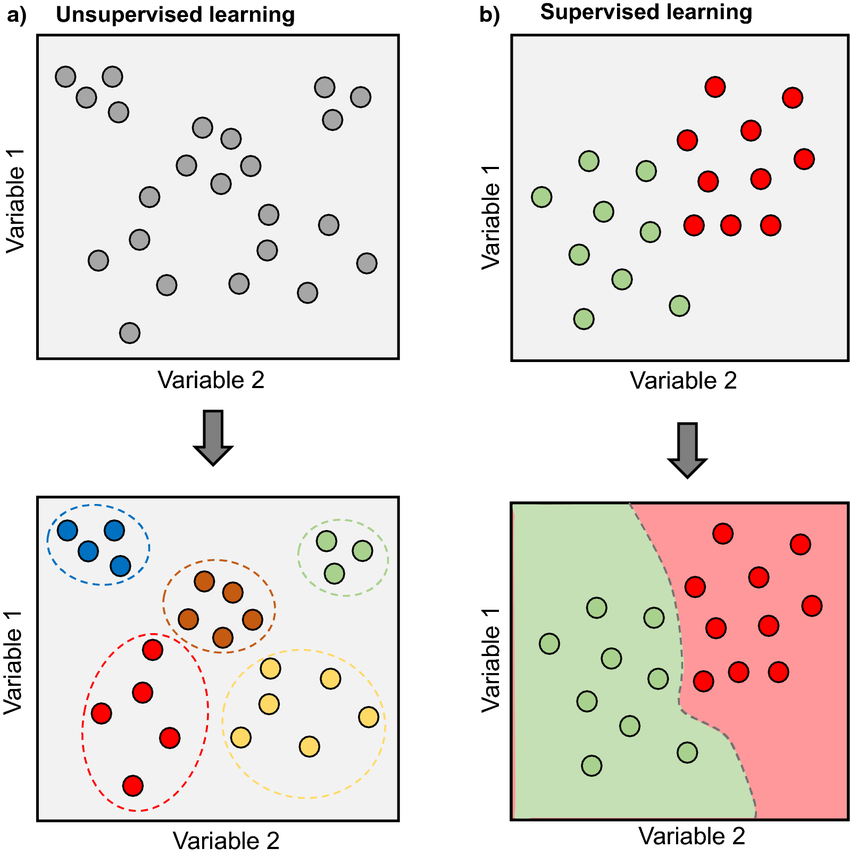
Bildquelle: Morimoto & Ponton (2021): Virtual reality in biology: could we become virtual naturalists?
Supervised Learning
- Ein statistisches Modell wird zur Vorhersage oder Schätzung einer Antwortvariable (Zielvariable, Output) auf der Grundlage einer oder mehrerer unabhängiger Variablen (Inputs) erstellt.
- Dabei ‘lernt’ der Algorithmus aus einem Trainingsdatensatz, indem er iterativ Vorhersagen zu den Daten macht und die vorhergesagte mit der richtigen Antwort vergleicht.
- Ziel ist es, dass der Algorithmus eine Zuordnung von Eingangsvariablen zu Ausgangsvariablen erlernt, die es ihm ermöglicht, Vorhersagen oder Klassifizierungen für neue, nicht gekennzeichnete Daten zu treffen → Optimierung ist das Ziel
Supervised Learning | Häufige Verwendung
- Klassifizierung verschiedener Dateitypen wie Bilder, Dokumente oder geschriebene Wörter.
- Vorhersage zukünftiger Trends und Ergebnisse durch Lernen von Mustern in Trainingsdaten mittels Regression
Supervised Learning | Vorgehensweise
- Die Daten erhalten (viele!!!)
- Daten aufbereiten
- Daten in Trainings- und Validierungs-/Testdaten aufteilen
- Einen Modelltyp auswählen
- Sich für eine Verlustfunktion entscheiden
- Optimierungsalgorithmus (und Lernrate) festlegen [ML]
- Anpassen (“Trainieren”) des Modells
- Bewertung der Modellleistung anhand von Validierungs-/Testdaten (und anderen unabhängigen Datensätzen)
Was ist Optimimierung?
- Wir versuchen, die Parameter eines Modells zu bestimmen (z. B. lineare/logistische Regression).
- Wir berechnen die Verlustfunktion für die Parameter unseres Modells zur Bewertung wie gut das Modell die Zielgröße der Trainingsdaten vorhersagt.
- Der Verlust ist die Abweichung zwischen Vorhersage und Beobachtung
- Typische Verlustfunktion, die für die Modellgenauigkeit herangezogen wird: mittlerer quadratischer Vorhersagefehler bzw. ‘Mean Squared Error’. MSE = \frac{1}{n}\sum_{i=1}^{n}(Y_i - \hat{Y}_i)^2
Ein paar Kompromisse…
- Vorhersagegenauigkeit versus Interpretierbarkeit.
- Lineare Modelle sind leicht interpretierbar, andere wie ‘thin-plate splines’ sind es nicht.
- Gute Anpassung versus Überanpassung oder Unteranpassung.
- Wie wissen wir, wann die Anpassung genau richtig ist?
- Sparsamkeit versus Blackbox.
- Oft bevorzugen wir ein einfacheres Modell mit weniger Variablen gegenüber einem Blackbox-Prädiktor, der alle einschließt.
Flexibilität vs. Interpretierbarkeit
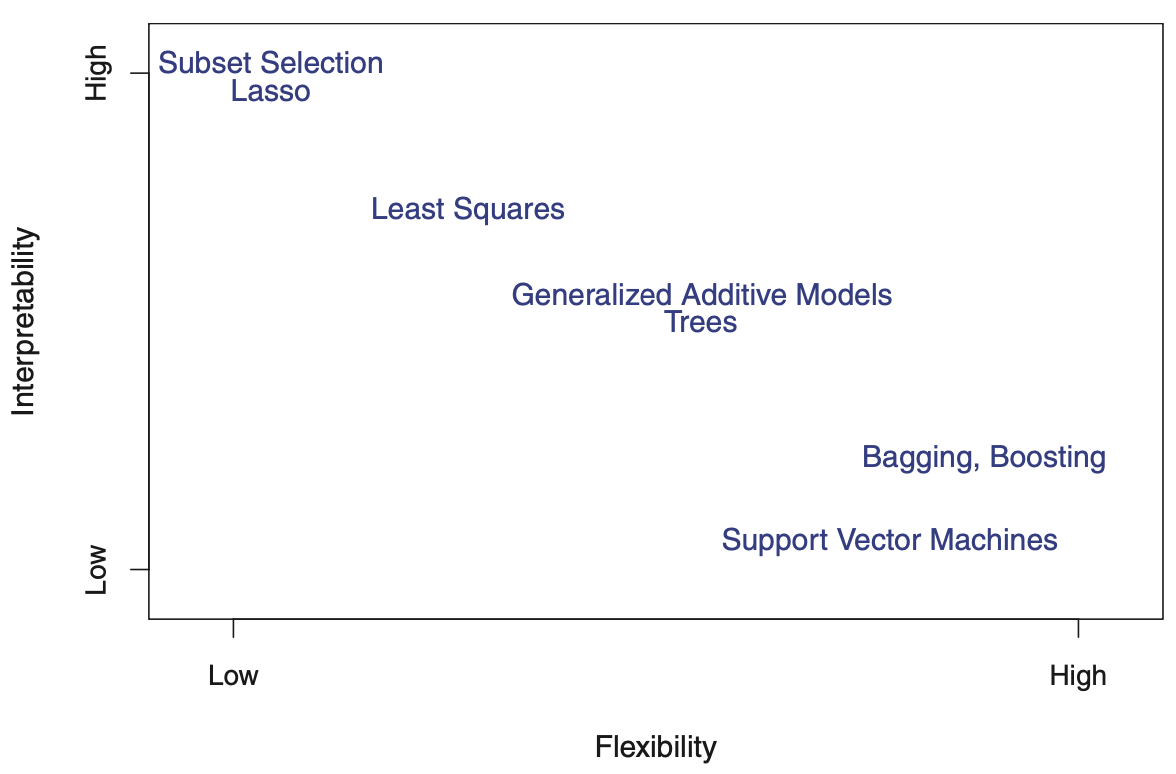
Bildquelle: James et al. (2013): Introduction to Statistical Learning
Performance von Trainings- vs. Testdaten
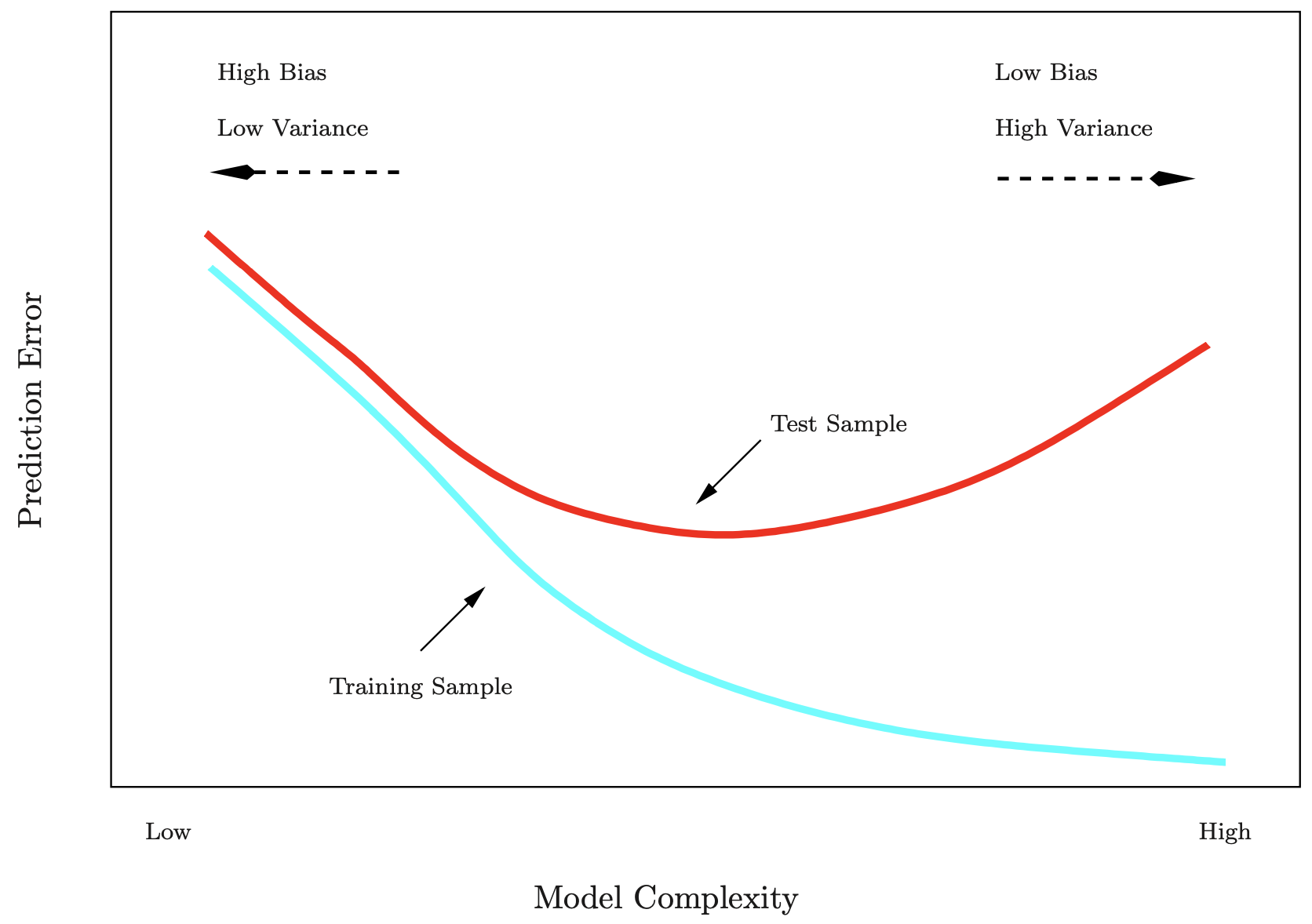
Die Trainingsfehlerrate (MSE_{train}) unterscheidet sich oft erheblich von der Testfehlerrate (MSE_{test}). Insbesondere bei sehr flexiblen Modellen kann die Trainingsfehlerrate die Testfehlerrate drastisch unterschätzen.
Bildquelle: James et al. (2013): Introduction to Statistical Learning
Supervised Learning | Beliebte Methoden
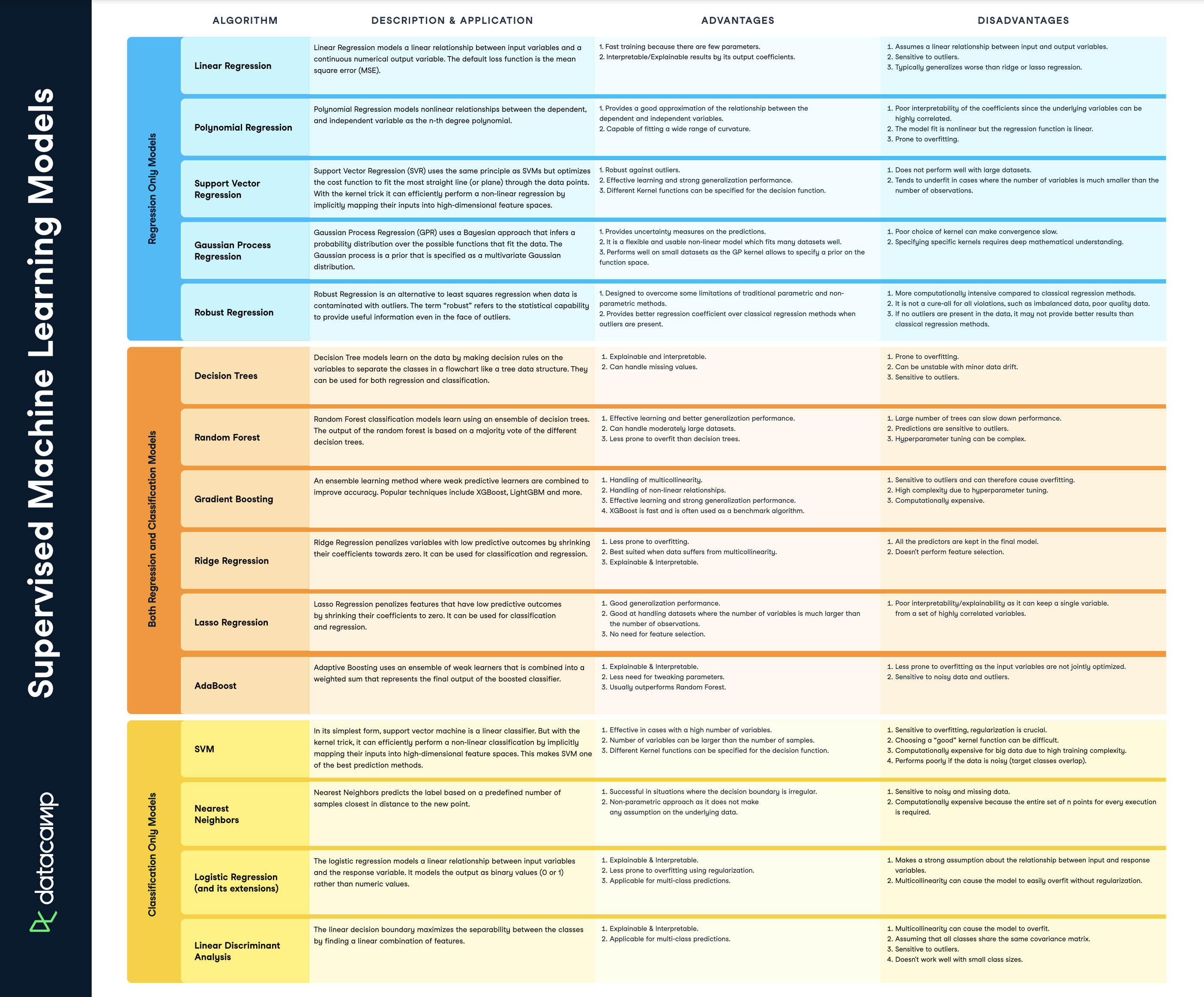
Download-link dieses Cheatsheets: [datacamp.com](https://www.datacamp.com/cheat-sheet/)
Live-Demo in R

Zurück zu Primärdaten
Aber fangen wir erstmal wieder mit kleineren Datensätzen aus (eigenen) Datenerhebungen an …
Zur Erinnerung…
Häufiger Designtyp: CRD
und entsprechende (parametrische) Analysen
Vollständig randomisiertes Design - Completely Randomised Design (CRD)
- Alle Versuchseinheiten sind unabhängig und werden den Kombinationen an Behandlungsstufen zufällig zugeordnet.
- Unterscheidet in
- 1 (fester) Faktor, 2 Gruppen → t-Test (oder 1-way ANOVA)
- 1 (fester) Faktor, mehrere Gruppen → 1-way ANOVA (oder Regression)
- 2 (feste) Faktoren, gekreuzt → 2-way crossed ANOVA
- 1 (fester) Faktor mit Unterproben (Pseudoreplikation) → 2-way nested ANOVA / LME
- 1 (fester) Faktor, 1 Kovariate → ANCOVA
- 1 (zufälliger) Faktor, 1 Kovariate → LME
Feste vs. zufällige Effekte
Feste Effekte → Behandlungseffekte
- Die meisten Standardtests gehen von festen Faktoren aus.
- Die einzelnen Faktorstufen beziehen sich auf den Mittelwert von Y.
Zufällige Effekte → biologische/technische Eff.
- Die einzelnen Faktorstufen beziehen sich auf die Varianz von Y.
- Zufällige Effekte sind meist weniger interessant.
- Haben meist viele Faktorstufen
Auswahl des Effekttypen
| Fragen, die Sie sich stellen sollten | Fest | Zufällig |
|---|---|---|
| Interessiert an den Unterschieden zwischen den Faktorstufen? | ☑️︎ | |
| Ist der Faktor ein Behandlungseffekt? | ☑️ | |
| Sind die Faktorstufen informativ? | ☑️ | |
| Repräsentieren die Faktorstufen experimentelle Interventionen? | ☑️ | |
| Sind die Faktorstufen speziell ausgewählt? | ☑️ | |
| Kommen alle Stufen des Faktors im Experiment vor? | ☑️ | |
| Kommen die Faktorstufen von der gleichen Population? | ☑️ | |
| Gibt es viele Faktorstufen? | ☑️ | |
| Soll auf andere Faktorstufen, die nicht im Experiment vorkommen, verallgemeinert werden? | ☑️ | |
| Gibt es ein verschachteltes (nested) oder hierarchisches (Sub)sampling? | ☑️ |
Quelle: Lazic (2016) Experimental Design for Laboratory Biologists
Completely Randomised Design (CRD)
Alle experimentellen Einheiten sind unabhängig und können zufällig allen Kombinationen von Behandlungsstufen zugeteilt werden.
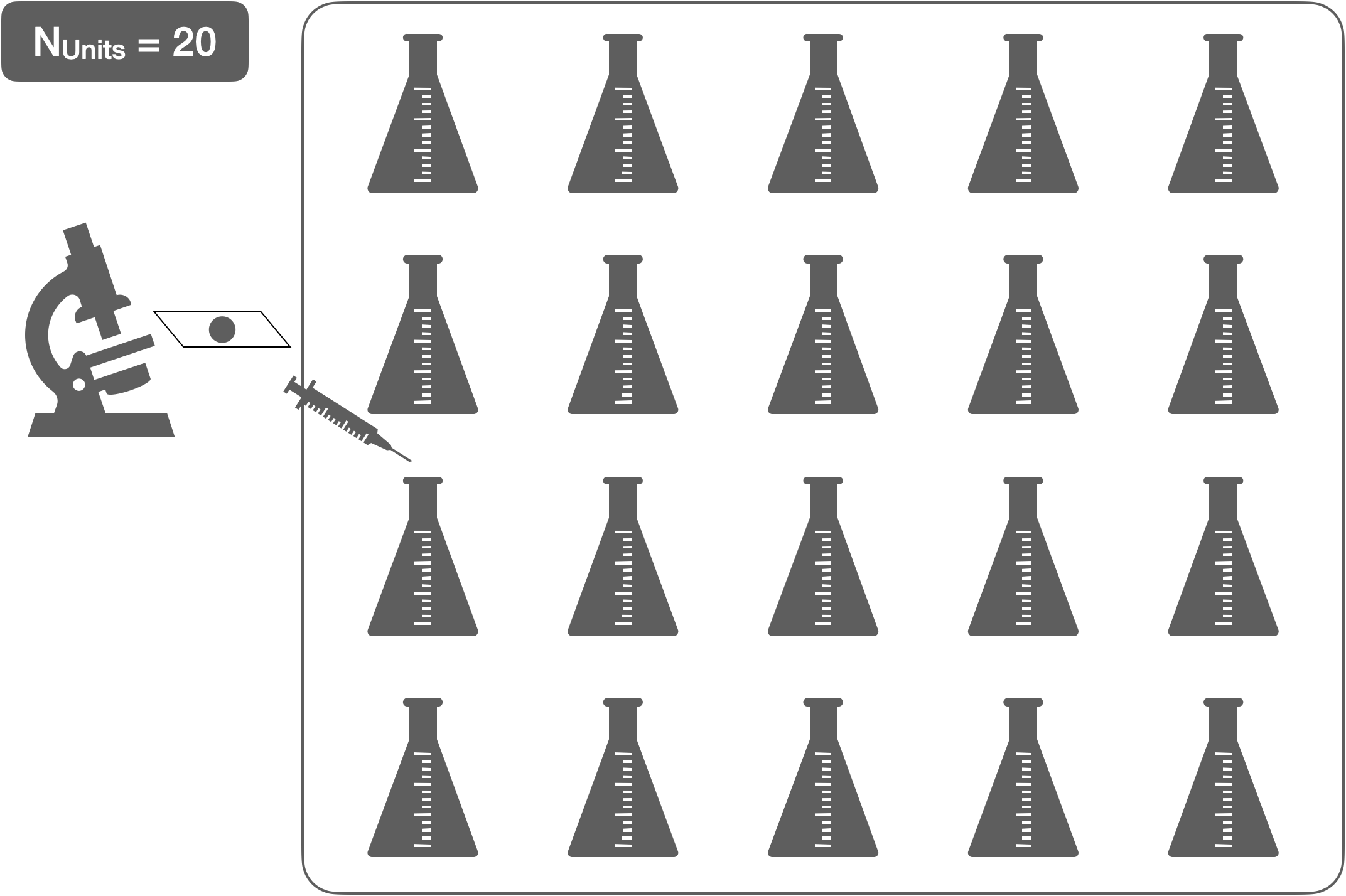
CRD - 2 gekreuzte Faktoren (je 2 Gruppen)
Gekreuztes Design
- N: 20 (Erlenmeyerkolben)
- n: 5 (EK pro Dosis/Typ)
- Ergebnis: Zellzahl
- Behandlungseffekte:
- Dosis (fest) = {0, 100}
- Typ (fest) = {1, 2}
- Dosis (fest) = {0, 100}
- Analyse: 2-way crossed/factorial ANOVA

CRD - 1 Faktor (2 Gruppen) mit Unterprobe
Verschachteltes Design
- N: 8 (Exp. Units: Erlenmeyerkolben)
- n: 3 (Objektträger als Unterproben pro EK)
- Ergebnis: Zellzahl
- Effekte:
- Behandlungseff.: Dosis (fest) = {0, 100}
- Technischer Eff.: EK (zufällig) = {1,2,3,..,8}
- Analyse: 2-way nested ANOVA (oder ‘summary measure’ analysis)
- Freiheitsgrade:
\begin{align*} \text{Ergebnis} &= \text{Dosis} + \text{Fehler}\\ \text{(N - 1)} &= \text{(p - 1)} + \text{(N - p)}\\ \text{(7)} &= \text{(1)} + \text{(6)} \end{align*}

Vergleich beider Designs
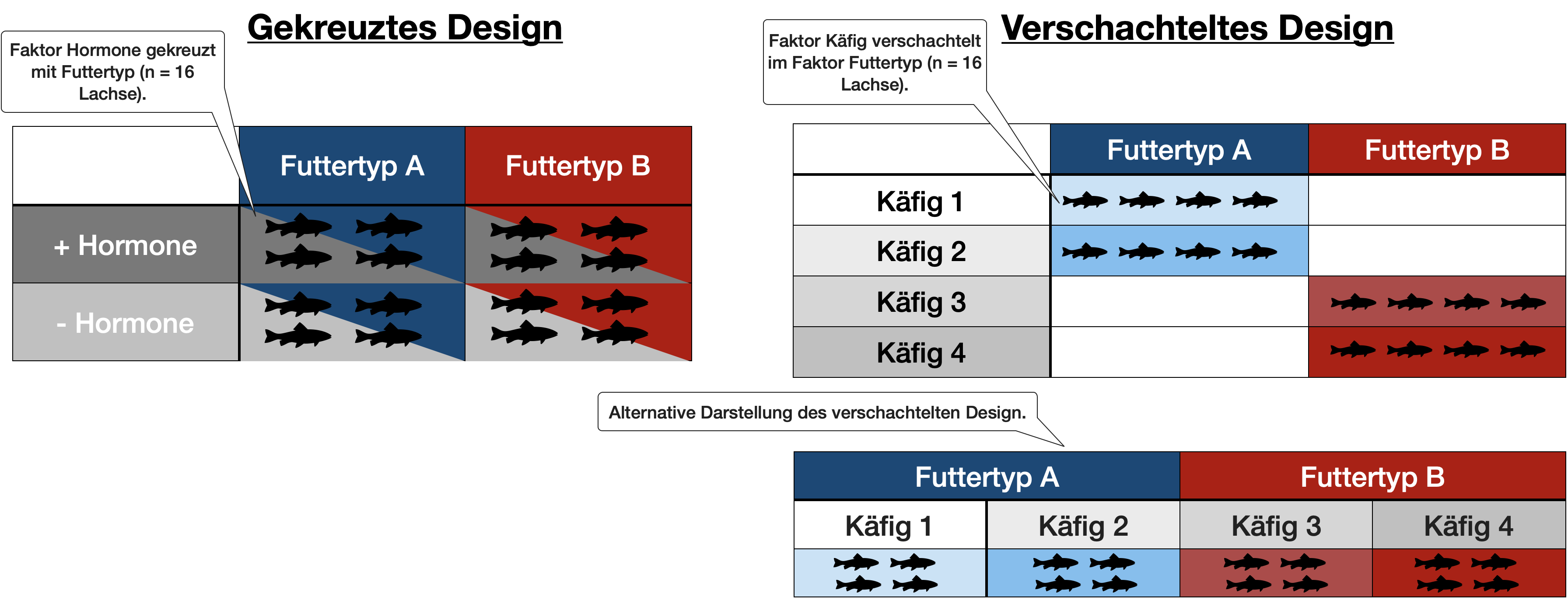
CRD - 1 Faktor mit Unterprobe | Ansätze
- Falscher Ansatz:
- 1-faktorielle ANOVA, ohne Berücksichtigung der Pseudoreplikation
- Richtige Ansätze:
- 1-faktorielle ANOVA mit gemittelten Unterprobewerte (‘summary measure’ analysis).
- Lineares Gemischtes Modell (LME) mit dem R Paket nlme (oder lme4).
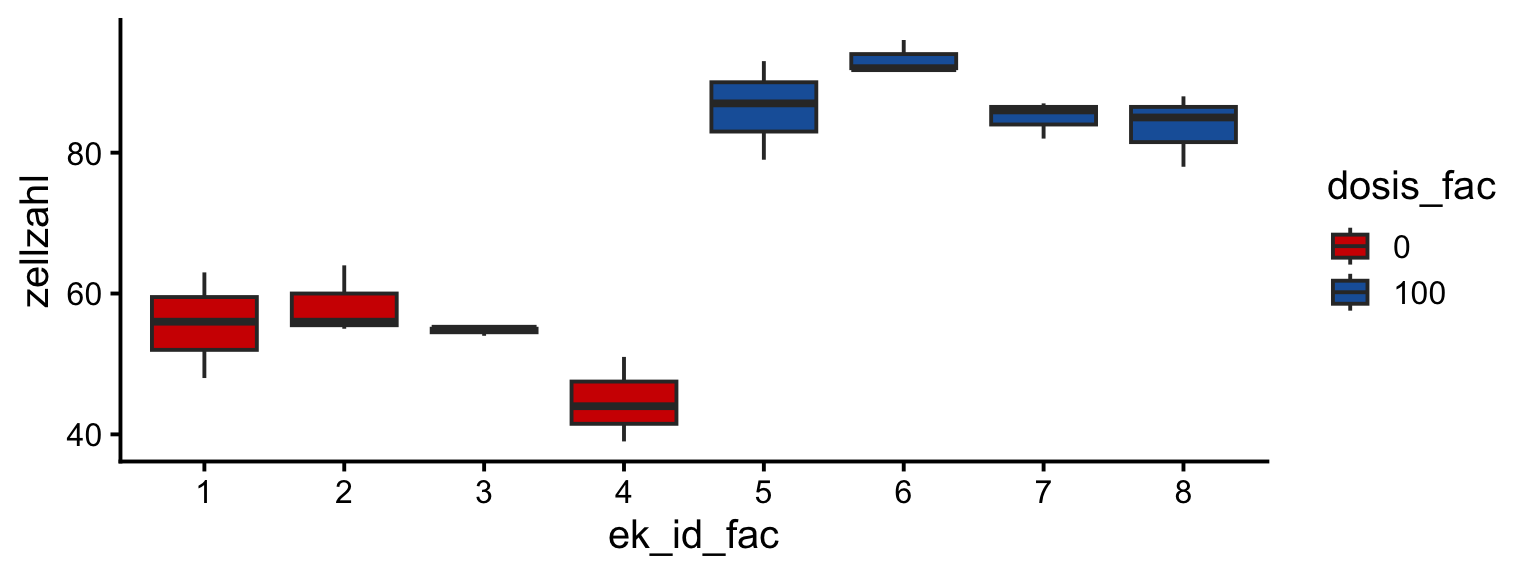
CRD - 1 Faktor mit Unterprobe | Falscher Ansatz
![]()
- Die Freiheitsgerade der Residuen betragen hier 22!
- Die FG zur Berechnung des F- und p-Werts der Behandlungsvariable (Dosis) dürfen nicht höher sein als die experimentellen Einheiten selbst (hier 8).
- Dadurch ist der F-Wert viel höher und der p-Wert viel niedriger als korrekt wäre.
CRD - 1 Faktor mit Unterprobe | Ansatz 1
![]()
1-faktorielle ANOVA mit gemittelten Unterprobewerten
Df Sum Sq Mean Sq F value Pr(>F)
dosis_fac 1 2278 2278.1 83.87 9.54e-05 ***
Residuals 6 163 27.2
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1- Hier wird das Problem der Pseudoreplikation durch die Mittelwertbildung gelöst.
Übungen
Übungswoche 1
![]()
Summary Measure Analysis’ der DS2 Fallstudie
Vorbereitung @home (s. Handbuch)
- Auffrischungsquiz (siehe Moodle)
- Fallstudiendaten zu Aufgabe 2.2 (Mehrstichprobentest) vorbereiten:
- Neues Skript bzw. Notebook erstellen.
- Benötigte Daten in R laden und aufbereiten.
- Mittelwertbildung der Kronblattbreite für jede Pflanze.
Fragen..??

Total gelangweilt?

Dann testen Sie doch Ihr Wissen in folgendem Abschlussquiz…
Abschlussquiz
![]()

Bei weiteren Fragen: saskia.otto(at)uni-hamburg.de

Diese Arbeit is lizenziert unter einer Creative Commons Attribution-ShareAlike 4.0 International License mit Ausnahme der entliehenen und mit Quellenangabe versehenen Abbildungen.
Kurswebseite: Data Science 3