Rangsummenbildung
# A tibble: 3 × 2
Species sum_ranks
<fct> <dbl>
1 setosa 1482
2 versicolor 4132.
3 virginica 5710.
Data Science 2
Saskia Otto & Monika Eberhard
Universität Hamburg, IMF & IZS
Sommersemester 2025
Nach Abschluss dieser VL und Übung..
![]()
Hat der Käfigtyp einen Einfluss auf das mittlere Gewicht des Atlantischen Lachses (Salmo salar) bzw. gibt es Unterschiede im durchschnittlichen Lachsgewicht zwischen den vier getesteten Käfigtypen?

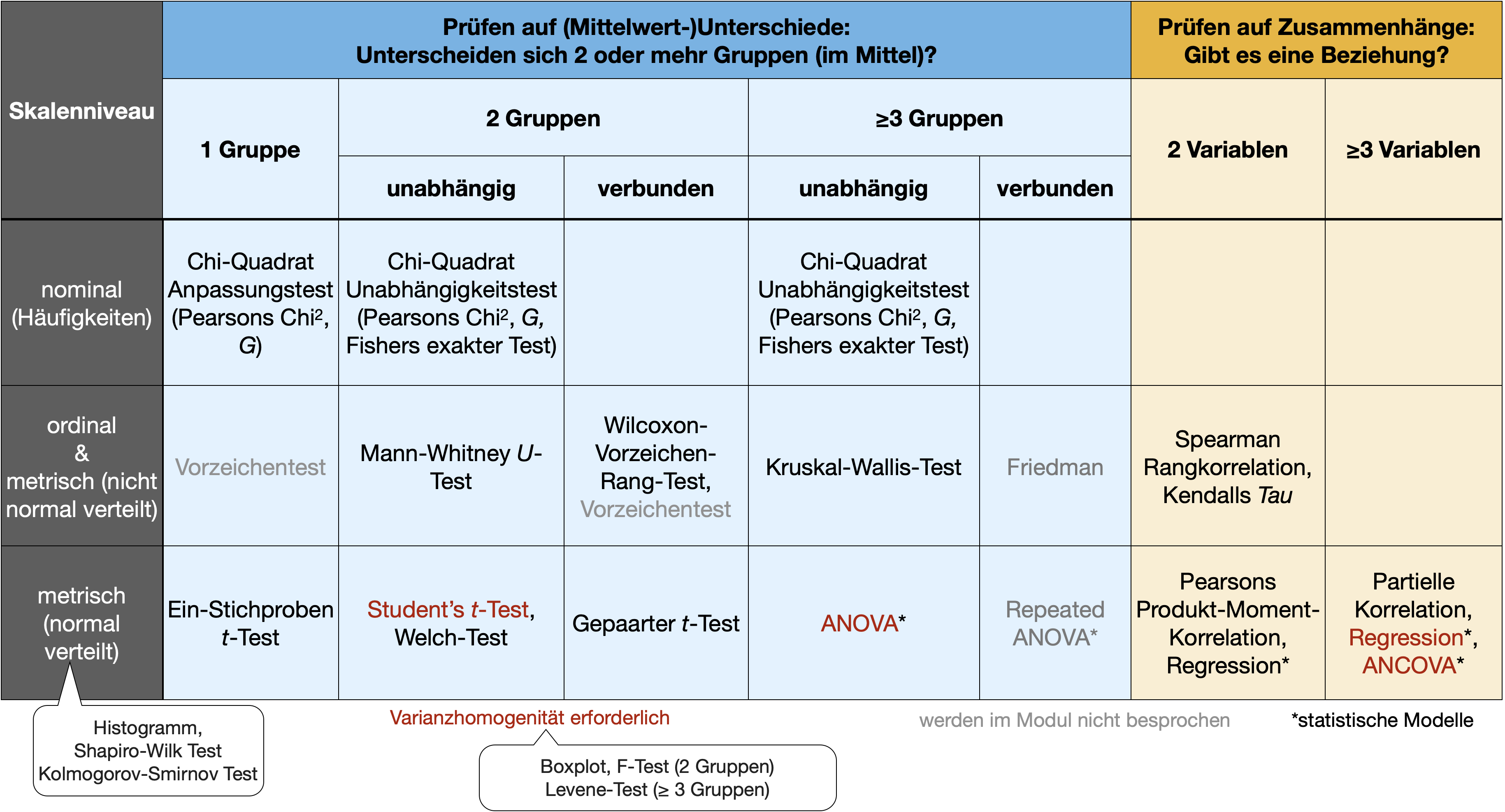


‘Familywise Error Rate’ (FWER)

H=\frac{12}{N*(N+1)}*\left( \sum\frac{R_i^2}{n_i} \right)-3*(N+1)
N = Gesamter Stichprobenumfang, n_i = Anzahl Messung pro Gruppe i, R_i = Summe der Ränge pro Gruppe i

Rangsummenbildung
# A tibble: 3 × 2
Species sum_ranks
<fct> <dbl>
1 setosa 1482
2 versicolor 4132.
3 virginica 5710.kruskal.test()
Vektorsyntax
Kruskal-Wallis rank sum test
data: iris$Sepal.Length and iris$Species
Kruskal-Wallis chi-squared = 97, df = 2, p-value <2e-16F_r=\left( \frac{12}{n*k*(k+1)}*\left( \sum R_i^2\right)\right)-3*n*(k+1)
n = Anzahl der Blöcke, k = Anzahl der Gruppen, R_i = Summe der Ränge pro Gruppe i
![]()
01:30
![]()
Es soll die Hypothese getestet werden, dass sich die durchschnittliche Kronblattweite der drei Iris Arten unterscheidet.
Kruskal-Wallis rank sum test
data: Petal.Width by Species
Kruskal-Wallis chi-squared = 131, df = 2, p-value <2e-16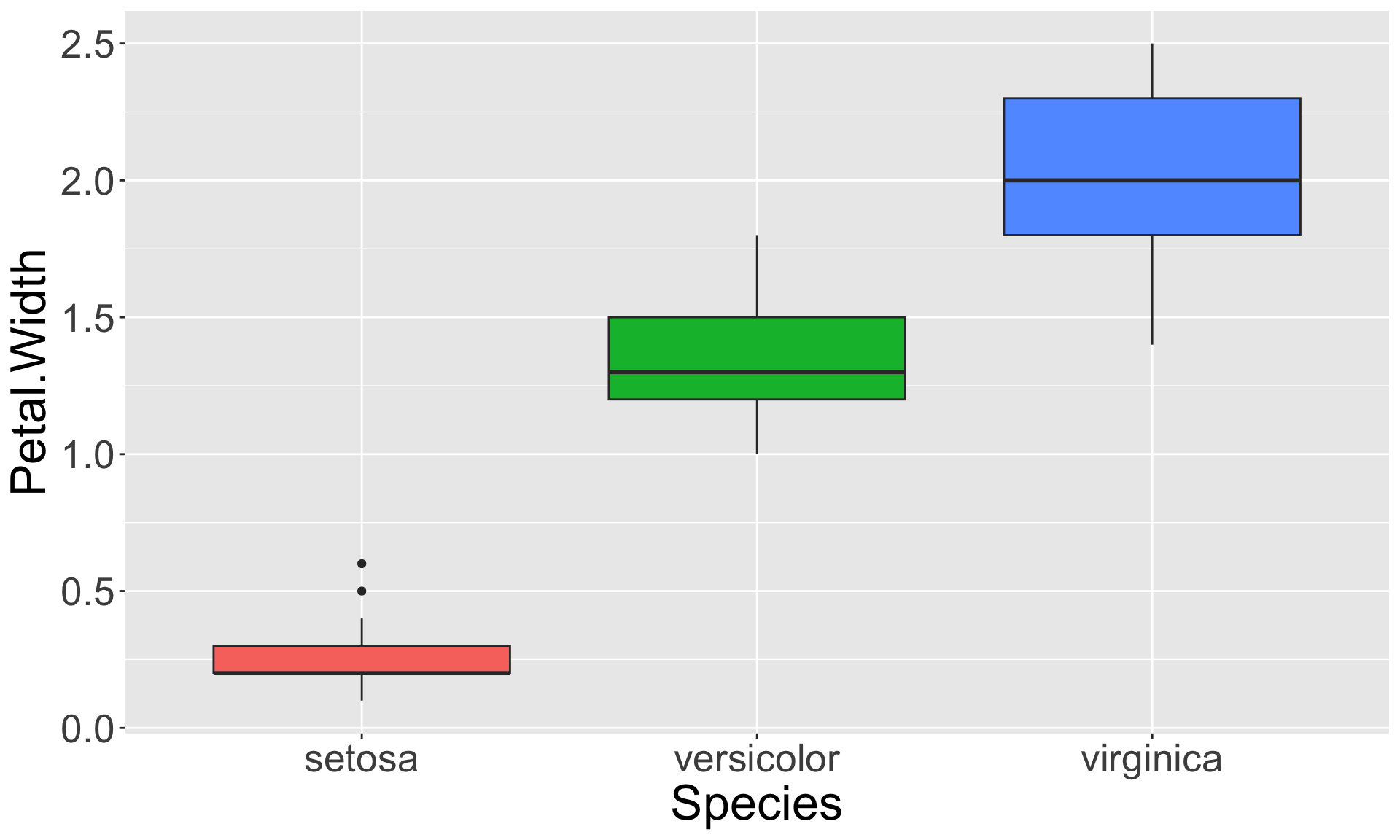

factor definieren!![]()

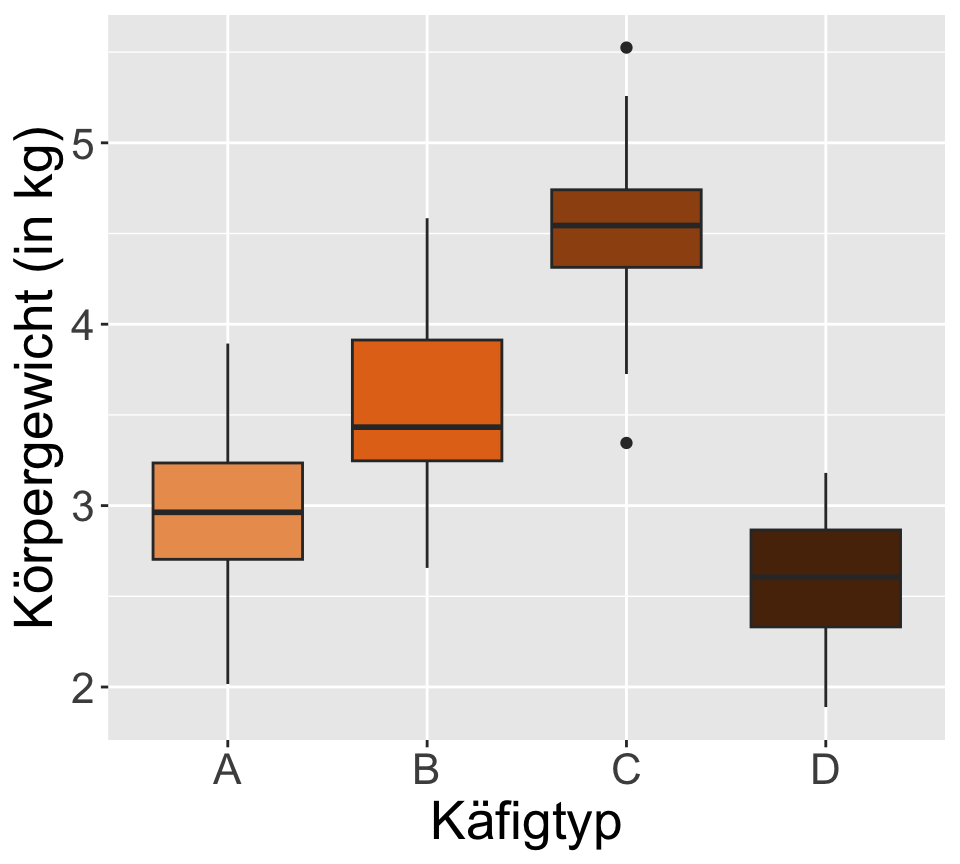
| Replikat | Typ A | Typ B | Typ C | Typ D |
|---|---|---|---|---|
| 1 | 2.0 | 2.7 | 2.9 | 2.2 |
| 2 | 2.7 | 2.0 | 1.9 | 2.0 |
| 3 | 2.2 | 3.8 | 2.7 | 3.1 |
| 4 | 3.1 | 1.5 | 2.5 | 2.7 |
| … | … | … | … | … |
| Gruppenmittelwert | 2.5 | 2.5 | 2.5 | 2.5 |
| Gruppenvarianz | 0.25 | 0.99 | 0.19 | 0.25 |
| Replikat | Typ A | Typ B | Typ C | Typ D |
|---|---|---|---|---|
| 1 | 5.0 | 4.1 | 2.9 | 0.8 |
| 2 | 5.0 | 4.1 | 2.9 | 0.8 |
| 3 | 5.0 | 4.1 | 2.9 | 0.8 |
| 4 | 5.0 | 4.1 | 2.9 | 0.8 |
| … | … | … | … | … |
| Gruppenmittelwert | 5.0 | 4.1 | 2.9 | 0.8 |
| Gruppenvarianz | 0 | 0 | 0 | 0 |
| Sources of variation | SS | df | MS | F |
|---|---|---|---|---|
| Groups (between) | n_i\sum_{i=1}^{p}(\bar{y_i}-\bar{y})^2 | p-1 | \frac{SS_{Groups}}{(p-1)} | \frac{MS_{Groups}}{MS_{Residuals}} |
| Residuals (within) | \sum_{i=1}^{p}\sum_{j=1}^{n}(y_{ij}-\bar{y_i})^2 | p(n-1) | \frac{SS_{Residuals}}{p(n-1)} | |
| Total | \sum_{i=1}^{p}\sum_{j=1}^{n}(y_{ij}-\bar{y})^2 | pn-1 |
![]()
| Sources of variation | SS | df | MS | F | p |
|---|---|---|---|---|---|
| Groups | 50.66 | 4-1 | 16.888 | 83.77 | <2e-16 |
| Residuals (or within) | 18.55 | 4*(24-1) | 0.202 | ||
| Total | 69.21 | 4*24-1 |

aov()![]()
Nachdem sich zeigte, dass alle Annahmen erfüllt sind, kann die ANOVA durchgeführt werden:
Testen auf Normalverteilung INNERHALB JEDER Stichprobe mittels Shapiro-Wilk-Test:
Shapiro-Wilk normality test
data: salmon$weight[salmon$cages == "A"]
W = 1, p-value = 0.7
Shapiro-Wilk normality test
data: salmon$weight[salmon$cages == "B"]
W = 1, p-value = 0.8
Shapiro-Wilk normality test
data: salmon$weight[salmon$cages == "C"]
W = 1, p-value = 0.8
Shapiro-Wilk normality test
data: salmon$weight[salmon$cages == "D"]
W = 1, p-value = 0.5→ Es kann bei allen vier Tests die H0 angenommen werden: die Daten sind jeweils normal verteilt.
Testen auf Varianzhomogenität ZWISCHEN den Stichproben mittels Levene-Test:
Levene's Test for Homogeneity of Variance (center = median)
Df F value Pr(>F)
group 3 0.53 0.66
92 → Die H0 kann angenommen werden: die Daten sind varianzhomogen.
'data.frame': 96 obs. of 2 variables:
$ cages : Factor w/ 4 levels "A","B","C","D": 1 1 1 1 1 1 1 1 1 1 ...
$ weight: num 2.72 2.88 3.78 3.04 3.06 ... cages weight
1 A 2.72
2 A 2.88
3 A 3.78
4 A 3.04
5 A 3.06
6 A 3.86
7 A 3.23
8 A 2.37
9 A 2.66
10 A 2.78
11 A 3.61
12 A 3.18
13 A 3.20
14 A 3.06
15 A 2.72
16 A 3.89
17 A 3.25
18 A 2.02
19 A 3.35
20 A 2.76
21 A 2.47
22 A 2.89
23 A 2.49
24 A 2.64
25 B 3.19
26 B 2.66
27 B 3.92
28 B 3.58
29 B 2.93
30 B 4.13
31 B 3.71
32 B 3.35
33 B 3.95
34 B 3.94
35 B 3.91
36 B 3.84
37 B 3.78
38 B 3.47
39 B 3.35
40 B 3.31
41 B 3.15
42 B 3.40
43 B 2.87
44 B 4.58
45 B 4.10
46 B 2.94
47 B 3.30
48 B 3.27
49 C 4.89
50 C 4.46
51 C 4.63
52 C 4.49
53 C 4.48
54 C 5.18
55 C 4.39
56 C 5.26
57 C 3.73
58 C 4.79
59 C 4.56
60 C 4.61
61 C 4.69
62 C 4.25
63 C 4.33
64 C 3.99
65 C 3.96
66 C 4.65
67 C 4.72
68 C 4.53
69 C 4.96
70 C 5.53
71 C 4.25
72 C 3.35
73 D 3.00
74 D 2.15
75 D 2.16
76 D 3.01
77 D 2.36
78 D 1.89
79 D 2.59
80 D 2.43
81 D 2.50
82 D 2.69
83 D 2.31
84 D 2.82
85 D 2.39
86 D 2.67
87 D 3.05
88 D 2.72
89 D 2.34
90 D 3.07
91 D 3.00
92 D 2.77
93 D 2.62
94 D 2.19
95 D 3.18
96 D 2.20![]()
Diese Diagnostikplots sind bei der 1-faktoriellen ANOVA allerdings nicht so wichtig.
\eta^2 = \frac{SS_{Groups}}{SS_{Total}}
![]()
plot.design()Alternative Formulierung der H0

\alpha_{adj} = \frac{\text{Signifikanzniveau}~\alpha}{\text{Anzahl der durchgeführten Tests}} \Rightarrow \frac{0.05}{5} = 0.01
p.adjust() wandelt unkorrigierte p-Werte in korrigierte mittels Bonferroni-, Bonferroni-Holm- und weiteren Korrekturen um.![]()
Zuerst müssen die p-Werte der paarweisen t-Tests extrahiert werden:
# Datensatz ins weite Format bringen
sw <- salmon |> mutate(id = rep(1:24, 4)) |>
pivot_wider(names_from = "cages", values_from = "weight")
# p-Werte der paarweisen t-Tests im Vektor speichern
p_vals <- c(
t.test(sw$A, sw$B, equal.var = TRUE)$p.value,
t.test(sw$A, sw$C, equal.var = TRUE)$p.value,
t.test(sw$A, sw$D, equal.var = TRUE)$p.value,
t.test(sw$B, sw$C, equal.var = TRUE)$p.value,
t.test(sw$B, sw$D, equal.var = TRUE)$p.value,
t.test(sw$C, sw$D, equal.var = TRUE)$p.value
)![]()
Nun kann die p.adjust() Funktion angewendet werden:
TukeyHSD()![]()
Tukey multiple comparisons of means
95% family-wise confidence level
Fit: aov(formula = weight ~ cages, data = salmon)
$cages
diff lwr upr p adj
B-A 0.530 0.191 0.8691 0.001
C-A 1.532 1.193 1.8715 0.000
D-A -0.408 -0.747 -0.0687 0.012
C-B 1.002 0.663 1.3415 0.000
D-B -0.938 -1.277 -0.5987 0.000
D-C -1.940 -2.279 -1.6010 0.000![]()
![]()
![]()

Bei weiteren Fragen: saskia.otto(at)uni-hamburg.de

Diese Arbeit is lizenziert unter einer Creative Commons Attribution-ShareAlike 4.0 International License mit Ausnahme der entliehenen und mit Quellenangabe versehenen Abbildungen.
Kurswebseite: Data Science 2