
7-Klassische Tests zum Prüfen auf Unterschiede: nominale Daten
Data Science 2
Saskia Otto & Monika Eberhard
Universität Hamburg, IMF & IZS
Sommersemester 2025
Lernziele
Nach Abschluss dieser VL und Übung..
- können Sie eine Verteilung auf ihre Anpassungsgüte mit dem Pearson Chi-Quadrat und dem G-Test testen.
- können Sie zwei Variablen auf Unabhängigkeit mit dem Pearson Chi-Quadrat testen.
- wissen Sie, welche Voraussetzungen für den Pearson Chi-Quadrat geprüft werden müssen und wann Sie besser die Yates Korrektur oder den Exakten Test nach Fisher anwenden.
Heutige Fragen
Sind Mitarbeiter des Gesundheitswesens, die keine COVID-19 Vorerkrankung hatten, verstärkt geimpft worden?



Unterscheiden sich zwei Tannenhäher- und Eichelhäherarten in ihrer Häufigkeit in verschiedenen Waldtypen?
Bildquellen: Wikipedia (Tannenhäher unter (CC BY-SA 2.0 Lizenz) und Eichelhäher unter CC BY-SA 4.0 Lizenz)
Vergleich von Häufigkeiten

Tests für nominale Daten

Häufigkeitsdaten in der Biologie
- Viele biologische Daten liegen in Form von Zählungen (ganzen Zahlen, im Englischen count data) für ein oder mehrere Merkmale vor:
- Anzahl vorkommender vs. abwesender Arten
- Anzahl überlebender vs. verstorbener Tiere
- die Anzahl der Äste an einem Baum
- Anzahl der Beobachtungen verschiedener Phänotypen (z.B. runde grüne Erbsen, runzlige gelbe Erbsen, ..)

Bildquelle: Ravindran, 2016, PNAS
Beobachtete vs. erwartete Häufigkeiten
Frage, die sich meist stellt:
- Weichen die beobachten Werte von den zu erwartenden Werten ab?
- Sind zwei oder mehr Merkmale voneinander unabhängig oder weisen die beobachten Werte auf eine systematische Abhängigkeit hin?
Der Chi-Quadrat-Test | 1
- bezeichnet eine Gruppe von Hypothesentests mit Chi-Quadrat-verteilter Testprüfgröße.
- wird in folgende Typen unterteilt:
Grundtypen
- Anpassungstest (im Englischen goodness-of-fit test) → prüft, ob vorliegende Daten auf eine bestimmte Weise verteilt sind.
- Unabhängigkeitstest (contingency table test) → prüft, ob zwei oder mehr Merkmale stochastisch unabhängig sind.
- Homogenitätstest → prüft, ob zwei oder mehr Stichproben derselben Verteilung bzw. einer homogenen Grundgesamtheit entstammen.
Der Chi-Quadrat-Test | 2
Es gibt drei Varianten die Signifikanz der Unterschiede zwischen beobachteten und erwarteten Häufigkeiten zu beurteilen:
Testvarianten
- Chi-Quadrat-Test nach Pearson (mit oder ohne Yates Korrektur)
- G-Test (auch Log-Likelihood-Test genannt)
- kann jederzeit anstelle des Pearsons \chi^2-Test angewendet werden.
- soll sensitiver sein, allerdings eher zum Fehler 1. Art führen.
- Exakter Test nach Fisher (Fisher’s exact test)
Chi-Quadrat-Tests in R

Built-in Funktion chisq.test()
Anpassungtest: 1 Merkmal

Pearsons Chi-Quadrat-Test \chi ^2 vs. G-Test
Vorgehensweise
- Erwartete Häufigkeit muss anhand von Wahrscheinlichkeiten berechnet werden.
- Vergleich mit beobachteter Häufigkeit
- Berechnung des \chi ^2 – bzw. G-Wertes.
- Vergleich des \chi ^2- bzw. G-Wertes mit dem kritischen \chi ^2-Wert bei (n-1) Freiheitsgraden und einer Signifikanzschwelle von \alpha = 0.05. Es wird immer 1-seitig getestet1!
\chi^2=\sum\frac{(Beobachtungswert_i - Erwartungswert_i)^2}{Erwartungswert_i} \; \text{ und } \; G = 2\sum_{i=1}^{n}B_i\cdot ln\frac{B_i}{E_i}
Beispiel mit gleichen Wahrscheinlichkeiten | Daten
![]()
300maliges Würfeln eines 6-seitigen Würfels
| Augenzahl | Beobachtet | Erwartet |
|---|---|---|
| 1 | 59 | |
| 2 | 33 | |
| 3 | 48 | |
| 4 | 51 | |
| 5 | 65 | |
| 6 | 44 | |
| Gesamt (N) | 300 |
Beispiel mit gleichen Wahrscheinlichkeiten | Erwartungswerte
![]()
300maliges würfeln eines 6-seitigen Würfels
| Augenzahl | Beobachtet | Erwartet (P(X)*N) |
|---|---|---|
| 1 | 59 | 1/6*300 = 50 |
| 2 | 33 | 1/6*300 = 50 |
| 3 | 48 | 1/6*300 = 50 |
| 4 | 51 | 1/6*300 = 50 |
| 5 | 65 | 1/6*300 = 50 |
| 6 | 44 | 1/6*300 = 50 |
| Gesamt (N) | 300 | 300 |
H0: Es gibt keinen Unterschied zwischen den beobachteten und erwarteten Werten.
Beispiel mit gleichen Wahrscheinlichkeiten | Kennwertberechnung
![]()
\chi^2=\frac{(59-50)^2}{50}+\frac{(33-50)^2}{50}+\frac{(48-50)^2}{50}+\frac{(51-50)^2}{50}+\frac{(65-50)^2}{50}+\frac{(44-50)^2}{50} = 12.72
G = 2(59\cdot ln\frac{59}{50}+33\cdot ln\frac{33}{50}+48\cdot ln\frac{48}{50}+51\cdot ln\frac{51}{50}+65\cdot ln\frac{65}{50}+44\cdot ln\frac{44}{50}) = 13.07
Beispiel mit gleichen Wahrscheinlichkeiten | Signifikanz
![]()
Vergleich mit kritischem \chi^2-Wert
- Unser \chi^2 ist größer als der obere kritische \chi^2-Wert → wir können unsere H0 ablehnen.
- Die Interpretation für unseren G-Wert ist die gleiche → H0 wird abgelehnt.
Beispiel mit gleichen Wahrscheinlichkeiten | Automatische Berechnung
![]()
Beispiel mit ungleichen Wahrscheinlichkeiten | Daten
Kreuzungsexperiment von Mendel
Bei einem Kreuzungsexperiment an Erbsen beobachtete Gregor Mendel folgende Phänotypen:
| Augenzahl | Beobachtet |
|---|---|
| rund-gelbe Erbsen | 79 |
| rund-grüne Erbsen | 27 |
| runzlig-gelbe Erbsen | 24 |
| runzlig-grüne Erbsen | 8 |
| Gesamt | 138 |
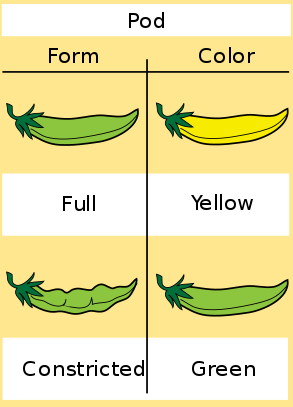
Bildquelle: Wikipedia - Mariana Ruiz (CC0 Lizenz)
Beispiel mit ungleichen Wahrscheinlichkeiten | Erwartungswerte
![]()
Kreuzungsexperiment von Mendel
Aufgrund seines genetischen Modells erwartete er eine Verteilung der Nachkommen im Verhältnis 9:3:3:1:
| Augenzahl | Beobachtet | Erwartet |
|---|---|---|
| rund-gelbe Erbsen | 79 | 9/16*138 = 77.625 |
| rund-grüne Erbsen | 27 | 3/16*138 = 25.875 |
| runzlig-gelbe Erbsen | 24 | 3/16*138 = 25.875 |
| runzlig-grüne Erbsen | 8 | 1/16*138 = 8.625 |
| Gesamt | 138 | 138 |
- Da es 4 Kategorien gibt, ist n = 4 und die Anzahl der Freiheitsgrade FG = n-1 = 3.
Beispiel mit ungleichen Wahrscheinlichkeiten | Signifikanz
![]()
Berechnung des Kennwerts
Beispiel mit ungleichen Wahrscheinlichkeiten | Built-in Funktion
![]()
- Es gibt keinen Unterschied zwischen den beobachteten und erwarteten Werten (\chi^2 = 0.254, df = 3, p = 0.97).
Unabhängigkeitstest: Vergleich von 2 oder mehr Merkmalen

Kontingenztafeln
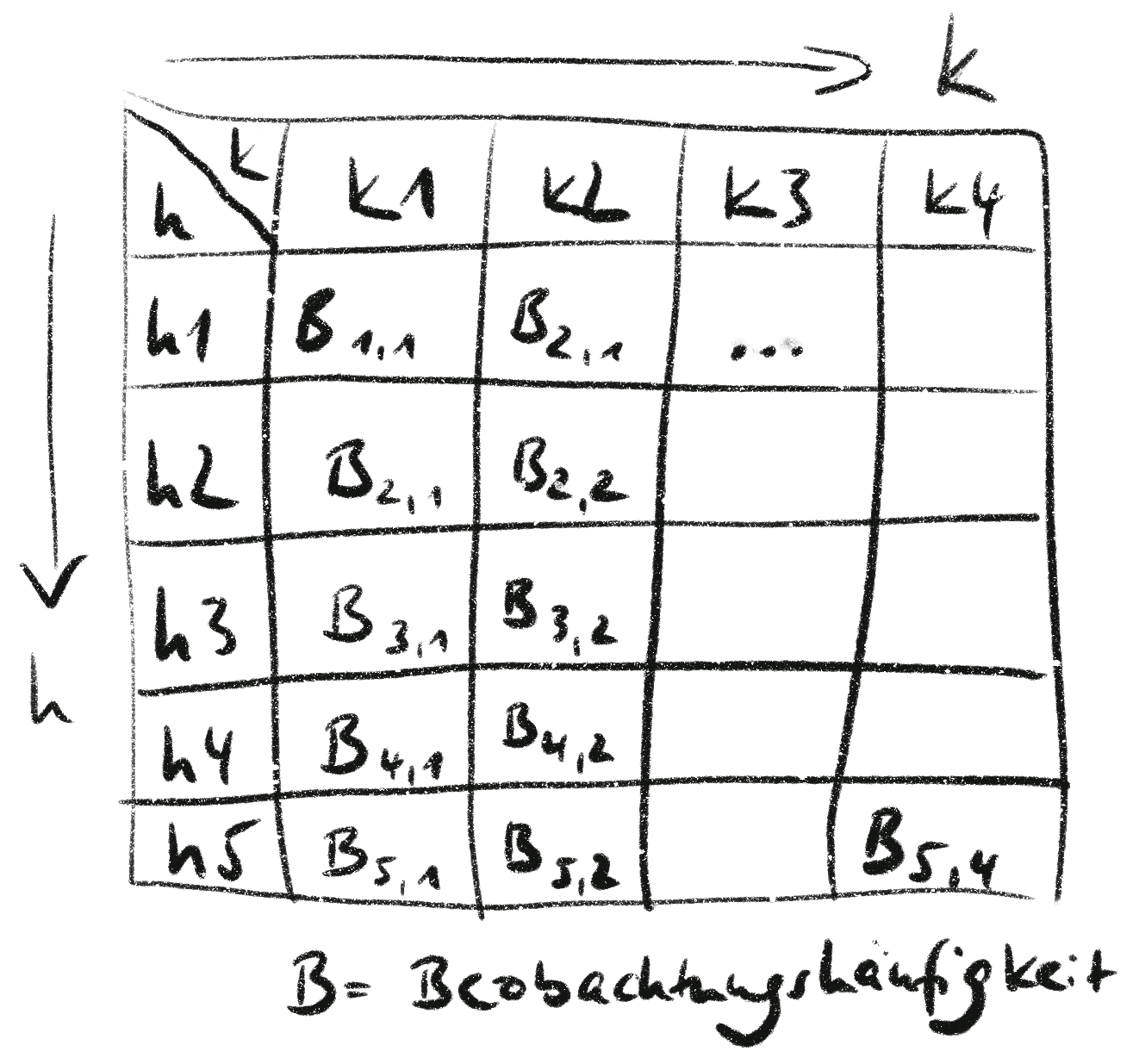
- 1 Merkmal: Einfach-Klassifizierung → nur 1 Zeile bzw. Spalte
- 2 Merkmale: Zweifach-Klassifizierung → h Zeilen und k Spalten mit (h-1)(k-1) Freiheitsgraden
- In der Statistik sind Kontingenzen alle Ereignisse, die möglicherweise eintreten können.
- Eine Kontingenztafel zeigt die Anzahl, wie oft jede der Kontingenzen in einer bestimmten Stichprobe tatsächlich beobachtet wurde.
Pearsons Chi-Quadrat-Test
Beispiel: COVID-19-Impfstoffabdeckung bei Mitarbeitern des Gesundheitswesens in England
![]()
Sind Mitarbeiter des Gesundheitswesens, die keine COVID-19 Vorerkrankung hatten, verstärkt geimpft worden?
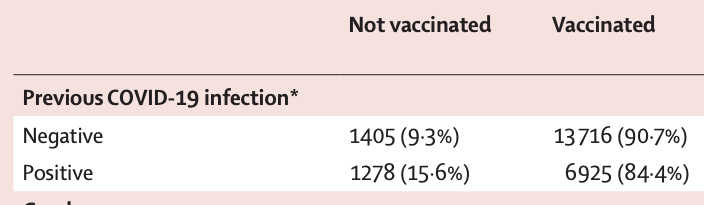
H0: Es gibt keinen Zusammenhang zwischen der Impfrate und COVID-19 Vorerkrankungen, die Verteilungen in beiden Merkmalen sind unabhängig voneinander.

Artikel: Hall et al., 2021, The Lancet
Beispiel: COVID-19-Impfstoffabdeckung | Daten
![]()
Beobachtungswerte (aus Tabelle 1)
| COVID-19 Vorerkrankung | Nicht Geimpft | Geimpft | Gesamt |
|---|---|---|---|
| Negativ | 1405 | 13716 | 15121 |
| Positiv | 1278 | 6925 | 8203 |
| Gesamt | 2683 | 20641 | 23324 |
Beispiel: COVID-19-Impfstoffabdeckung | Wahrscheinlichkeiten 1
![]()
| COVID-19 Vorerkr. | Nicht Geimpft | Geimpft | Gesamt |
|---|---|---|---|
| Negativ | P(neg. \cap nicht~geimpft) | P(neg. \cap geimpft) | 15121 |
| Positiv | P(positiv \cap nicht~geimpft) | P(positiv \cap geimpft) | 8203 |
| Gesamt | 2683 | 20641 | 23324 |
- Unter der H0 sollte die spezielle Multiplikationsregel (Regel 5) angewendet werden können, sprich Ereignisse A und B sind voneinander unabhängig → P(A \cap B) = P(A)*P(B)
- Nun müssen die Wahrscheinlichkeiten nur noch aus den Randhäufigkeiten ermittelt werden, z.B:
- 15121 von insgesamt 23324 hatten keine Vorerkrankung → P(negativ)=\frac{15121}{23324}= 0.648
- 20641 von insgesamt 23324 wurden geimpft → P(geimpft)=\frac{20641}{23324}= 0.885
- Somit ist P(negativ \cap geimpft) = \frac{15121}{23324}*\frac{20641}{23324} = 0.5737269
Beispiel: COVID-19-Impfstoffabdeckung | Wahrscheinlichkeiten 2
![]()
| COVID-19 Vorerkr. | Nicht Geimpft | Geimpft | Gesamt |
|---|---|---|---|
| Negativ | \frac{15121}{23324}*\frac{2683}{23324} = 0.0746 | \frac{15121}{23324}*\frac{20641}{23324} = 0.5737 | 15121 |
| Positiv | \frac{8203}{23324}*\frac{2683}{23324} = 0.0404 | \frac{8203}{23324}*\frac{20641}{23324} = 0.3112 | 8203 |
| Gesamt | 2683 | 20641 | 23324 |
- Der Erwartungswert ist entsprechend E = P * N
Beispiel: COVID-19-Impfstoffabdeckung | Erwartungswerte
![]()
| COVID-19 Vorerkr. | Nicht Geimpft | Geimpft | Gesamt |
|---|---|---|---|
| Negativ | 0.0746*N \approx 1740 | 0.5737*N \approx 13381 | 15121 |
| Positiv | 0.0404*N \approx 943 | 0.3112*N \approx 7260 | 8203 |
| Gesamt | 2683 | 20641 | 23324 (N) |
- Wichtig ist, dass die Randhäufigkeiten für Erwartungs- und Beobachtungswerte erhalten bleiben: kontingent heißt auch ‘abhängig von’ oder ‘bedingt durch’.
- Die Formel lässt sich sogar noch verkürzen: E_{negativ, nicht~geimpft} = \frac{Zeilensumme* Spaltensumme}{Gesamtsumme} = \frac{15121*2683}{23324}
Beispiel: COVID-19-Impfstoffabdeckung | Berechnung
![]()
Manuell (\chi^2 und G)
Freiheitsgrade hier: (Anzahl Zeilen der Kontingenztafel - 1)*(Anzahl Spalten der Kontingenztafel - 1)
Voraussetzungen
beim Pearsons Chi-Quadrat-Test (und G-Test)
- Es können nur Häufigkeitsverteilungen getestet werden, keine Proportionen oder Prozentsätze.
- Alle Erwartungswerte sollten mind. ≥ 2 sein, mindestens 80% sollten ≥ 5 sein.
- \chi^2 reagiert bei kleinen Erwartungswerten sehr empfindlich auf kleine Abweichungen.
- Dadurch zunehmende Gefahr eines statistischen Fehler 2. Art.
- Errechneter \chi^2-Wert basiert auf beschränkten Datenmengen, wird aber zur Signifikanz mit kontinuierlichen Verteilung verglichen
- Bei kleinen Stichproben (N<20) empfielt sich Stetigkeitskorrektur nach Yates (Standardeinstellung in R mit
correct = TRUE)
- Bei kleinen Stichproben (N<20) empfielt sich Stetigkeitskorrektur nach Yates (Standardeinstellung in R mit
\chi^2_{korrigiert} = \sum\frac{(|B_i-E_i|-0.5)^2}{E_i}
Stetigkeitskorrektur nach Yates | COVID-19 Bsp.
![]()
- Nachteil der Korrektur soll allerdings sein, dass Werte konservativ sind (p-Werte sind tendenziell höher) → einige empfehlen daher nur bei df = 1 die Korrektur.
Your turn …
![]()
03:00
Quiz zum Nachmachen | Kelchblattbreite
![]()
Ausgangssituation
Die Messungen der Kelchblattbreite im iris Datensatzes lassen sich in folgende drei Größenklassen einteilen:
Code
df <- iris |>
# Mit cut() können wir eine metrische Variable kategorial machen:
mutate(sw_class = cut(Sepal.Width, breaks = 3) ) |>
group_by(Species, sw_class) |>
# shortuct für summarise(n = n())
count() |>
pivot_wider(names_from = Species, values_from = n,
values_fill = 0)
df$sw_class <- c("< 2.8 cm", "2.8-4 cm", "> 4 cm")
sw_sizeclass <- as.matrix(df[ ,2:4])
rownames(sw_sizeclass) <- df$sw_class
knitr::kable(df, format = "html", col.names = c(
"Größenklasse", "I. setosa", "I. versicolor", "I. virginica")) |>
kableExtra::kable_styling(position = "center", font_size = 20)| Größenklasse | I. setosa | I. versicolor | I. virginica |
|---|---|---|---|
| < 2.8 cm | 1 | 27 | 19 |
| 2.8-4 cm | 36 | 23 | 29 |
| > 4 cm | 13 | 0 | 2 |
Pearson Chi-Quadrat
Nun wird ermittelt, ob das Größenmerkmal und die Artzugehörigkeit voneinander abhängen:
Quiz zum Nachmachen | Frage 1
![]()
Pearson Chi-Quadrat
Nun wird ermittelt, ob das Größenmerkmal und die Artzugehörigkeit voneinander abhängen:
Quiz zum Nachmachen | Frage 2
![]()
Pearson Chi-Quadrat
Nun wird ermittelt, ob das Größenmerkmal und die Artzugehörigkeit voneinander abhängen:
Quiz zum Nachmachen | Frage 3
![]()
Pearson Chi-Quadrat
Nun wird ermittelt, ob das Größenmerkmal und die Artzugehörigkeit voneinander abhängen:
Exakter Test nach Fisher

Alternativen, wenn Voraussetzung nicht erfüllt ist
Was tun, wenn beim Pearson’s \chi^2-Test oder beim G-Test zu viele der erwarteten Häufigkeiten <5 sind?
- Klassen zusammenfassen
- Exakter Test nach Fisher (Fisher’s Exact Test)
Exakter Test nach Fisher
- Von Ronal A. Fisher 1934 der Royal Statistical Society vorgestellt → kam allerdings zu der Zeit nicht an.
- Heute allgemeingültiger Ansatz der gerade bei kleinen bis mittelgroßen Stichproben genutzt wird.
- Bei großen Stichproben (> 500) soll sich das Verfahren asymptotisch einem \chi^2-Test mit einem FG annähern.
- Mit diesem Permutationstest können exakte p-Werte berechnet werden (der Pearson’s \chi^2- sowie G-Test sind nur Annäherungen).
Exakter Test nach Fisher | Wahrscheinlichkeit
| Merkmale M1 und M2 | + | - | Summe |
|---|---|---|---|
| + | a | b | a + b |
| - | c | d | c + d |
| Summe | a + c | b + d | a + b + c + d = n |
Die Wahrscheinlichkeit für ein bestimmtes Ergebnis ist gegeben durch
p=\frac{(a+b)!(c+d)!(a+c)!(b+d)!}{a!b!c!d!n!}
Bsp: Habitatpräferenz | Daten & Wahrscheinlichkeit
![]()
Unterscheiden sich zwei Tannenhäher- und Eichelhäherarten in ihrer Häufigkeit in verschiedenen Waldtypen?
Beobachtungen
| Waldtyp | Tannenhäher | Eichelhäher | Summe |
|---|---|---|---|
| Nadelwald | 6 | 2 | 8 |
| Laubwald | 4 | 8 | 12 |
| Summe | 10 | 10 | 20 |
Bsp: Habitatpräferenz | Wahrscheinlichkeit extremer Werte
![]()
Wir müssen die Wahrscheinlichkeit von Ergebnissen berechnen, die noch extremer sind als dieses. Es gibt zwei davon (bei gleichen Randsummen):
Ergebnisvariante 1
| Waldtyp | TH | EH | Summe |
|---|---|---|---|
| Nadelwald | 7 | 1 | 8 |
| Laubwald | 3 | 9 | 12 |
| Summe | 10 | 10 | 20 |
Ergebnisvariante 2
| Waldtyp | TH | EH | Summe |
|---|---|---|---|
| Nadelwald | 8 | 0 | 8 |
| Laubwald | 2 | 10 | 12 |
| Summe | 10 | 10 | 20 |
Bsp: Habitatpräferenz | Ergebnis
![]()
Diese 3 Wahrscheinlichkeiten müssen nun addiert werden und dann mit 2 multipliziert werden für einen 2-seitigen Test:
Shortcut: Die built-in Funktion fisher.test()
Fragen?
Abschlussquiz
![]()

Bei weiteren Fragen: saskia.otto(at)uni-hamburg.de

Diese Arbeit is lizenziert unter einer Creative Commons Attribution-ShareAlike 4.0 International License mit Ausnahme der entliehenen und mit Quellenangabe versehenen Abbildungen.
Kurswebseite: Data Science 2