6-Klassische Tests zum Prüfen auf Unterschiede: metrische/ordinale Daten
Data Science 2
Saskia Otto & Monika Eberhard
Universität Hamburg, IMF & IZS
Sommersemester 2025
Lernziele
Nach Abschluss dieser VL und Übung..
- können Sie auf Unterschiede zwischen zwei Mittelwerten für unabhängige UND gepaarte Stichproben testen.
- wissen sie wann sie den parametrische t-Test verwenden können und wann besser den nicht-parametrischen Mann-Whitney U-Test bzw. Wilcoxon-Vorzeichen-Rang-Test.
Heutige Fragen
Unterscheidet sich das durchschnittliche Zugverhalten zwischen Buchfinken und Mönchsgrasmücken?



Singen Kohlmeisen in der Stadt durchschnittlich lauter als auf dem Land?
Bildquellen rechts oben: Wikipedia (Buchfink unter (CC BY-SA 2.5 Lizenz) und Mönchsgrasmücke unter CC0 Lizenz)
Bildquelle links unten: Luc Viator-Wikimedia (CC-BY-SA 2.0 Lizenz)
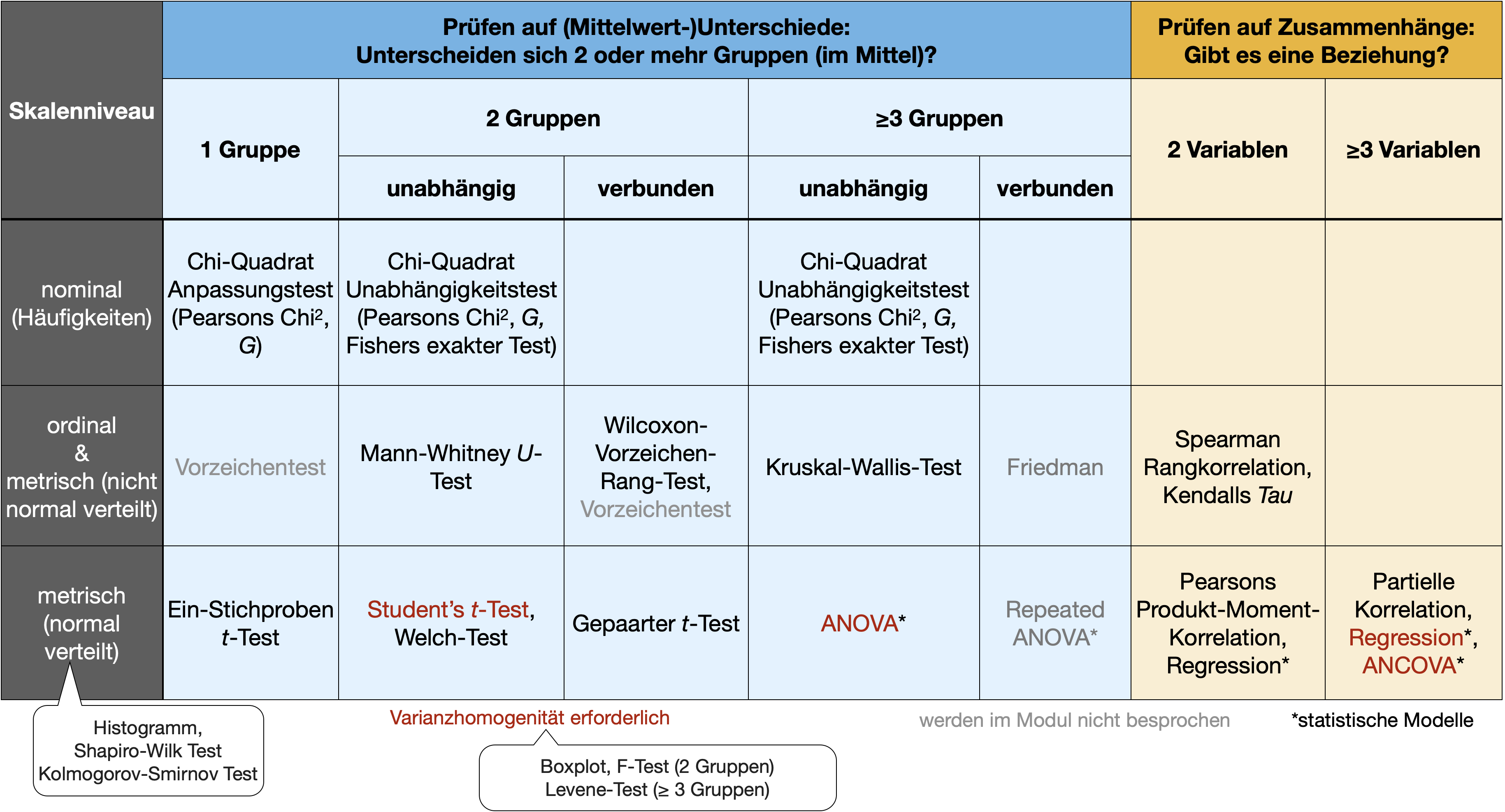
t-Test: Parametrischer Ein- und Zweistichprobentest

Mittelwertvergleich bei metrischen Daten

Zur Erinnerung
Parametrische Tests
- Verteilungsabhängig
- Arbeiten mit \bar{X}, s^2, s
- Nur für metrische Daten
- Annahmen:
- Unabhängigkeit (außer gepaarter t-Test)
- Varianzhomogenität (gleiche Varianzen)
- Normalität (Normalverteilung)
- Tests:
- F-Test,
- t-Test für 1 und 2 Stichproben
- Varianzanalyse (ANOVA), Kovarianzanalyse (ANCOVA)
- Pearson Produkt-Moment-Korrelationskoeffizient, lineare Regression
Prüfen der Annahmen
- Schritt 1 - Normalverteilung:
- Grafisch: Histogramm, Boxplot
- Test: Shapiro-Wilk-Test
- Schritt 2 - Varianzhomogenität:
- Grafisch: Boxplot
- Test: F-Test
t-Test in R

Built-in Funktion t.test()
t-Test für 1 Stichprobe
- Hier wird getestet, ob sich der Mittelwert einer einzelnen Stichprobe von einem vorgegebenen Populationsmittelwert \mu_0 unterscheidet.
- Die Teststatistik t folgt einer t-Verteilung.
Formel der Kenngröße t
t=\frac{\bar{X}-\mu_0}{s_{\bar{X}}}=\frac{\bar{X}-\mu_0}{\frac{s}{\sqrt{n}}}\Rightarrow~~\text{wenn}~\mu_0 = 0: \frac{\bar{X}}{\frac{s}{\sqrt{n}}}
- \bar{X} = Stichprobenmittelwert
- \mu_0 = theoretischer Populationsmittelwert
- s_{\bar{X}} = Standardschätzfehler des Mittelwerts
Hypothesen
- zweiseitig:
- H_0: \mu = \mu_0
- H_A: \mu \neq \mu_0
- linksseitig:
- H_0: \mu \geq \mu_0
- H_A: \mu < \mu_0
- rechtsseitig:
- H_0: \mu \leq \mu_0
- H_A: \mu > \mu_0
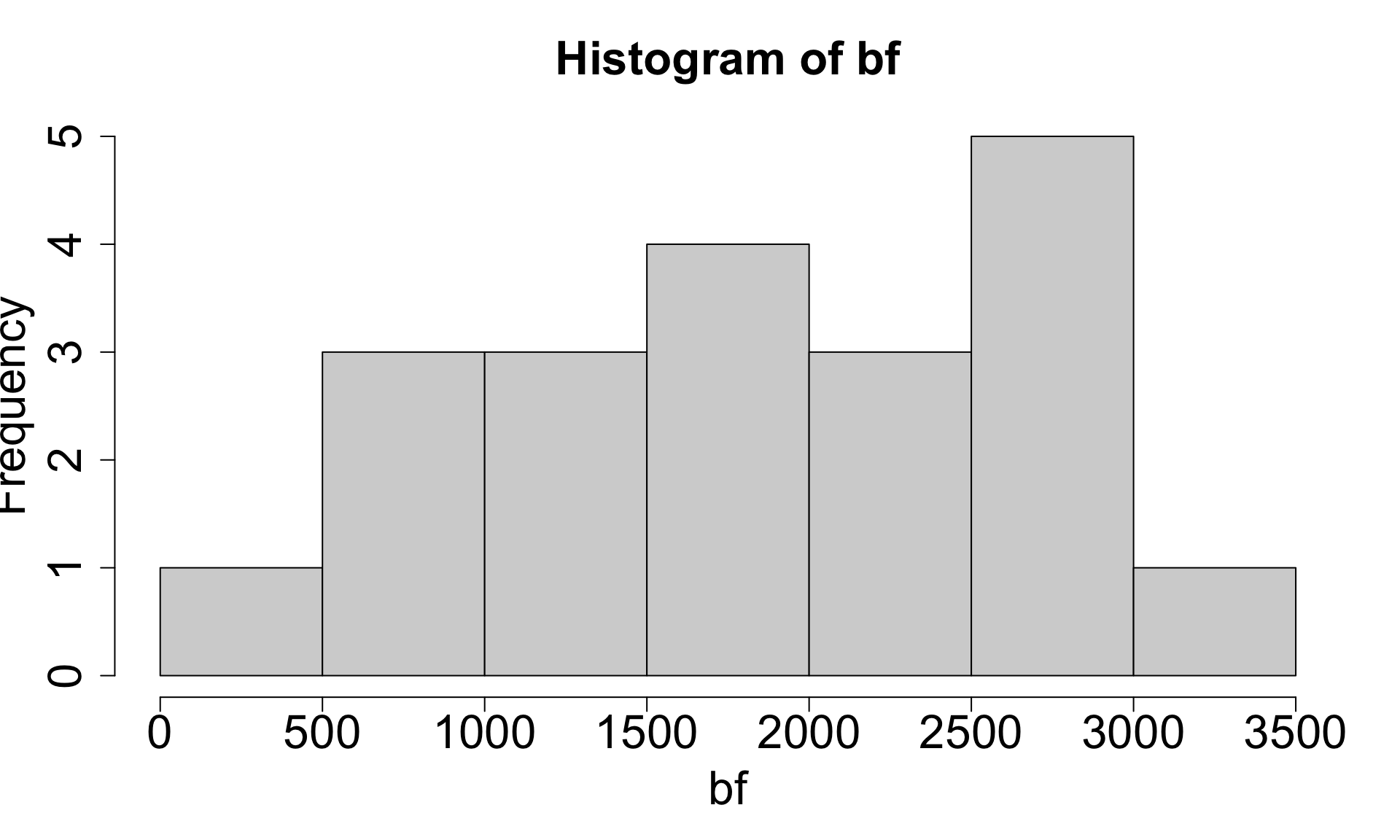
t-Test für 1 Stichprobe | Bsp. Zugverhalten 1

Forschungsfrage
Unterscheidet sich unsere Stichprobe zum Zugverhalten des Buchfinks (BF1) von einer anderen Stichprobe (BF2), für die wir nur den Mittelwert kennen?
Bekannte Statistiken
| Kenngröße | BF1 | BF2 |
|---|---|---|
| Mittelwert \bar{X} | 1800 km | 1697 km |
| Standardabw. s | ±900 km | ? |
| Stichprobengröße n | 20 | ? |
Die 8 Schritte
| Kennwert: | \mu bzw. \bar{X} |
| H0: | BF1 = BF2 |
| HA: | BF1 \neq BF2 |
| Voraussetzung: | Normalverteilung |
| Teststatistik: | t |
| alpha: | 0.05 |
| FG: | n-1 = 19 |
| p-Wert: | Vergleich t-Wert mit t_{krit} aus einer t-Verteilung |
Bildquelle: Wikipedia (CC BY-SA 2.5 Lizenz)
t-Test für 1 Stichprobe | Bsp. Zugverhalten 2

t-Test für 1 Stichprobe | Bsp. Zugverhalten 3

t-Test für 1 Stichprobe | Bsp. Zugverhalten 4

- Die Teststatistik t liegt zwischen dem unteren und oberen t_{krit}, somit kann die H_0 nicht verworfen werden.
- Anders ausgedrückt:
- Die Wahrscheinlichkeit ein statistisches Ergebnis wie das beobachtete zu erhalten, wenn die Nullhypothese wahr wäre, liegt bei 61.5%.
- Somit können wir die H_0 nicht ablehnen (erst bei <5%).
ZUSAMMENFASSUNG: → Es gibt keinen signifikanten Unterschied zwischen unserer Stichprobe und der Vergleichsstichprobe (t = 0.51, df = 19, p > 0.5).
t-Test bei 2 unabhängigen Stichproben | 1
Student’s t-Test bei GLEICHEN Varianzen
- Der am häufigsten verwendete Zweistichproben-Test.
- Der Stichprobenumfang beider Gruppen darf ungleich sein.
- Theoretisch kann der t-Test auch bei sehr kleinen Stichprobenumfängen (z. B. 10) verwendet werden.
- Testet die Nullhypothese, dass die Mittelwerte von zwei Populationen gleich sind:
- H_0: \mu_1 = \mu_2 bzw. \mu_1-\mu_2=0
t-Test bei 2 unabhängigen Stichproben | 2
Student’s t-Test bei GLEICHEN Varianzen
Formel der Kenngröße t
t=\frac{(\bar{X}_1-\bar{X}_2) -(\mu_1 - \mu_2)}{s_p \sqrt{\frac{1}{n_1} + \frac{1}{n_2}}} \Rightarrow~~\text{wenn}~~\mu_1-\mu_2=0:~t = \frac{(\bar{X}_1-\bar{X}_2)}{s_p \sqrt{\frac{1}{n_1} + \frac{1}{n_2}}}
s_p = \text{Standardabweichung der gepoolten Stichprobe:}
s_p = \sqrt{\frac{(n_1-1)s_1^2 + (n_2 -1)s_2^2}{(n_1-1) + (n_2-1)}} \text{wenn } n \text{ identisch ist: }s_p = \sqrt{\frac{s_1^2 + s_2^2}{2}}
t-Test bei 2 unabhängigen Stichproben | 3
Welch’s t-Test bei UNGLEICHEN Varianzen
- Was ist, wenn die Varianzen nicht gleich sind?
- → gepoolte Schätzung der Standardabweichung ungeeignet
- → t-Verteilung mit (n_1-1)+(n_2-1) Freiheitsgraden keine gute Annäherung an die Stichprobenverteilung
- Anpassung der Teststatistik t und der Freiheitsgrade:
Formel der Kenngröße t
t=\frac{(\bar{X}_1-\bar{X}_2) -(\mu_1 - \mu_2)}{\sqrt{\frac{s_1^2}{n_1} + \frac{s_2^2}{n_2}}}
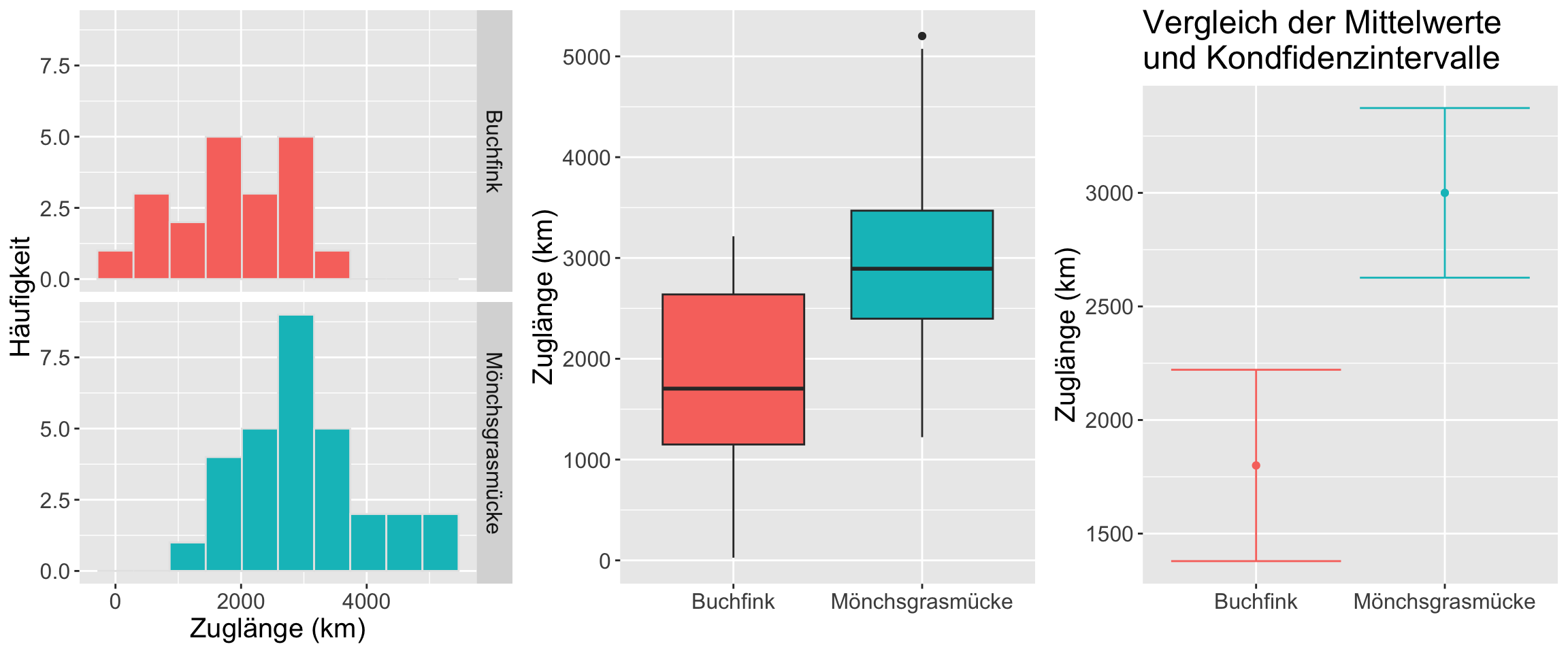
Bsp. Buchfinken vs. Mönchsgrasmücken | 1
Unterscheidet sich das durchschnittliche Zugverhalten zwischen Buchfinken und Mönchsgrasmücken?


| Kenngröße | Buchfink | Mönchsgrasmücke |
|---|---|---|
| Mittelwert x̅ | 1800 km | 3000 km |
| Standardabweichung s | ±900 km | ±1000 km |
| Stichprobengröße n | 20 | 30 |
Bildquellen: Wikipedia (Buchfink unter (CC BY-SA 2.5 Lizenz) und Mönchsgrasmücke unter CC0 Lizenz)
Bsp. Buchfinken vs. Mönchsgrasmücken | 2
Testen der Annahmen
Shapiro-Wilk normality test
data: bf
W = 1, p-value = 0.5
Shapiro-Wilk normality test
data: mgm
W = 1, p-value = 0.6→ Die H0 wird jeweils angenommen, da p > 0.05: beide Stichproben sind normal verteilt.
F test to compare two variances
data: bf and mgm
F = 0.8, num df = 19, denom df = 29, p-value = 0.6
alternative hypothesis: true ratio of variances is not equal to 1
95 percent confidence interval:
0.363 1.946
sample estimates:
ratio of variances
0.81 → Die H0 wird angenommen, da p > 0.05: die Varianzen beider Gruppen sind gleich.
Bsp. Buchfinken vs. Mönchsgrasmücken | 3
| Kennwert: | \mu bzw. \bar{X} |
| H0: | \mu_{BF} = \mu_{MGM} |
| HA: | \mu_{BF} \neq \mu_{MGM} |
| Voraussetzung: | Normalität/Varianzhomogenität |
| Teststatistik: | t = \frac{(\bar{X}_{BF}-\bar{X}_{MGM})}{s_p \sqrt{\frac{1}{n_{BF}} + \frac{1}{n_{MGM}}}} |
| alpha: | 0.05 |
| FG: | (n_{BF}-1)+(n_{MGM}-1) = 19+29 = 48 |
| p-Wert: | Vergleich t-Wert mit t_{krit} aus einer t-Verteilung |
Student’s t-Test
- Da der p-Wert mit 0.00008 < alpha (0.05) ist, kann die H_0 abgelehnt werden.
- Die Zuglänge ist bei Buchfinken signifikant kleiner als bei Mönchsgrasmücken (t = -4.3, df = 48, p < 0.001).
Bsp. Buchfinken vs. Mönchsgrasmücken | 4
Manuelle Berechnung
Zur Erinnerung: t = \frac{(\bar{X}_1-\bar{X}_2)}{s_p \sqrt{\frac{1}{n_1} + \frac{1}{n_2}}}\text{ mit }s_p = \sqrt{\frac{(n_1-1)s_1^2 + (n_2 -1)s_2^2}{(n_1-1) + (n_2-1)}}
Wenn Stichproben abhängig sind

Abb. links: Fotoaufnahmen des Lake Powell, USA, am Zusammenfluss mit dem Dirty Devil River (Quelle: https://pubs.usgs.gov/fs/2004/3062/)
Gepaarter t-Test | 1
2 abhängige Stichproben
- Testet die Nullhypothese, dass die Differenz zwischen den gepaarten Beobachtungen Null ist:
- H_0: d = \mu_1-\mu_2 = 0
Vorgehensweise
- Berechnung der Differenzen der gepaarten Beobachtung.
- Berechnung des Mittelwerts und des Standardschätzfehlers aus diesen Differenzen.
Formel der Kenngröße
t = \frac{\bar{d}-\mu_d}{s_{\bar{d}}} = \frac{\bar{d}-\mu_d}{\frac{s}{\sqrt{n}}}
\text{mit }FG = n-1
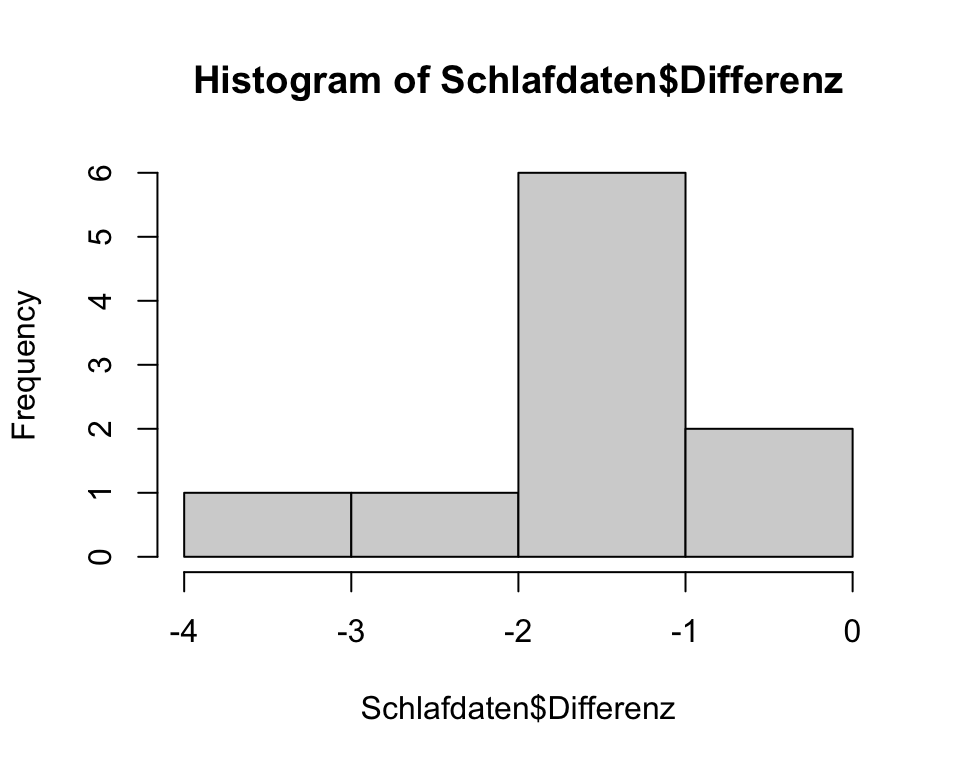
Bsp. Pharmazeutische Schlafstudie | 1
| Patient | Medikament1 | Medikament2 | Differenz |
|---|---|---|---|
| 1 | 0.7 | 1.9 | -1.2 |
| 2 | -1.6 | 0.8 | -2.4 |
| 3 | -0.2 | 1.1 | -1.3 |
| 4 | -1.2 | 0.1 | -1.3 |
| 5 | -0.1 | -0.1 | 0.0 |
| 6 | 3.4 | 4.4 | -1.0 |
| 7 | 3.7 | 5.5 | -1.8 |
| 8 | 0.8 | 1.6 | -0.8 |
| 9 | 0.7 | 4.6 | -3.9 |
| 10 | 2.0 | 3.4 | -1.4 |
Bsp. Pharmazeutische Schlafstudie | 2
Prüfung auf Normalverteilung
- Da sich der gepaarte t-Test auf die paarweisen Differenzen bezieht, muss diese normal verteilt sein!
- Die Varianzhomogeniät zwischen den beiden Gruppen fällt entsprechend weg.
Bsp. Pharmazeutische Schlafstudie | 3
Anwendung des gepaarten t-Tests
Paired t-test
data: Schlafdaten$Medikament1 and Schlafdaten$Medikament2
t = -5, df = 9, p-value = 0.001
alternative hypothesis: true mean difference is not equal to 0
95 percent confidence interval:
-2.258 -0.762
sample estimates:
mean difference
-1.51 - Da der p-Wert < 0.05 ist, kann die H_0 abgelehnt werden.
- → Es gibt einen sign. Unterschied in der Wirksamkeit zwischen den beiden Medikamenten (t = -4.06, FG = 9, p = 0.003). Im Durchschnitt schliefen die Probanden 1.58 Stunden länger mit Medikament 2.
Bsp. Pharmazeutische Schlafstudie | 4
Vergleich zum 1-Stichproben t-Test
- Im gepaarten t-Test betrachten wir eigentlich nur eine Stichprobe - die der Differenzen.
- Die gemittelte Differenz wird dann gegen den Referenzwert Null verglichen (zur Erinnerung: H_0: d = \mu_1-\mu_2 = 0).
- Somit erhalten wir das gleiche Ergebnis, wenn wir den 1-Stichproben t-Test mit der Differenz Vektor und einem mu von 0 durchführen:
Übersicht der t-Test Anwendungen

- Einstichproben t-Test, zweiseitige Hypothese (Standard):
t.test(x, mu)
- Unabhängiger Zweistichproben-t-Test (gleiche Varianzen), zweiseitig: H_A:µ1 ≠ µ2
t.test(x, y, var.equal = TRUE)
- Unabhängiger Zweistichproben-t-Test (gleiche Varianzen), einseitig: H_A:µ1 > µ2:
t.test(x, y, var.equal = TRUE, alternative = “greater”)
- Welch-Test (ungleiche Varianzen = default), einseitig: H_A:µ1 < µ2
t.test(x, y, alternative = “less”)
- Gepaarter Zweistichproben-t-Test (gleiche Varianzen) , zweiseitige Hypothese:
t.test(x, y, paired = TRUE, var.equal = TRUE)
- Gepaarter t-Test mit 2 Stichproben (ungleiche Varianzen) , einseitig: H_A:µ1 > µ2
t.test(x, y, paired = TRUE, alternative = “greater”)
Übersicht der Hypothesen bei den t-Tests

Your turn …
![]()
03:00
Quiz 1 | Testwahl
Kelchblattlänge in iris
![]()
Ausgangssituation:
- Es soll die Hypothese getestet werden, dass die durchschnittliche Kelchblattlänge der Art Iris setosa kleiner ist als bei der Art Iris virginica.
- Beide Stichproben sind normal verteilt (jeweils W = 0.97, p > 0.05).
- Die Varianzen beider Stichproben sind signifikant unterschiedlich (F_{(49,49)}=3.25, p < 0.001).
Quiz 2 | Funktionsaufruf
Kelchblattlänge in iris
![]()
Quiz 3 | Interpretation
Kelchblattlänge in iris
![]()
Nicht-Parametrische Tests - Teil 1

Vergleich zweier Stichproben mit ordinalen und metrischen (nicht normal verteilten) Daten


Zur Erinnerung
Nicht-Parametrische Tests
- Verteilungsfrei (keine Normalverteilung erforderlich)
- Arbeiten mit Median und Rängen
- Für ordinale, und nominale Daten
- Metrische Daten → Transformation in Rangdaten (ordinale Daten)
- Teststärke ist allgemein niedriger
- Nicht geeignet bei komplexem Design
- Tests:
- → Mann-Whitney U-Test
- → Wilcoxon-Vorzeichen-Rang-Test
- Chi-Quadrat-Tests
- Kruskal-Wallis-Test, Friedman-Test
- Rangkorrelation (z.B. Spearman, Kendall)
Äquivalent zum t-Test
- 2 unabhängige Stichproben
- Mann-Whitney U-Test
- = Mann-Whitney-Wilcoxon (MWW)-Test
- = Wilcoxon Rangsummentest (Wilcoxon Rank Sum Test)
- Teststärke nur 95% im Vergleich zum unabhängigen Zweistichproben-t-Test
- 2 gepaarte Stichproben
- Wilcoxon-Vorzeichen-Rang-Test
- Engl. Wilcoxon Signed Rank Test
- Teststärke nur 95% im Vergleich zum gepaarten Zweistichproben-t-Test
Beide ‘Wilcoxon’ Tests in R
Built-in Funktion wilcox.test()
Mann-Whitney U | Berechnung
- Bilden der Ränge für die Werte beider Stichproben zusammen1 (N=m+n).
- Bilden der Rangsummen für beide Gruppe2 (R_x und R_y)
- Berechnen von U_x und U_y:
- U_x = m*n+\frac{m(m+1)}{2}-R_x
- U_y = m*n+\frac{n(n+1)}{2}-R_y oder U_y = m*n-U_x
- Das kleinere der errechneten U nehmen3: U_{min} = min(U_x, U_y)
Mann-Whitney U | Berechnung
- Bilden der Ränge für die Werte beider Stichproben zusammen1 (N=m+n).
- Bilden der Rangsummen für beide Gruppe2 (R_x und R_y)
- Berechnen von U_x und U_y:
- U_x = m*n+\frac{m(m+1)}{2}-R_x
- U_y = m*n+\frac{n(n+1)}{2}-R_y oder U_y = m*n-U_x
- Das kleinere der errechneten U nehmen3: U_{min} = min(U_x, U_y)
Mann-Whitney U | Kohlmeisen Beispiel
Studie


…Great tits in cities sing faster and at a higher pitch compared to their conspecifics dwelling in forests, as reported in this issue by Slabbekoorn and den Boer-Visser [6]. They suggest that the birds changed their songs to make them stand out against the masking traffic noise in urban areas. ..
Bildquelle männliche Kohlmeise: Luc Viator-Wikimedia (CC-BY-SA 2.0 Lizenz)
Kohlmeisen Beispiel | Eigene Erhebung
Ausgangssituation
Vorgehensweise
- Messen von verschiedenen Individuen in der Stadt (m = 6) auf dem Land (n = 5).
- Sehr kleine Stichproben → Normalverteilung schwer testbar.
Fragestellung
- Singen Kohlmeisen in der Stadt lauter als auf dem Land?
- 1-seitige H_A: \mu_{M(Stadt)} > \mu_{M(Land)} bzw. \mu_{M(Stadt)} - \mu_{M(Land)}>0
- Unterscheiden sich die Gesänge von Meisen in der Stadt und auf dem Land in ihrer Lautstärke?
- 2-seitige H_A: \mu_{M(Stadt)} \neq \mu_{M(Land)} bzw. \mu_{M(Stadt)} - \mu_{M(Land)}\neq 0)
Kohlmeisen Beispiel | U-Werte
Messung
| Messort | Lautstärke (in dB) | Rang |
|---|---|---|
| Stadt | 52 | 8.5 |
| Stadt | 44 | 5 |
| Stadt | 64 | 11 |
| Stadt | 53 | 10 |
| Stadt | 37 | 4 |
| Stadt | 50 | 7 |
| Land | 47 | 6 |
| Land | 29 | 1 |
| Land | 32 | 3 |
| Land | 52 | 8.5 |
| Land | 31 | 2 |
Berechnung
Rangsummen:
R_{Stadt}=8.5+5+11+10+4+7=45.5
R_{Land}=6+1+3+8.5+2=29.5
U-Werte:
U_{Stadt}=6*5+\frac{6(6+1)}{2}-45.5 = 5.5
U_{Land}=6*5+\frac{5(5+1)}{2}-20.5 = 24.5
\Rightarrow U_{min} = U_{Stadt} = 5.5
Die Wilcoxon-Verteilung
Bestimmung von U_{krit} (bei 2-seitiger Hyopothese)
![]()
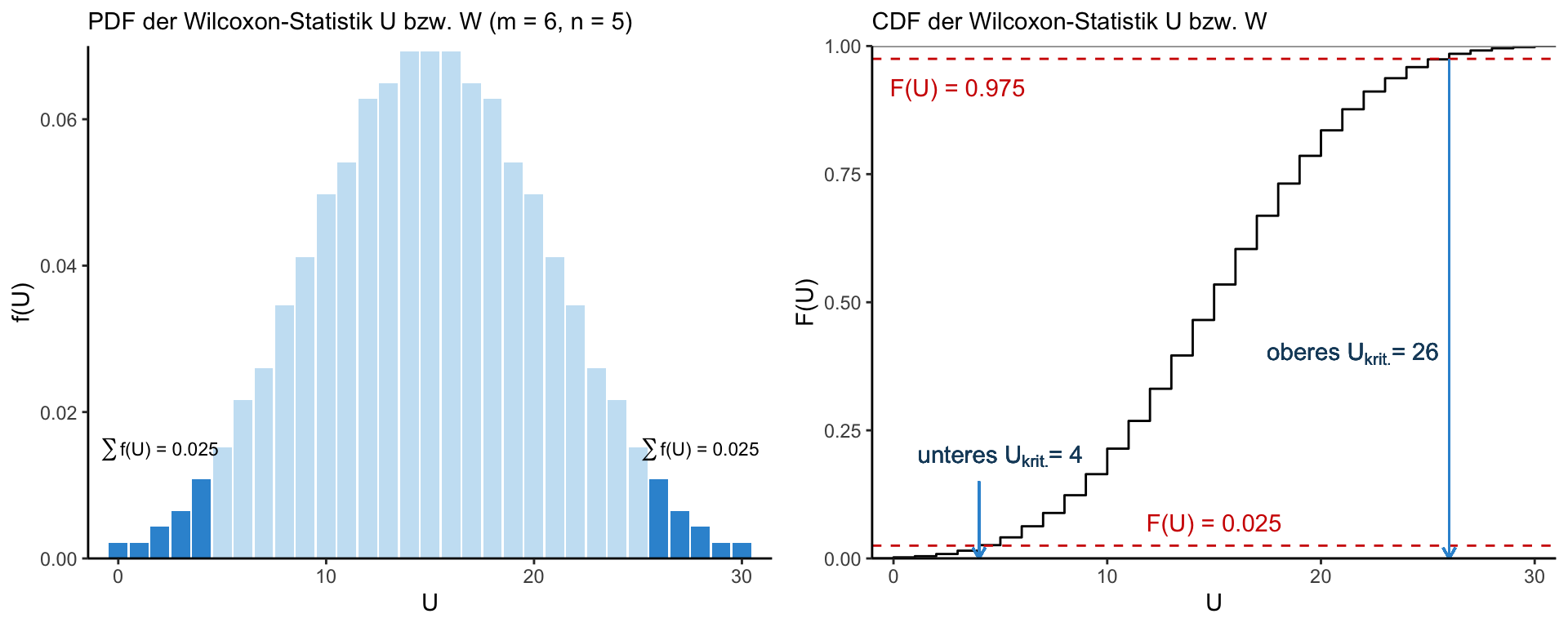
- Rangsumme der Ränge in einer Stichprobe = Zufallsvariable aus der kombinierten Rangverteilung
- Diese Rangverteilung wird Wilcoxon-Verteilung genannt und ist eine diskrete Verteilung → U_{krit} = (m, n, \alpha)
- Für große Stichproben (nicht mehr in den U-Tabellen abgebildet) wird allerdings ein Z-Wert ausgerechnet.
- Die Irrtumswahrscheinlichkeit für diesen Z-Wert kann in der Z-Tabelle nachgeschlagen bzw. in R mit
pnorm()ermittelt werden.
Kohlmeisen Beispiel | Signifikanz
Manuelle Berechnung
2-seitiger Test
[1] 4[1] 0.0823Fazit
- 2-seitiger Test:
- U_{Stadt} < U_{\alpha = 0.025} ??
→ H_A: \mu_{M(Stadt)} \neq \mu_{M(Land)} - Die beiden Gruppen unterscheiden sich nicht signifikant voneinander (2-seitiger Mann-Whitney U Test, U = 5.5, p > 0.05).
- U_{Stadt} < U_{\alpha = 0.025} ??
- 1-seitiger Test:
- U_{Stadt} < U_{\alpha = 0.05} ??
→ H_A: \mu_{M(Stadt)} > \mu_{M(Land)} - Die Lautstärke in der Stadt ist schwach signifikant höher (1-seitiger Mann-Whitney U Test, U = 5.5, p < 0.05).
- U_{Stadt} < U_{\alpha = 0.05} ??
Kohlmeisen Beispiel | Built-in Funktion 1
2-seitiger Test
Warning in wilcox.test.default(x = land, y = stadt): cannot compute exact
p-value with ties
Wilcoxon rank sum test with continuity correction
data: land and stadt
W = 6, p-value = 0.1
alternative hypothesis: true location shift is not equal to 0Warum die Warnung?
- Ein exakter p-Wert wird “standardmäßig” berechnet, wenn weniger als 50 Werte und keine gleichen Ränge (“ties”) vorhanden sind.
- Ansonsten wird eine Näherung auf Basis der Standardnormalverteilung verwendet.
Kohlmeisen Beispiel | Built-in Funktion 2
1-seitiger Test
Wilcoxon rank sum test with continuity correction
data: stadt and land
W = 24, p-value = 0.05
alternative hypothesis: true location shift is greater than 0Die Reihenfolge von x und y zählt für U (bzw. W)!
- Es wird immer der U-Wert der Stichprobe wiedergegeben, die in der Funktion dem
yArgument zugewiesen wird! - Falls dieser der größere ist, wird der obere U_{krit} verwendet.
Übersicht der Funktionsanwendungen

- Unabhängiger Zweistichproben-Test: Mann-Whitney U-Test, zweiseitige Hypothese (Standard): H_A: µ1 ≠ µ2
wilcox.test(x, y)
- Unabhängiger Zweistichproben-Test: Mann-Whitney U-Test, einseitig: H_A: µ1 > µ2:
wilcox.test(x, y, alternative = “greater”)
- Gepaarter Zweistichproben-Test: Wilcoxon-Vorzeichen-Rang-Test, zweiseitige Hypothese: H_A: µ1 ≠ µ2
wilcox.test(x, y, paired = TRUE)
- Gepaarter Zweistichproben-Test: Wilcoxon-Vorzeichen-Rang-Test, einseitig: H_A: µ1 < µ2
wilcox.test(x, y, paired = TRUE, alternative = “less”)
Mann-Whitney U-Test vs. t-Test
Vergleich Mann-Whitney U-Test vs. t-Test
Einfluss von Ausreißern
Unterscheiden sich mit Fadenwürmern infizierte und mit Interleucin-33 behandelte Mäuse von nicht behandelten Kontrollen in der Zahl adulter Fadenwürmer im Dünndarm?
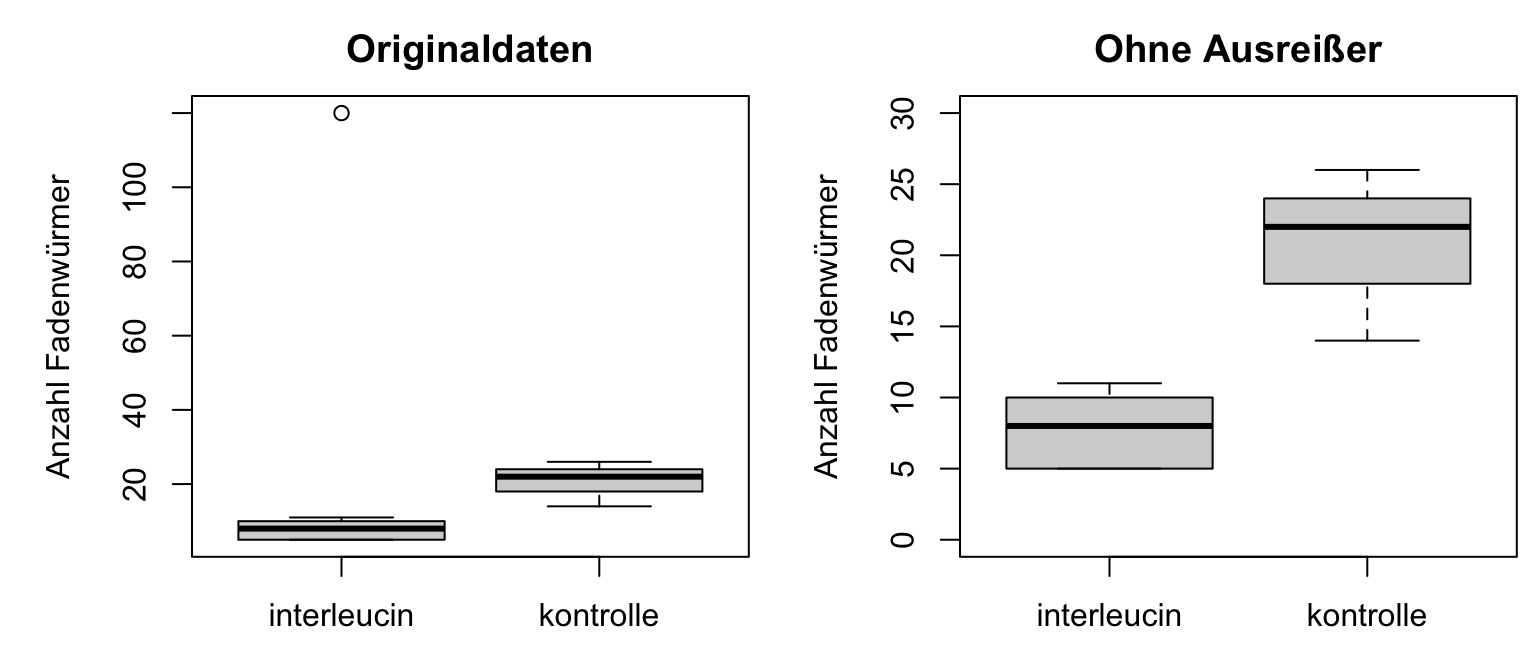
Testergebnisse mit dem Ausreißer
Mann-Whitney U-Test
→ H_0 kann abgelehnt werden. Es gibt einen signifikanten Unterschied.
Welch t-Test
Welch Two Sample t-test
data: wurm$interleucin and wurm$kontrolle
t = -0.08, df = 8, p-value = 0.9
alternative hypothesis: true difference in means is not equal to 0
95 percent confidence interval:
-29.9 27.9
sample estimates:
mean of x mean of y
20 21 → H_0 kann nicht abgelehnt werden. Es gibt keinen signifikanten Unterschied.
Testergebnisse ohne den Ausreißer
Mann-Whitney U-Test
→ H_0 kann weiterhin abgelehnt werden. Es gibt einen signifikanten Unterschied.
Welch t-Test
Welch Two Sample t-test
data: wurm$interleucin[-9] and wurm$kontrolle[-9]
t = -8, df = 12, p-value = 8e-06
alternative hypothesis: true difference in means is not equal to 0
95 percent confidence interval:
-17.08 -9.42
sample estimates:
mean of x mean of y
7.5 20.8 → Jetzt kann H_0 abgelehnt werden.
Nicht-parametrische Tests sind wesentlich weniger sensitiv gegenüber Ausreißern!
Übungsaufgaben
![]()
Übungstag 3
Einführung in die inferenzielle Statistik
![]()
- Aufgaben:
- 3.1 Hausaufgabe zur Vorbereitung:
Erstellung von Hypothesen - Korallenriffe - 3.2 Klassische Tests zum Prüfen auf Unterschiede zwischen 2 Stichproben
- 3.3 Nachbereitung Fallstudie: Frage 2.1
- 3.1 Hausaufgabe zur Vorbereitung:
- R Notebook-Skript:
- DS2_03_Uebungen_Hypothesen_Anpassungstests_Zweistichprobentests.Rmd
Fragen?
Abschlussquiz
![]()

Bei weiteren Fragen: saskia.otto(at)uni-hamburg.de

Diese Arbeit is lizenziert unter einer Creative Commons Attribution-ShareAlike 4.0 International License mit Ausnahme der entliehenen und mit Quellenangabe versehenen Abbildungen.
Kurswebseite: Data Science 2