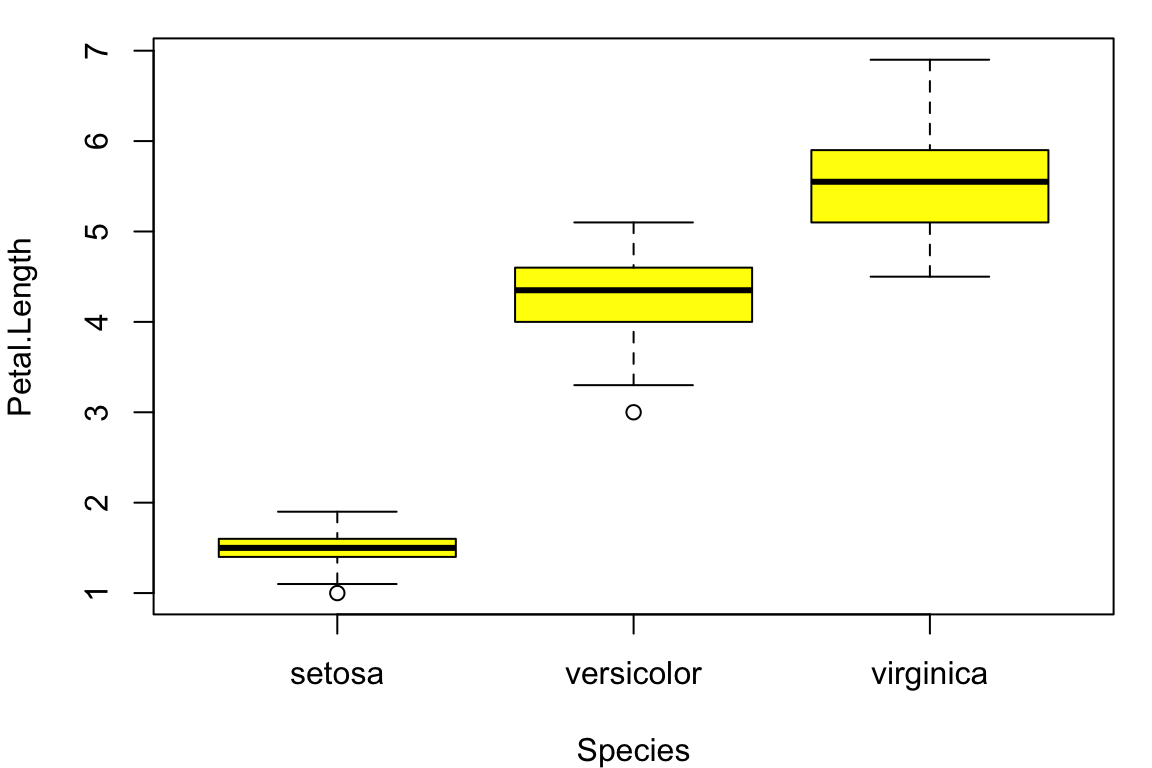Datenvisualisierung mit base R und ggplot2
Data Science 1 - Programmieren & Visualisieren
Saskia Otto & Monika Eberhard
Universität Hamburg, IMF
Wintersemester 2025/2026
Lernziele
![]()
Am Ende dieser VL- und Übungseinheit werden Sie
- einfache Diagramme zur Datenexploration mithilfe der R Basisfunktionen erstellen können.
- wissen, wie Grafiken als PDF, JPEG oder PNG Dateien gespeichert werden können.
- das Prinzip der geschichten Diagrammerstellung in ggplot2 verstanden haben.
- einen Überblick über die verschiedenen geom_XXX() Funktionen haben.
- die wichtigsten Diagrammtypen in den entsprechenden Kategorien mit ggplot2 erstellen können.
2 verbreitete Typen
Grafiken mit base R
- Funktionen sind bereits Bestandteil der Basisversion.
- Gut geeignet für eine einfache und schnelle Datenexploration.
- Nicht sehr intuitiv und weniger geeignet für komplexe Grafiken.
- Keine gute Hilfe bzw. Dokumentation.
Grafiken mit ggplot2 Paket
- Besser geeignet bei komplexeren Grafiken.
- Klarere Syntax.
- Besser dokumentiert.
- Viele Beispiele im Internet.

Grafiken mit base R
2 Funktionstypen
High-level Funktionen | Überblick
| Funktion | Beschreibung |
|---|---|
plot() |
Generische Funktion mit vielen Methoden (kontextabhängig) |
barplot() |
Säulen- bzw. Balkendiagram |
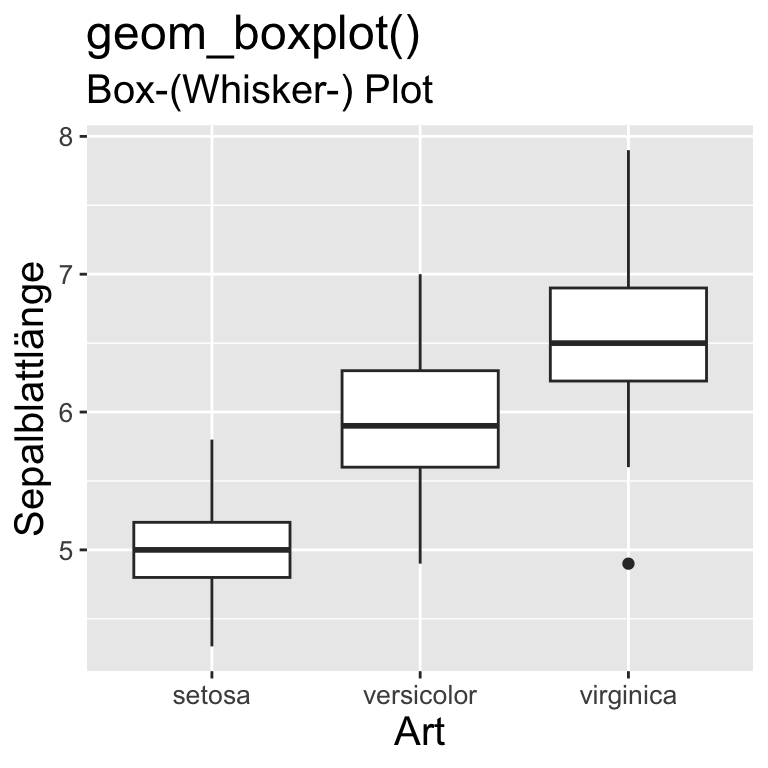
boxplot() |
Boxplot |
contour() |
Plot mit Höhenlinien |
coplot() |
sog. ‘conditional plot’ |
curve() |
Kurven über einem Intervall für eine Funktion |
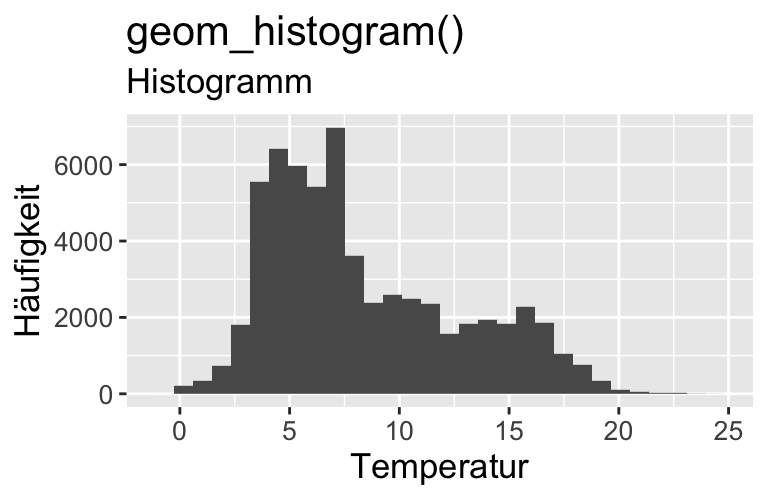
hist() |
Histogramm |
image() |
Erstellt kontextabhängige ‘Bildraster’ (auch in 3D) |
mosaicplot() |
Mosaikplot für kategorielle Daten |
pairs() |
Streudiagramm-Matrix für paarweise Gegenüberstellungen |
pie() |
Tortendiagramm |
qqplot() |
QQ-Plot |
High-level Funktionen | Beispiele
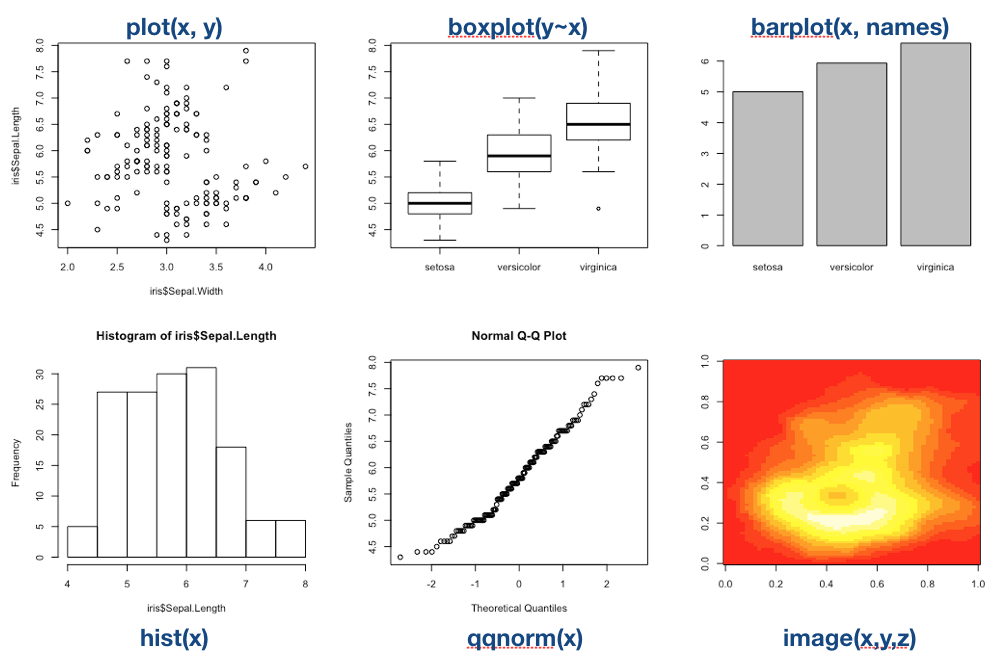
High-level Funktionen | Dokumentation
- Können über ihre Argumente modiziert und angepasst werden.
- In den Hilfen von
?plotund?plot.defaultwerden viele Argumente von Grafikfunktionen genannt und erklärt. - Es gibt jedoch auch globale Einstellungen für Grafiken, die alle nachfolgenden Grafiken verändern können:
par(). - Die Hilfe von
?parlistet wesentlich mehr Argumente, die auch in den high-level Funktionen genutzt werden können.
High-level Funktionen | Argumente
| Argument | Beschreibung |
|---|---|
| adj | Ausrichtung von Text (zentriert,…) |
| axes | Achsen sollen (nicht) eingezeichnet werden |
| bg | Hintergrundfarbe der Grafik |
| bty | Art der Box um die gezeichnete Grafik |
| cex | Größe der Schriftzeichen in der Grafik |
| col | Farben (der Linien, der Punkte, etc.) |
| las | Ausrichtung der Achsenbeschriftung |
| lty,lwd | Linientyp (gestrichelt,…) und Linienbreite |
| main,sub | Überschrift und Unterschrift |
| mfrow | mehrere Grafiken in einem Bild |
| pch | Darstellung eines Punktes |
| type | Typ der Darstellung (Linien, Punkte, Nichts) |
| xlab,ylab | x-/y-Achsenbeschriftung |
| xlim,ylim | Größe der Grafik in x-/y-Richtung |
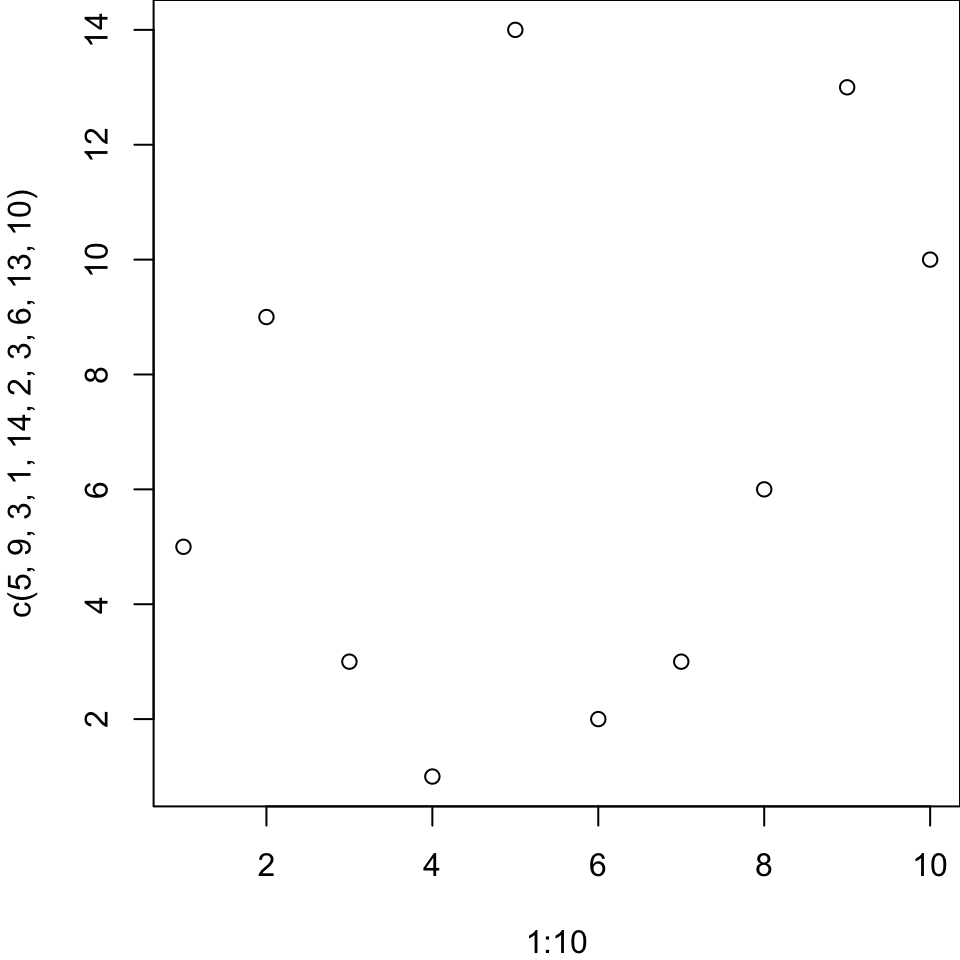
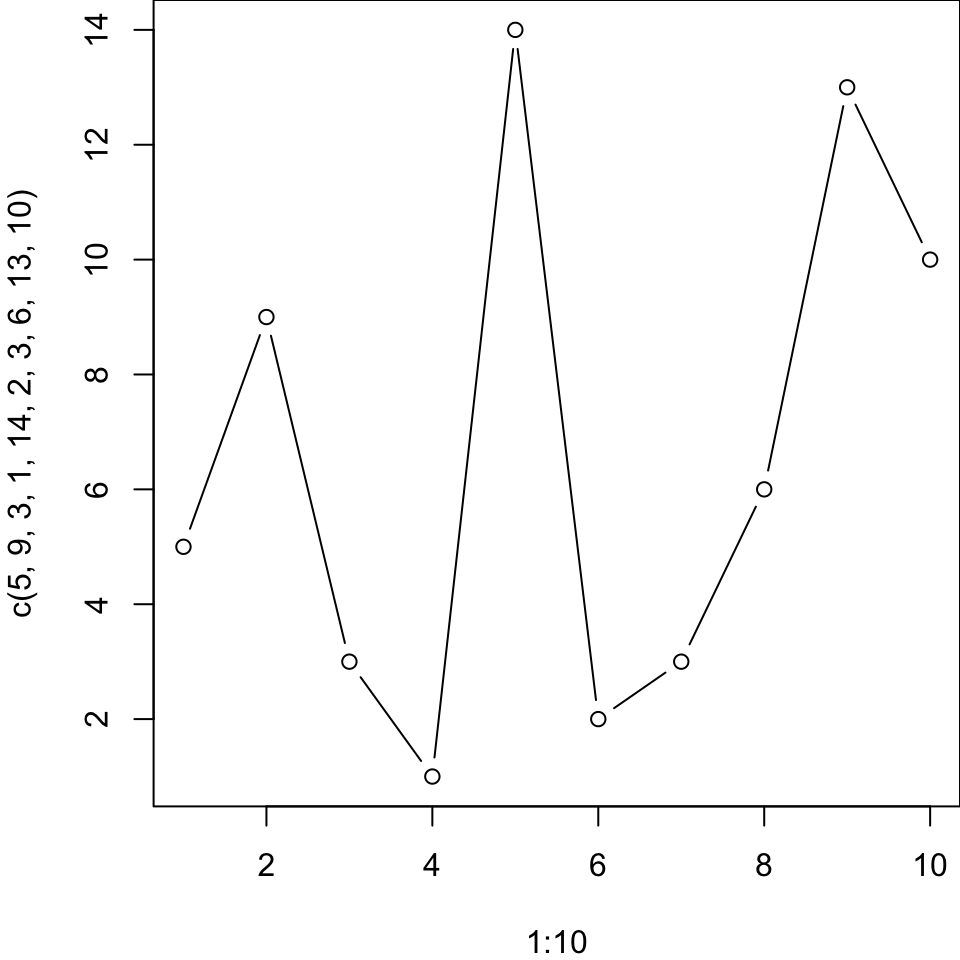
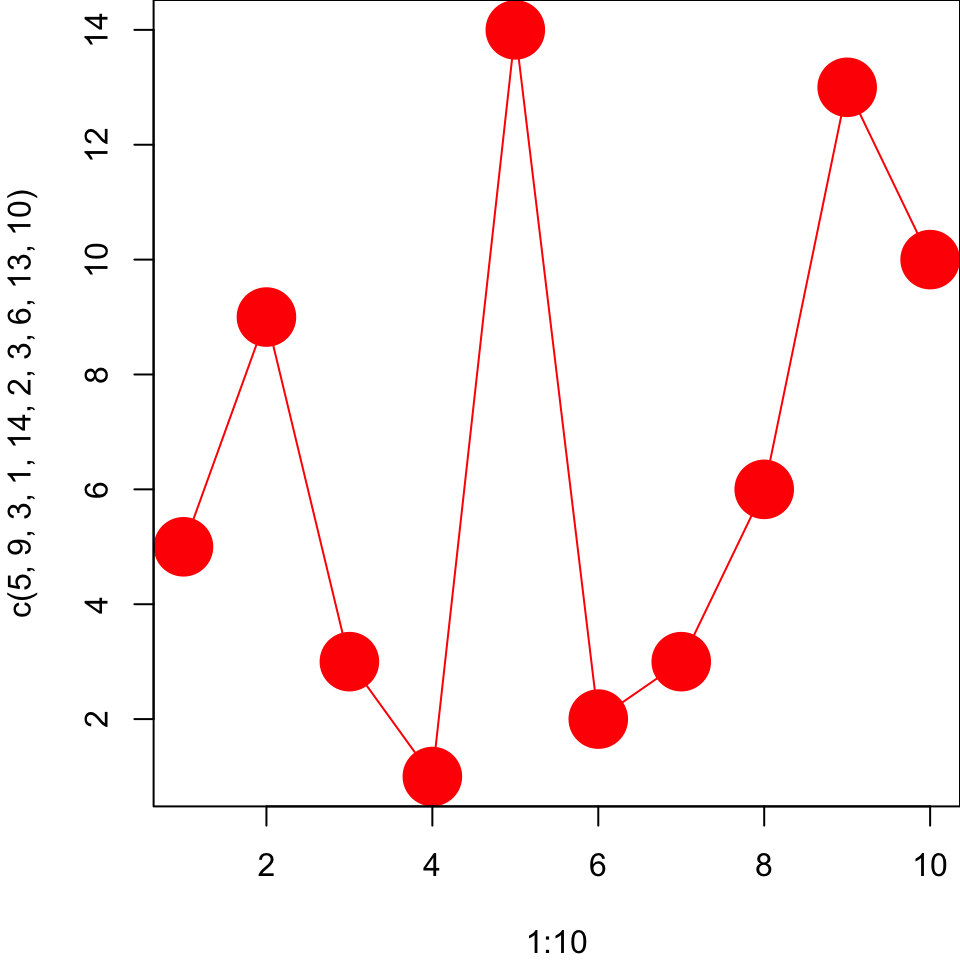
High-level Argumente | Demonstration 1
High-level Argumente | Demonstration 2
High-level Argumente | Demonstration 3
High-level Argumente | Demonstration 4
Wichtige low-level Grafikfunktionen
| Funktion | Beschreibung |
|---|---|
abline() |
Zeichnet eine kontextabhängige Linie |
arrows() |
Zeichnet Pfeile |
axis() |
Zeichnet Achsen (jede Achse einzeln!) |
grid() |
Zeichnet ein Gitternetz |
legend() |
Erstellt eine Legende im Plot |
lines() |
Zeichnet schrittweise Linien |
mtext() |
Schreibt Text in den Rand (s.unten) |
points() |
Zeichnet Punkte |
polygon() |
Zeichnet ausgefüllte Polygone |
rect() |
Zeichnet (vektorwertig) ein Rechteck |
segments() |
Zeichnet (vektorwertig) Linien |
text() |
Schreibt Text in den Plot |
title() |
Beschriftet den Plot |
Wo soll die Grafik erscheinen?
Die sog. ‘graphics devices’
![]()
Screen device (default) - Fenster im Programm
![]()
File device - Ausgabeformat als Rasterdatei (JPEG, PNG, TIFF,..) oder Vektordatei (PDF, EPS)
Ausgabeformate und ihre Funktionen
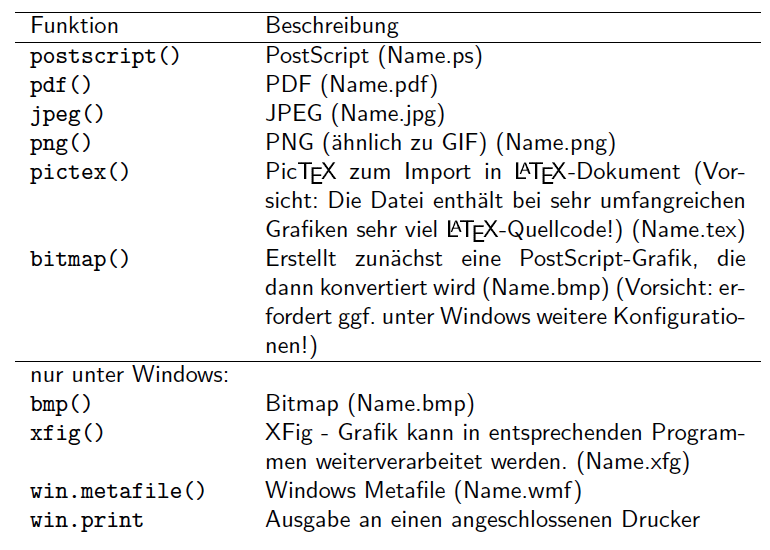
Ausgabeformate | Demonstration
Das Schließen mit dev.off() ist besonders wichtig, sonst werden alle folgenden Grafiken mit in dem PDF gespeichert und nicht in der Konsole angezeigt!
Visualisierung mit ‘ggplot2’
Ein System für kreative Grafiken

- Basierend auf ‘The Grammar of Graphics’.
- Das heutige Fundament für die Erstellung von Diagrammen in R nach dem visual encoding Prinzip
- Sehr gute Dokumentation.
- https://ggplot2.tidyverse.org
- Übersicht aller ggplot2 Funktionen gibt es auf https://ggplot2.tidyverse.org/reference/
- RStudio/Posit cheatsheet
- komplettes Buch (Onlineversion)
- Eine steigende Anzahl von Erweiterungen, die von ‘R Usern’ in der R Gemeinschaft entwickelt wurden: https://exts.ggplot2.tidyverse.org/


Gallerie der ggplot2 Erweiterungen

Weblink: https://exts.ggplot2.tidyverse.org
Umsetzung des visual encoding in ggplot2
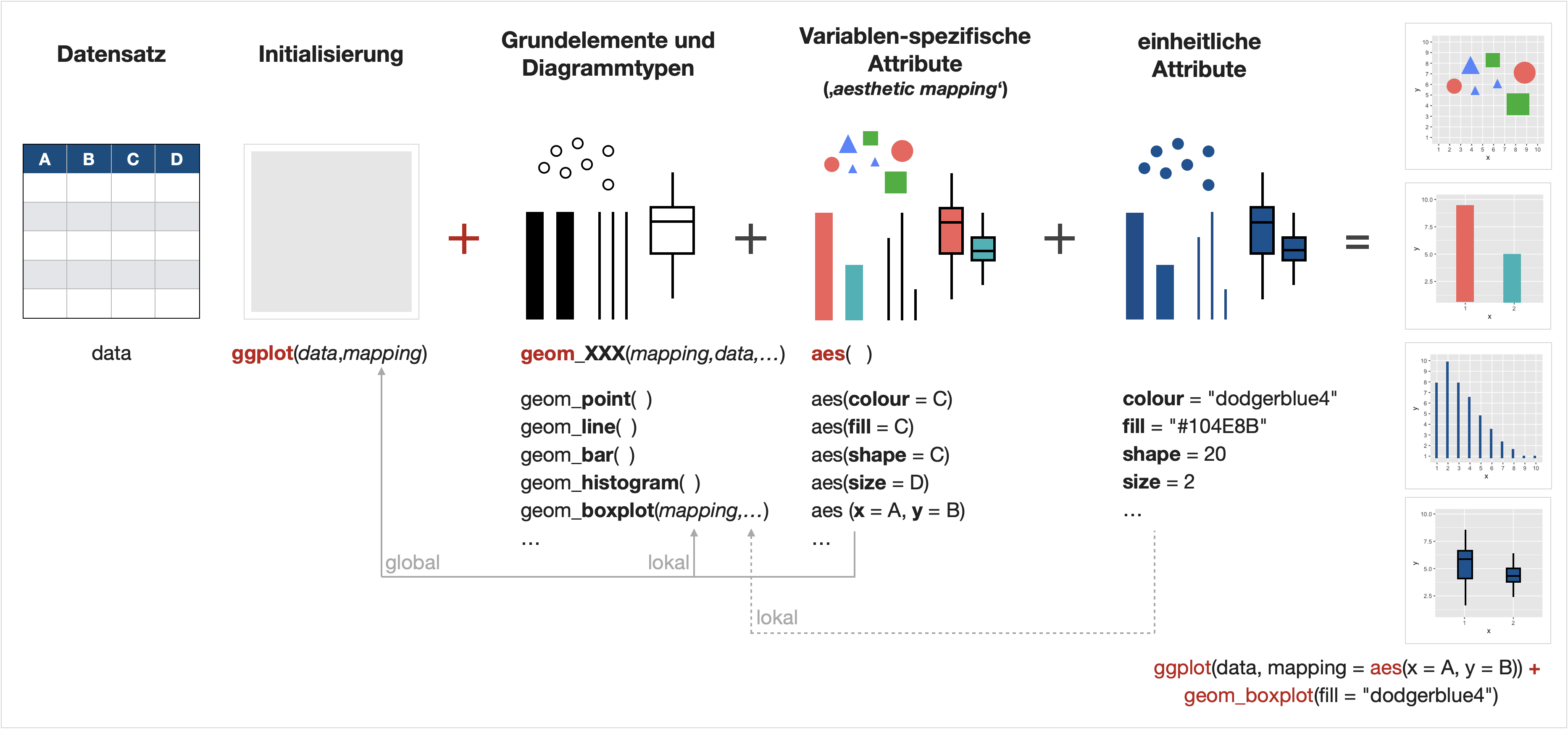
Die 8 Ebenen in ggplot2

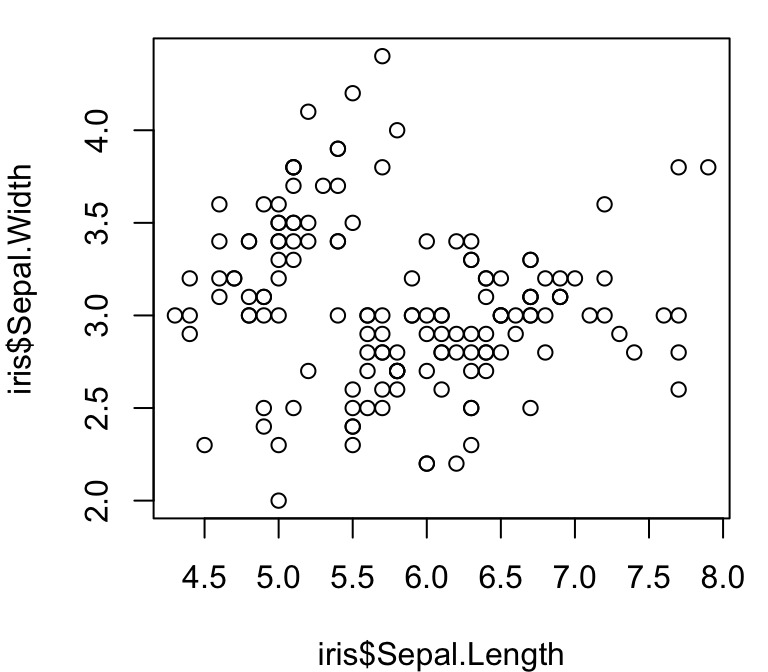
Eine Demonstration mit dem internen Datensatz ‘iris’
Fotos von Radomil Binek, Danielle Langlois, und Frank Mayfield (von links nach rechts); Zugriff über Wikipedia(unter CC-BY-SA 3.0 Lizenz).
Ebene 1 - Data
Grafik starten mit ggplot()
![]()
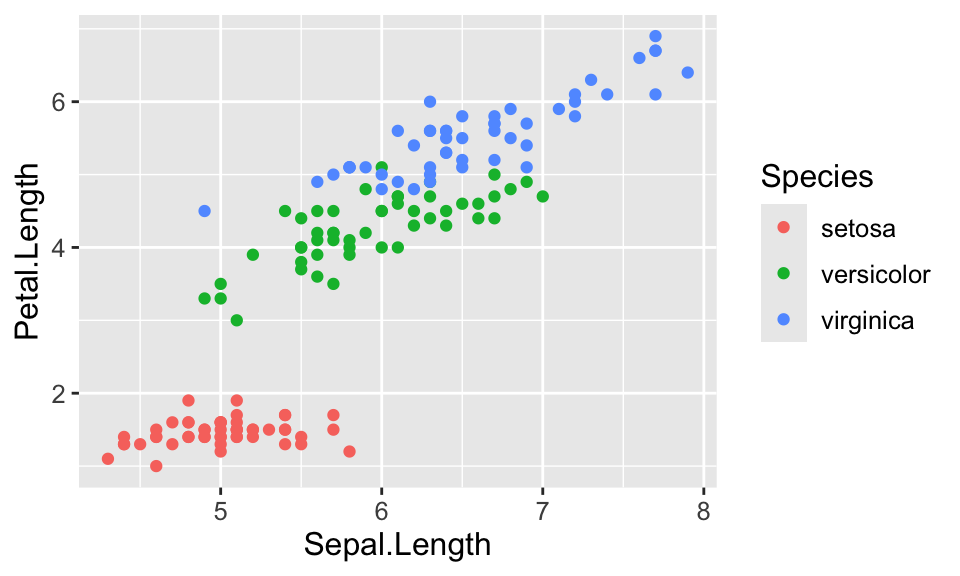
Ebene 2 - Aesthetic mapping | 1
Globale Variablenzuordnung von x und y
![]()
Ebene 3 - Geometries | 1
Punkte-Geometrie hinzufügen
![]()
Ebene 3 - Geometries | 2
Die Punktfarbe einheitlich anpassen
![]()
Ebene 2 - Aesthetic mapping | 2
Variablen-spezifische Farbwahl (lokal)
![]()
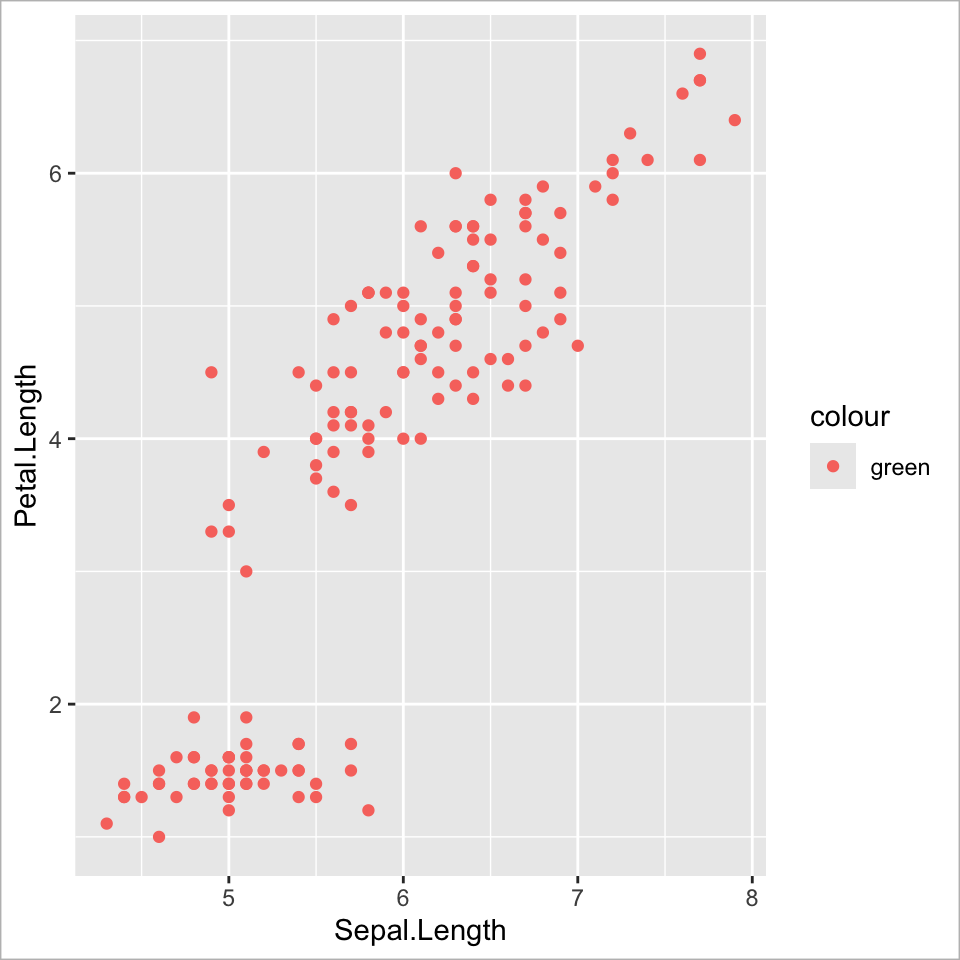
Ebene 2 - Aesthetic mapping | 3
Falsch: Einheitliche Farbwahl ‘mappen’
![]()
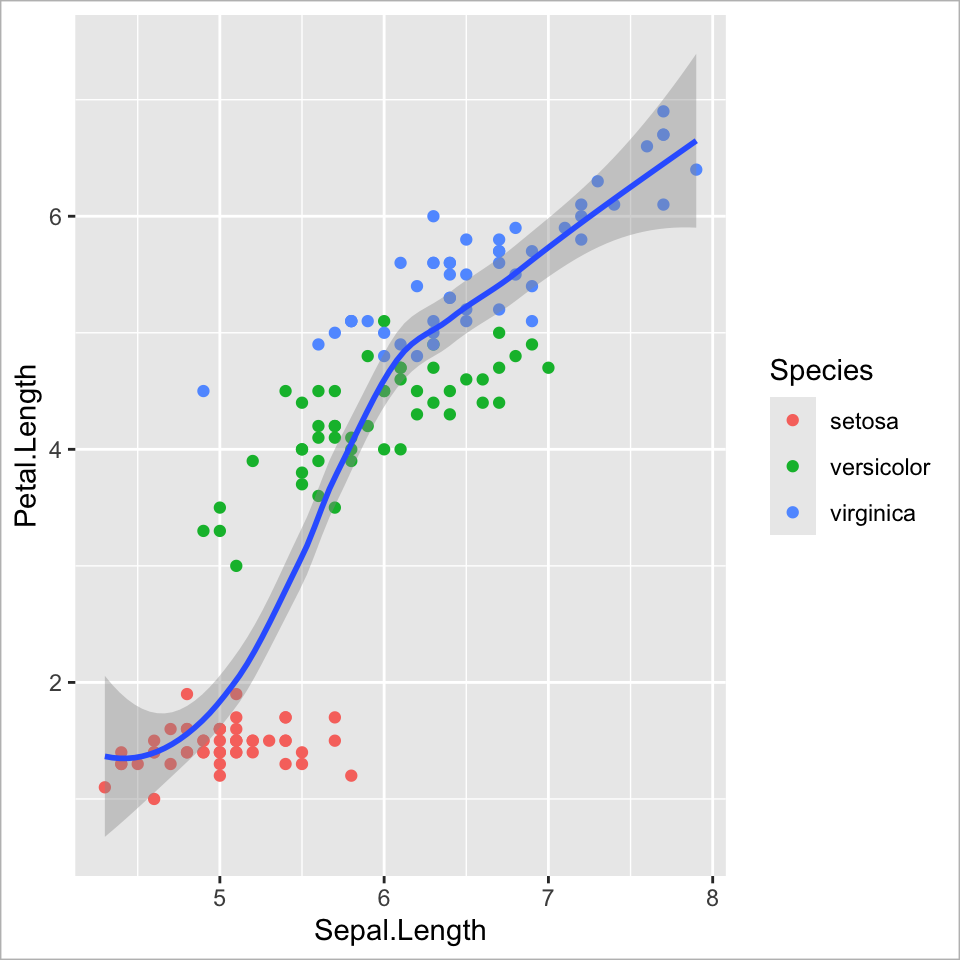
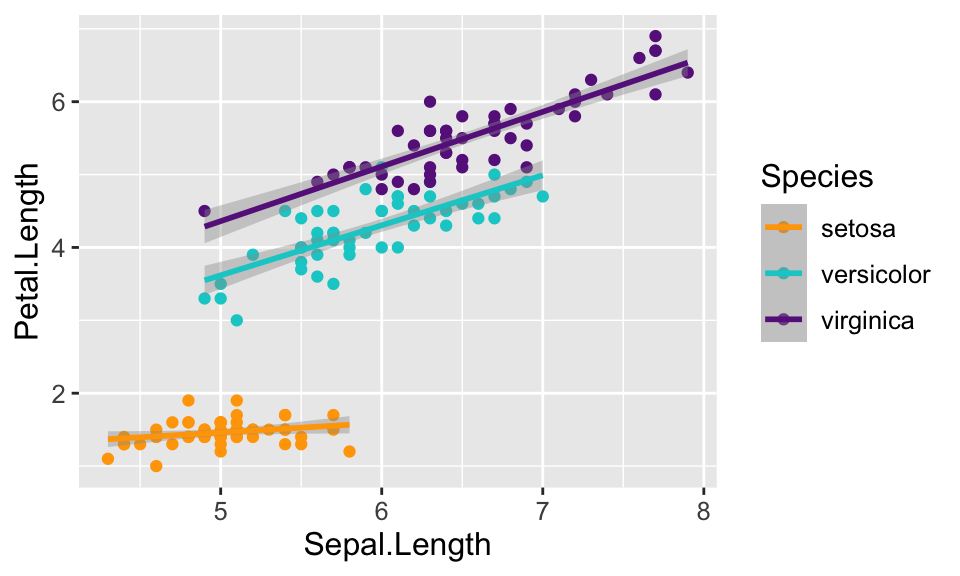
Ebene 3 - Geometries | 3
Weitere Geometrie hinzufügen: geglättete Kurve
![]()
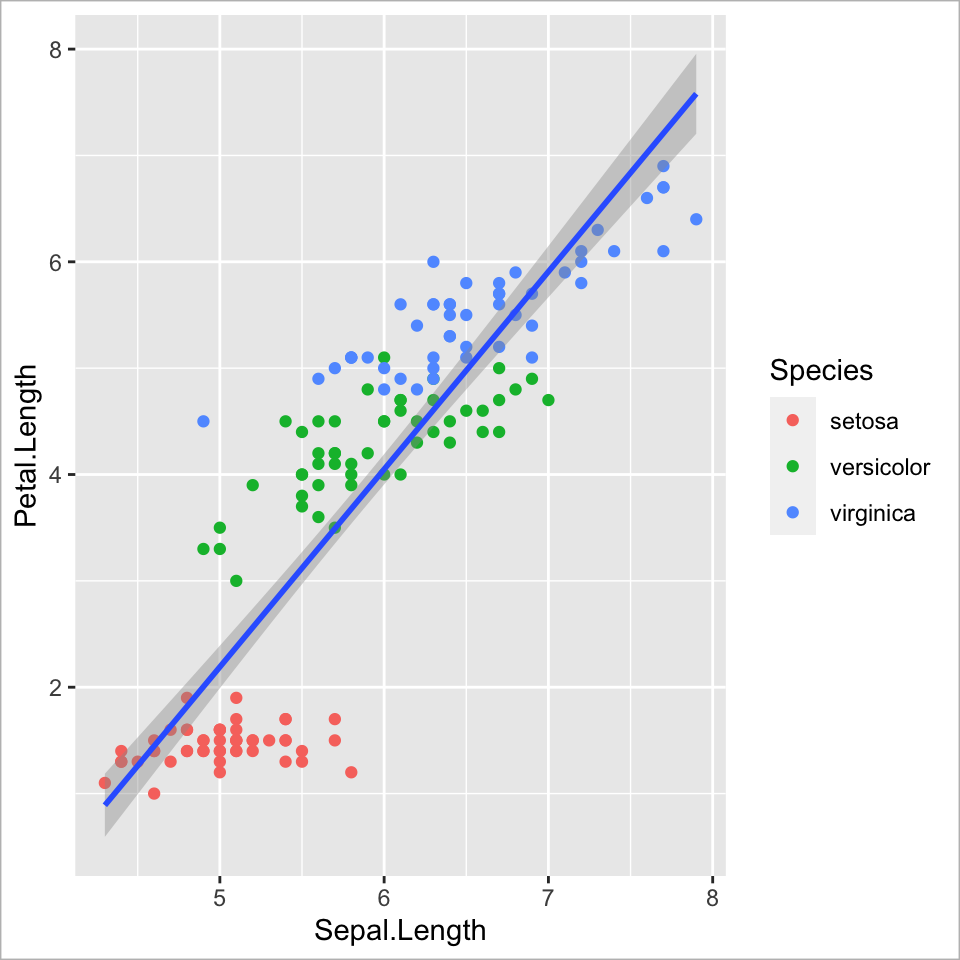
Ebene 3 - Geometries | 4
Weitere Geometrie hinzufügen: Trendlinie
![]()
Ebene 2 - Aesthetic mapping | 4
Art-spezifische Trendlinien über die Farbe (lokal)
![]()
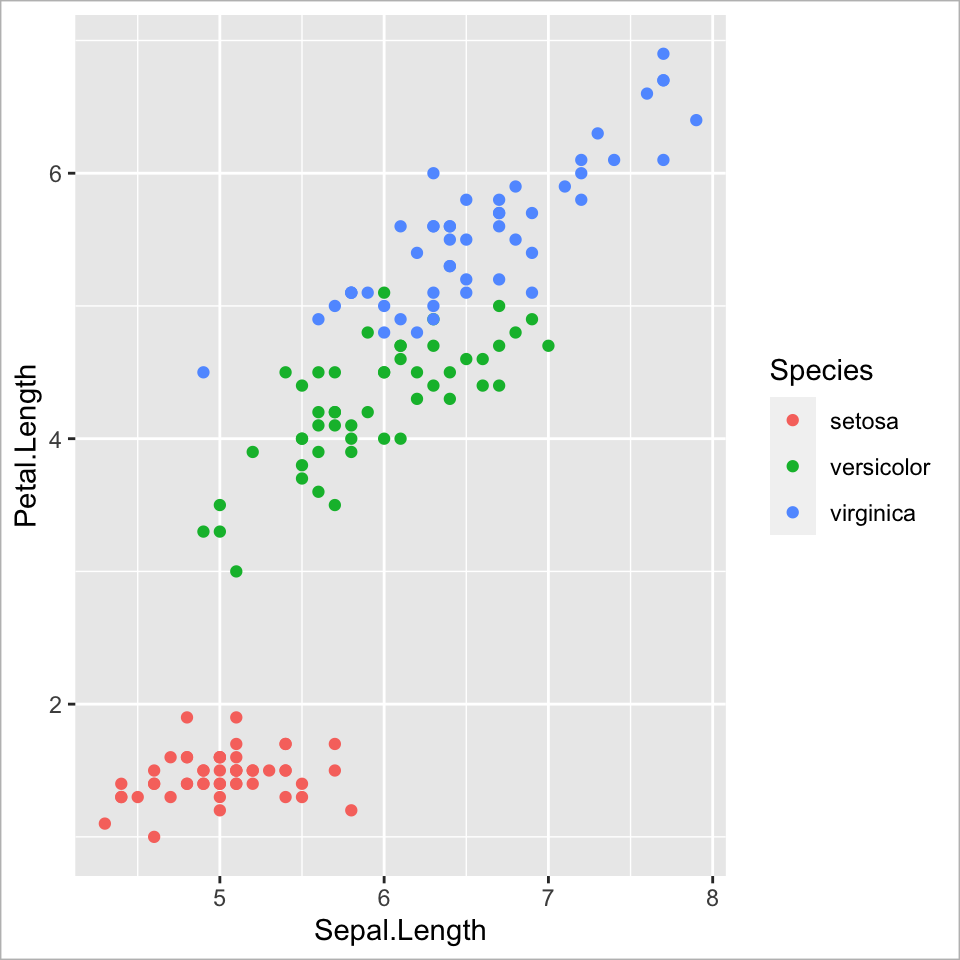
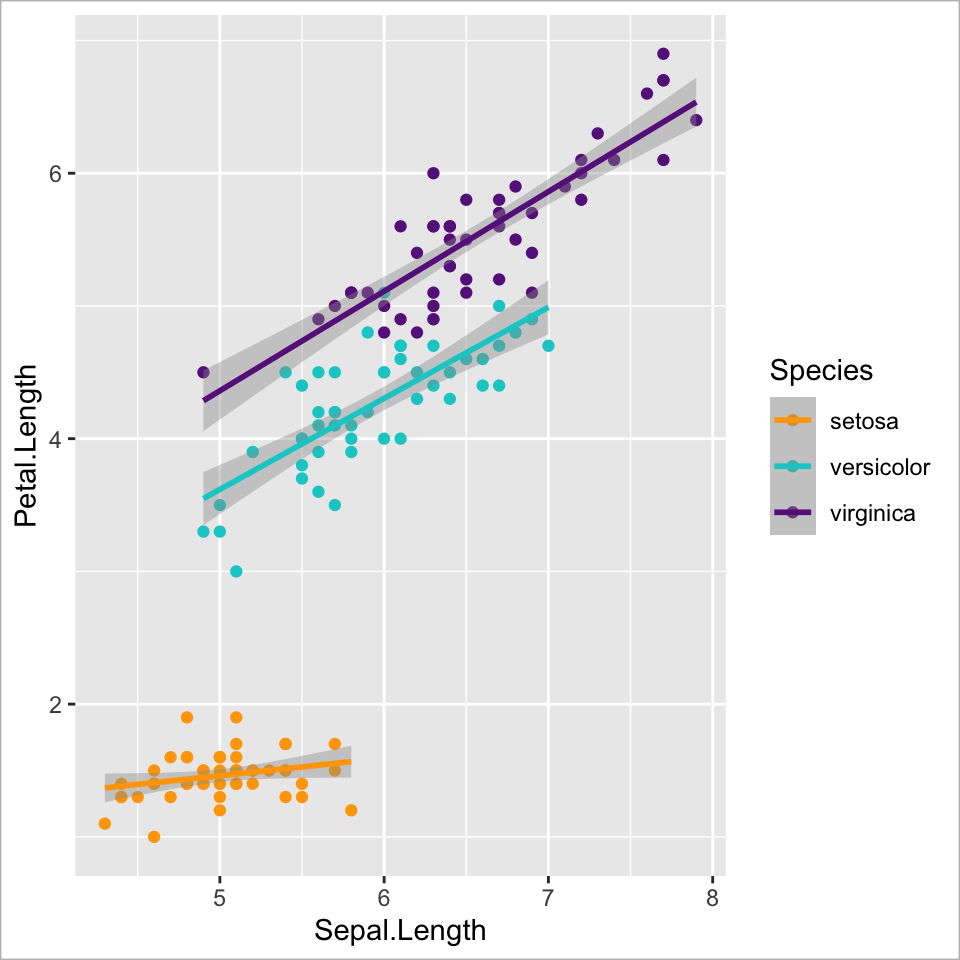
Ebene 2 - Aesthetic mapping
Globale Farbwahl (für alle Geometrien)
![]()
Ebene 4 - Scales
Farbskala anpassen
![]()
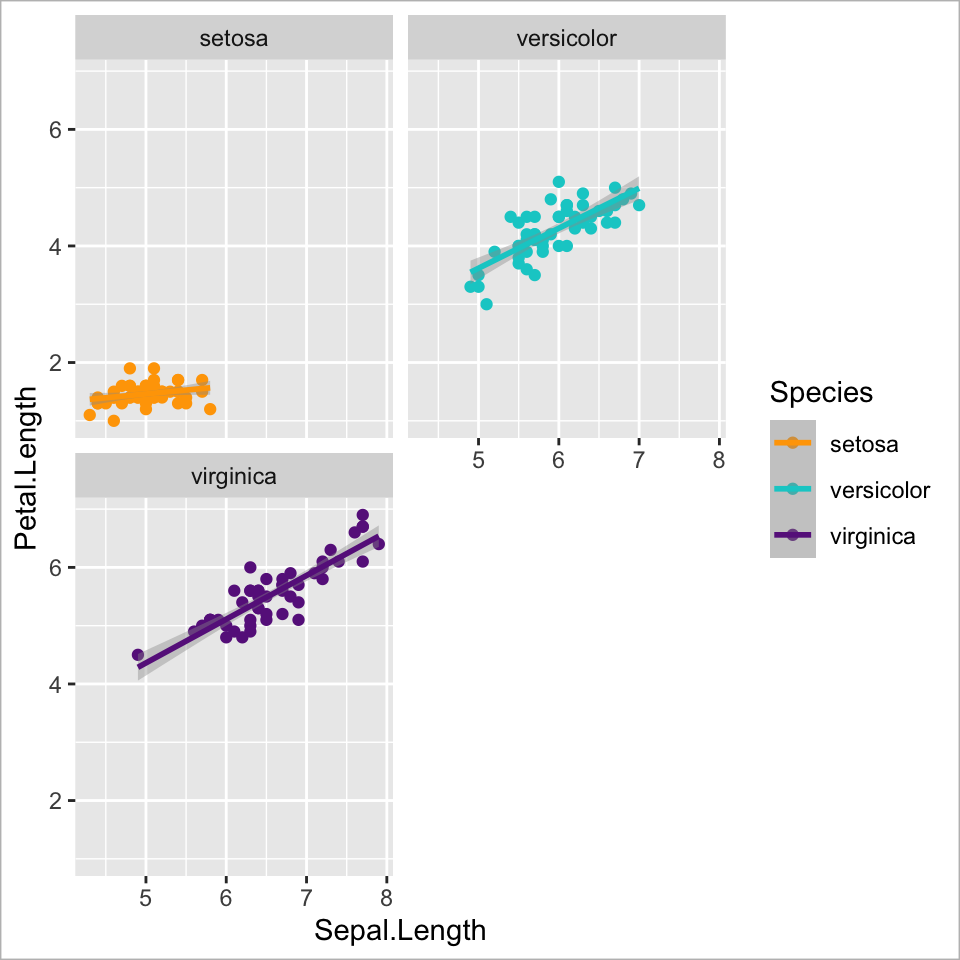
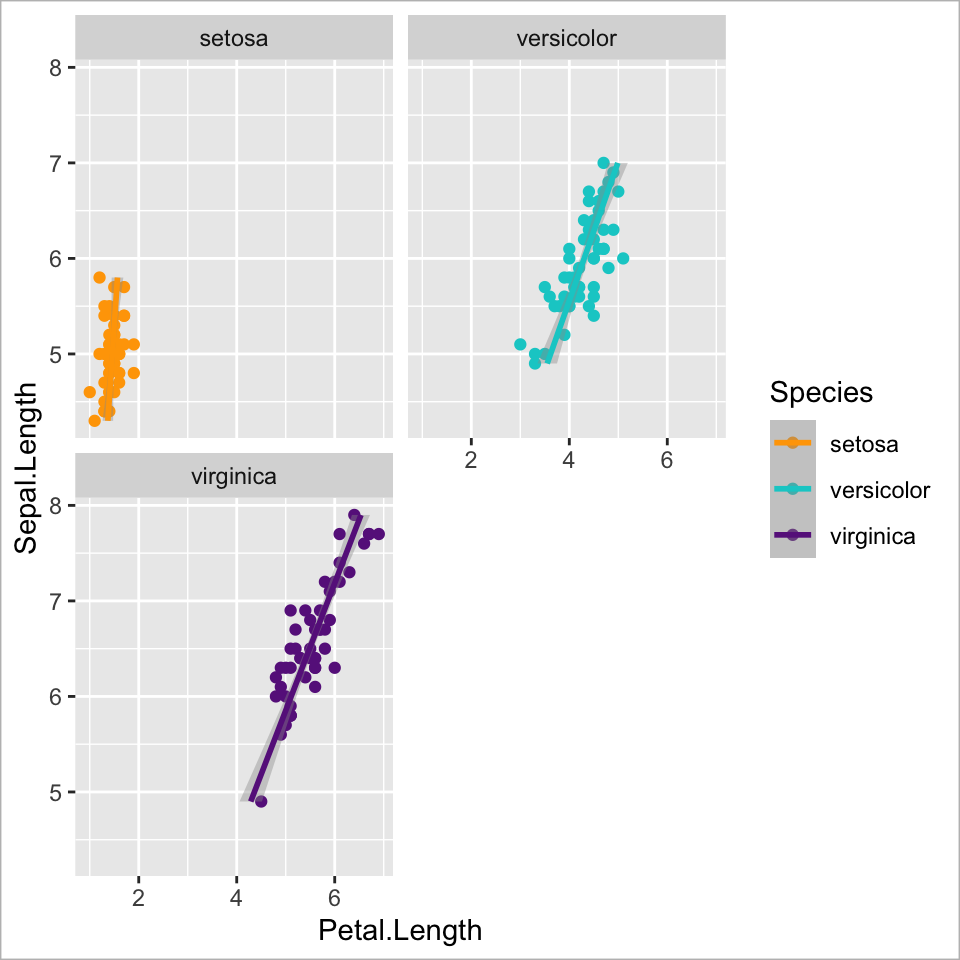
Ebene 5 - Facets
Datenpunkte aufteilen
![]()
Ebene 6 - Coordinates
Das Koordinatensystem bspw. drehen
![]()
Ebene 7 - Guides
Legende und Achsenbeschriftung anpassen
![]()
ggplot(
data = iris,
mapping = aes(
x = Sepal.Length,
y = Petal.Length,
colour = Species)
) +
geom_point() +
geom_smooth(method = "lm") +
scale_colour_manual(values =
c("orange","cyan3", "#68228B")) +
facet_wrap(~Species, nrow = 2) +
coord_flip() +
guides(colour = guide_none()) +
labs(x = "Länge Kelchblatt (cm)",
y = "Länge Kronblatt (cm)")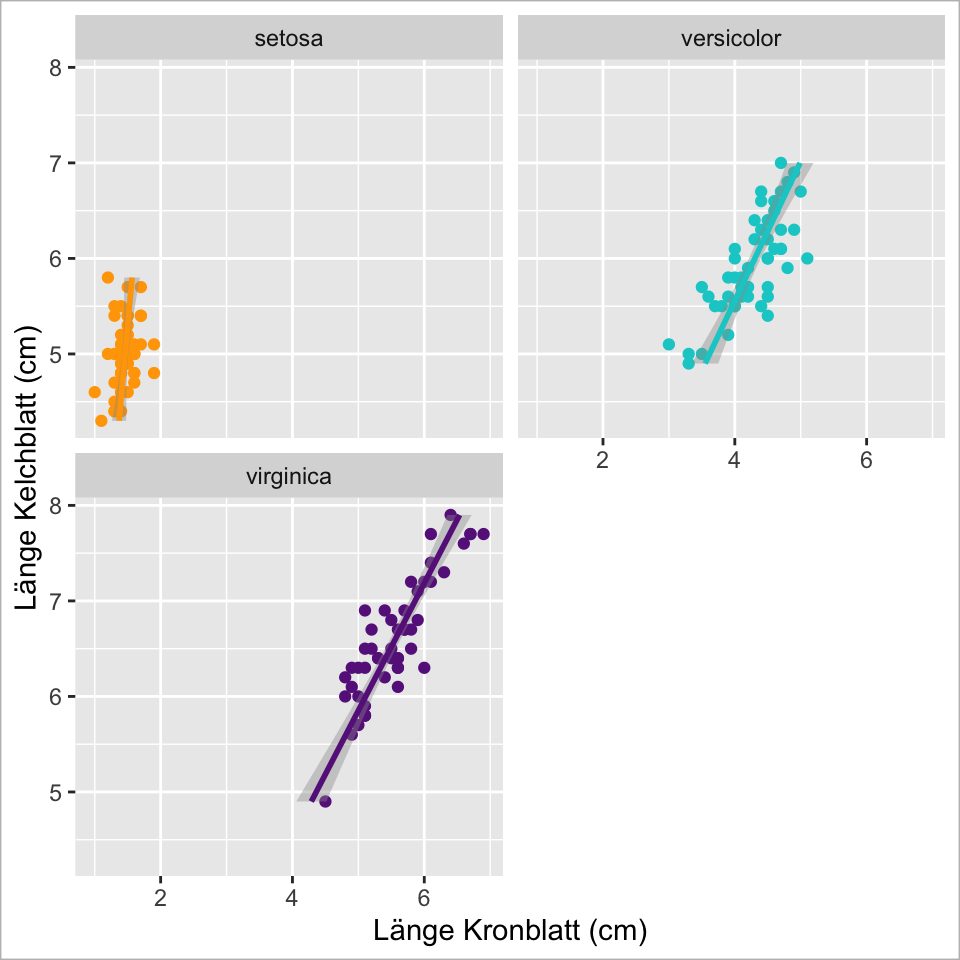
Ebene 8 - Theme
Ändern des Layouts (der Nicht-Datenelemente)
![]()
ggplot(
data = iris,
mapping = aes(
x = Sepal.Length,
y = Petal.Length,
colour = Species)
) +
geom_point() +
geom_smooth(method = "lm") +
scale_colour_manual(values =
c("orange","cyan3", "#68228B")) +
facet_wrap(~Species, nrow = 2) +
coord_flip() +
guides(colour = guide_none()) +
labs(x = "Länge Kelchblatt (cm)",
y = "Länge Kronblatt (cm)") +
theme_dark()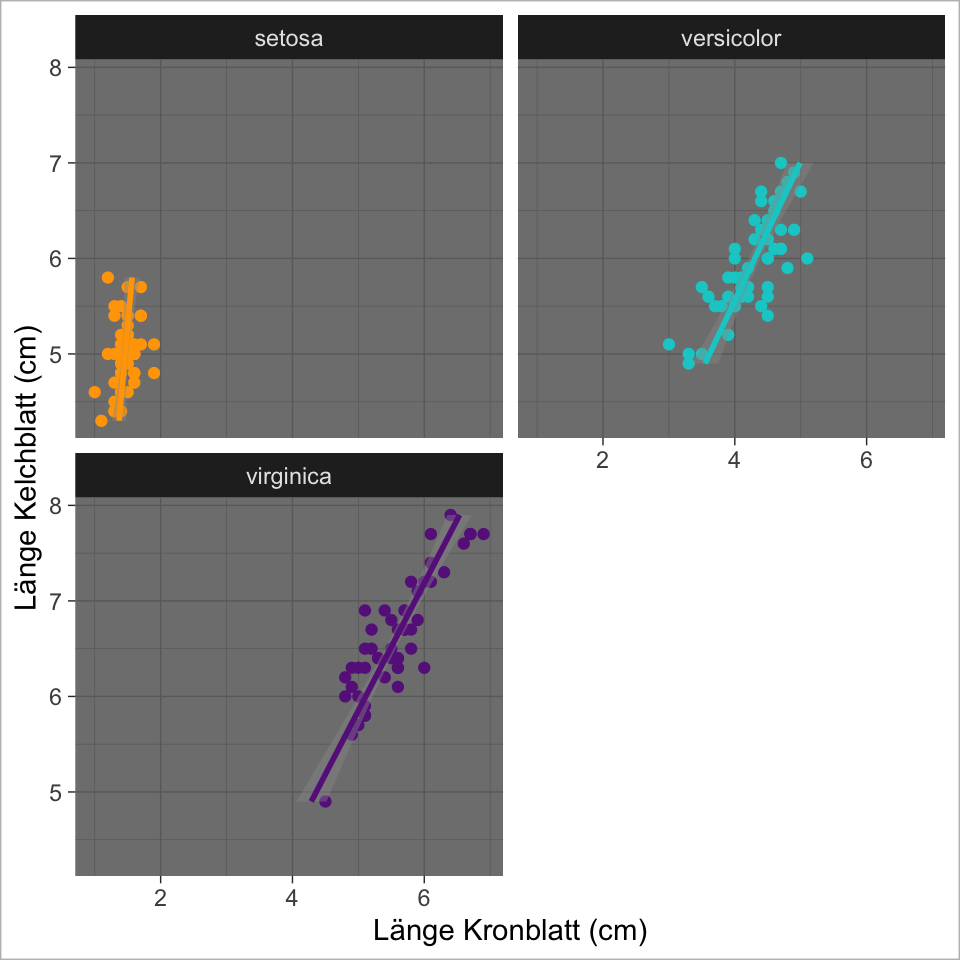
Zum Abschluss: speichere die Grafik
Mit ggsave()
Das letzte angezeigte Diagramm (im ‘screen device’) wird als PDF in 4x4 inch gespeichert (auch Standardeinstellung).
Zusammenfassung der einzelnen Ebenen
![]()
1. Daten
Starte mit dieser Funktion und übergebe einen Datensatz an die Funktion: ggplot(data)
2. Aesthetic mapping
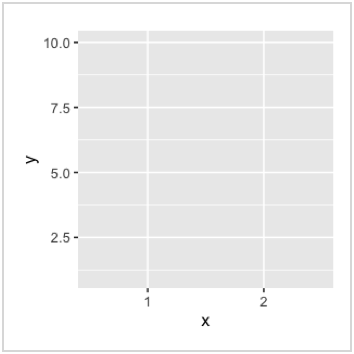
Als nächstes müssen die Datenvariablen den Koordinaten zugeordnet werden damit ein leeres Koordinatensystem dargestellt wird. Dies geschieht in der aes() Funktion. Weitere visuelle Eigenschaften (sog. aesthetics) sind z.B. Größe, Farbe, Form, etc.
- Zuordnung alleinstehend oder innerhalb von
ggplot()gilt für alle Ebenen. - Zuordnung in
geom_XXX()Funktion gilt nur für diese Ebene.
3. Geometrien
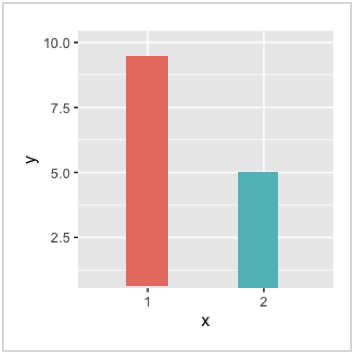
geom_XXX(): Kombiniere ein geometrisches Objekt/Grundelement, welches die Daten repräsentiert, mit den Abbildungseigenschaften, einer Statistik und einer Positionsanpassung, z.B. geom_point() oder geom_col().
4. ‘Scales’
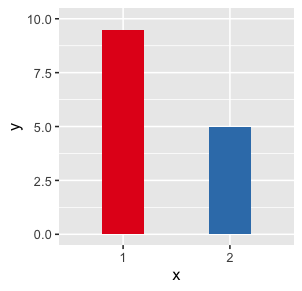 Sog. ‘scale’ Funktionen bestimmen wie Datenwerte in visuelle Eigenschaften übersetzt werden (überschreiben die Standardeinstellungen), wie z.B.
Sog. ‘scale’ Funktionen bestimmen wie Datenwerte in visuelle Eigenschaften übersetzt werden (überschreiben die Standardeinstellungen), wie z.B.scale_fill_manual().
5. ‘Faceting’
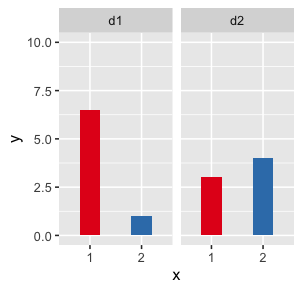
facet_XXX() Funktionen erstellen kleinere Diagramme, die verschiedene Teilmengen der Daten anzeigen; nützlich zur Untersuchung von Interaktionen mit kategorialen Variablen.
6. Koordinatensystem
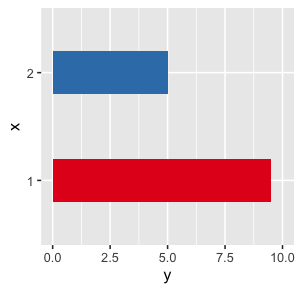 Funktionen wie
Funktionen wie coord_flip() bestimmen die Darstellung des XY-Koordinatensystems und somit die Positionierung der Daten.
7. ‘Guides’
Mit der guides() Funktion und weiteren guide_XXX() Helferfunktionen kann die Anzeige der Achsen und Legende bestimmt werden.
8. ‘Themes’
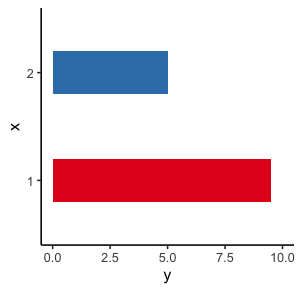 Sog. ‘theme’ Funktionen bestimmen die Anzeige aller Nicht-Datenelemente des Plots. Es können alle Einstellungen mit einem kompletten Thema wie
Sog. ‘theme’ Funktionen bestimmen die Anzeige aller Nicht-Datenelemente des Plots. Es können alle Einstellungen mit einem kompletten Thema wie theme_classic() überschrieben oder einzelne Einstellungen mit theme() verändert werden.
Grafik speichern
Das aktuell angezeigte ggplot Diagramm lässt sich mit ggsave() speichern: ggsave("plot.png", width = 5, height = 5) → speichert den Plot als PDF in 5x5 inch ab.
ggplots als Objekte abspeichern | 1
- Nützlich, um weitere Ebenen später hinzuzufügen,
- wenn der Basisplot Grundlage für mehrere Diagramme sein soll (reduziert Tipparbeit)
- oder wenn mehrere Diagramme kombiniert werden sollen.
ggplots als Objekte abspeichern | 2
- Nützlich, um weitere Ebenen später hinzuzufügen,
- wenn der Basisplot Grundlage für mehrere Diagramme sein soll (reduziert Tipparbeit)
- oder wenn mehrere Diagramme kombiniert werden sollen.
Zurück zu unseren Demos..

Von Calc zu R wechseln | Demo A
Lineare Regression zum
Fütterungsversuch beim Kabeljau
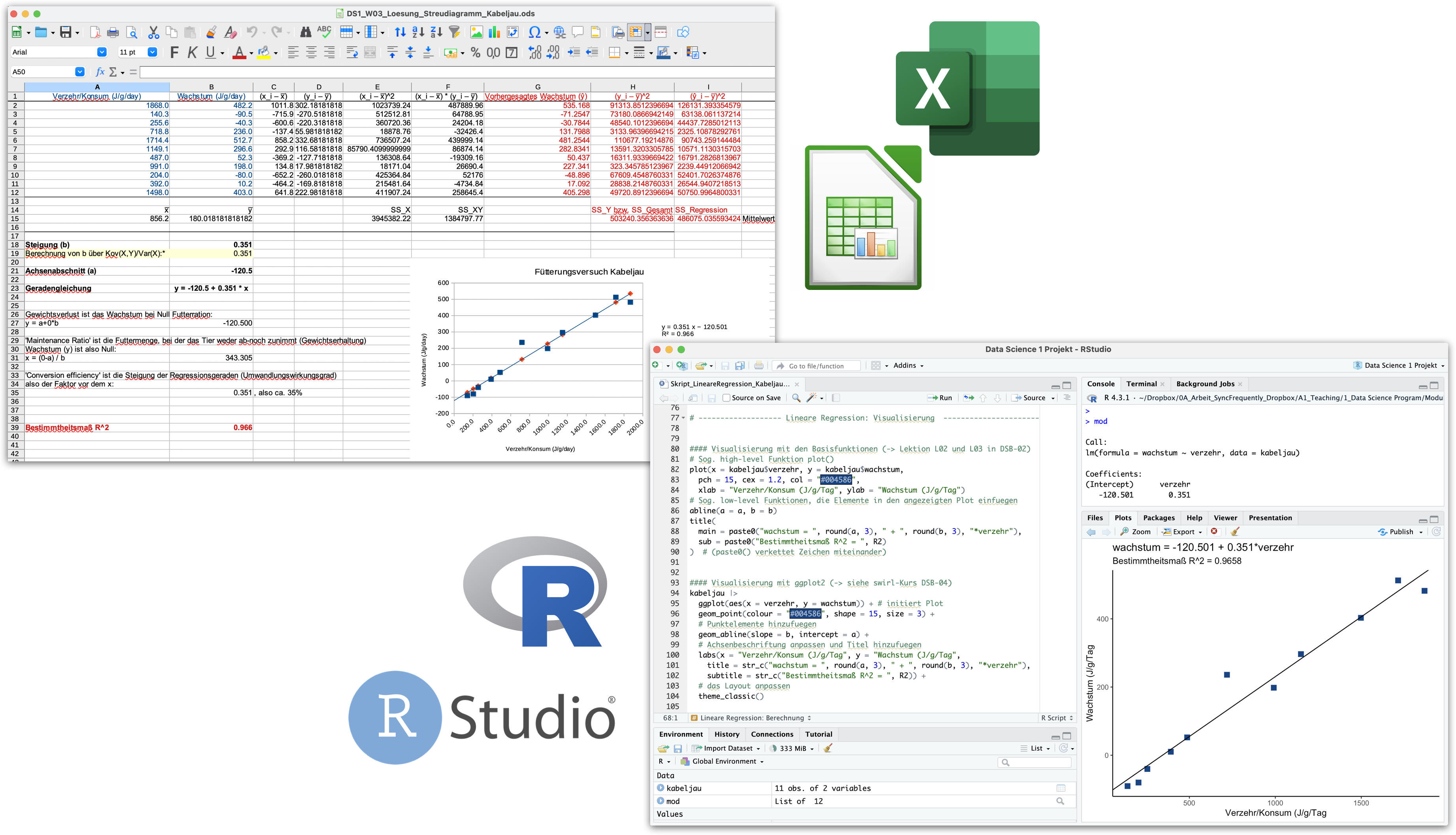
Was haben wir gerade gelernt?
# ------------------------------ Vorbereitung ----------------------------------
#### Laden von Paketen
library(tidyverse)
# library(readODS)
# -------------------- Import und Datenaufbereitung ----------------------------
#### Import einer Textdatei im CSV-Format (das gaengigste Format)
kabeljau_csv <- read.csv(file = "Wachstum-Futter.csv")
str(kabeljau_csv) # --> output ist ein data frame
#### Import von Calc- und Excel-Dateien (ODS- und XLSX-Format)
# Import einer ODS-Datei mit dem 'readODS' Paket
kabeljau <- readODS::read_ods("DS1_W03_Streudiagramm_Kabeljau.ods",
sheet = "Daten_Visualisierung")
str(kabeljau) # --> output ist ein tibble
# Import einer XLSX-Datei mit z.B. dem 'readxl' Paket
kabeljau_xlsx <- readxl::read_excel("DS1_W03_Streudiagramm_Kabeljau.xlsx",
sheet = "Daten_Visualisierung")
str(kabeljau_xlsx) # --> output ist ein tibble
#### Datensichtung und -transformation
# Anpassen der Spaltennamen
names(kabeljau) <- c("verzehr", "wachstum")
str(kabeljau)
# Wertebereich pruefen
summary(kabeljau)
# ---------------------- Lineare Regression: Berechnung ------------------------
# (-> Swirl-Lektion L03 in DSB-02-Datenexploration mit R)
#### Manuelle Berechnung
# Um Tipparbeit zu sparen, speichern wir die Spalten als einzelne Vektoren
x <- kabeljau$verzehr
y <- kabeljau$wachstum
# Steigungsparameter b berechnen
b <- cov(x = x, y = y)/ var(x) # der shortcut mit der Kovarianz und Varianz
b
# Achsenabschnitt a berechnen
a <- mean(y) - b*mean(x)
a
# Das Bestimmtheitsmass R^2 berechnen
y_obs <- a + b*x # die vorhergesagten Werte
ss_gesamt <- sum( (y - mean(y))^2 ) # Summenquadrate Gesamt
ss_regression <- sum( (y_obs - mean(y))^2 ) # Summenquadrate der Regression
R2 <- round(ss_regression/ss_gesamt, 4)
R2
##### Zum Vergleich die Regression automatisch berechnen mit lm()
# Erstellung des Modells
mod <- lm(formula = wachstum ~ verzehr, data = kabeljau)
mod
# Ausgabe nur der beiden Koeffizienten
coef(mod)
# Ausgabe aller wichtigen Statistiken des Modells, inklusive von R^2
# (mehr dazu in Data Science 2)
summary(mod)
# ------------------- Lineare Regression: Visualisierung ----------------------
#### Visualisierung mit den Basisfunktionen (-> Lektion L02 und L03 in DSB-02)
# Sog. high-level Funktion plot()
plot(x = kabeljau$verzehr, y = kabeljau$wachstum,
pch = 15, cex = 1.2, col = "#004586",
xlab = "Verzehr/Konsum (J/g/Tag", ylab = "Wachstum (J/g/Tag")
# Sog. low-level Funktionen, die Elemente in den angezeigten Plot einfuegen
abline(a = a, b = b)
title(
main = paste0("wachstum = ", round(a, 3), " + ", round(b, 3), "*verzehr"),
sub = paste0("Bestimmtheitsmaß R^2 = ", R2)
) # (paste0() verkettet Zeichen miteinander)
#### Visualisierung mit ggplot2 (-> siehe swirl-Kurs DSB-04)
kabeljau |>
ggplot(aes(x = verzehr, y = wachstum)) + # initiert Plot
geom_point(colour = "#004586", shape = 15, size = 3) +
# Punktelemente hinzufuegen
geom_abline(slope = b, intercept = a) +
# Achsenbeschriftung anpassen und Titel hinzufuegen
labs(x = "Verzehr/Konsum (J/g/Tag", y = "Wachstum (J/g/Tag",
title = str_c("wachstum = ", round(a, 3), " + ", round(b, 3), "*verzehr"),
subtitle = str_c("Bestimmtheitsmaß R^2 = ", R2)) +
# das Layout anpassen
theme_bw()DS1_VL04a_Demo_LineareRegression_Kabeljau.R
Von Calc zu R wechseln | Demo B
Deskriptive Statistik mit iris
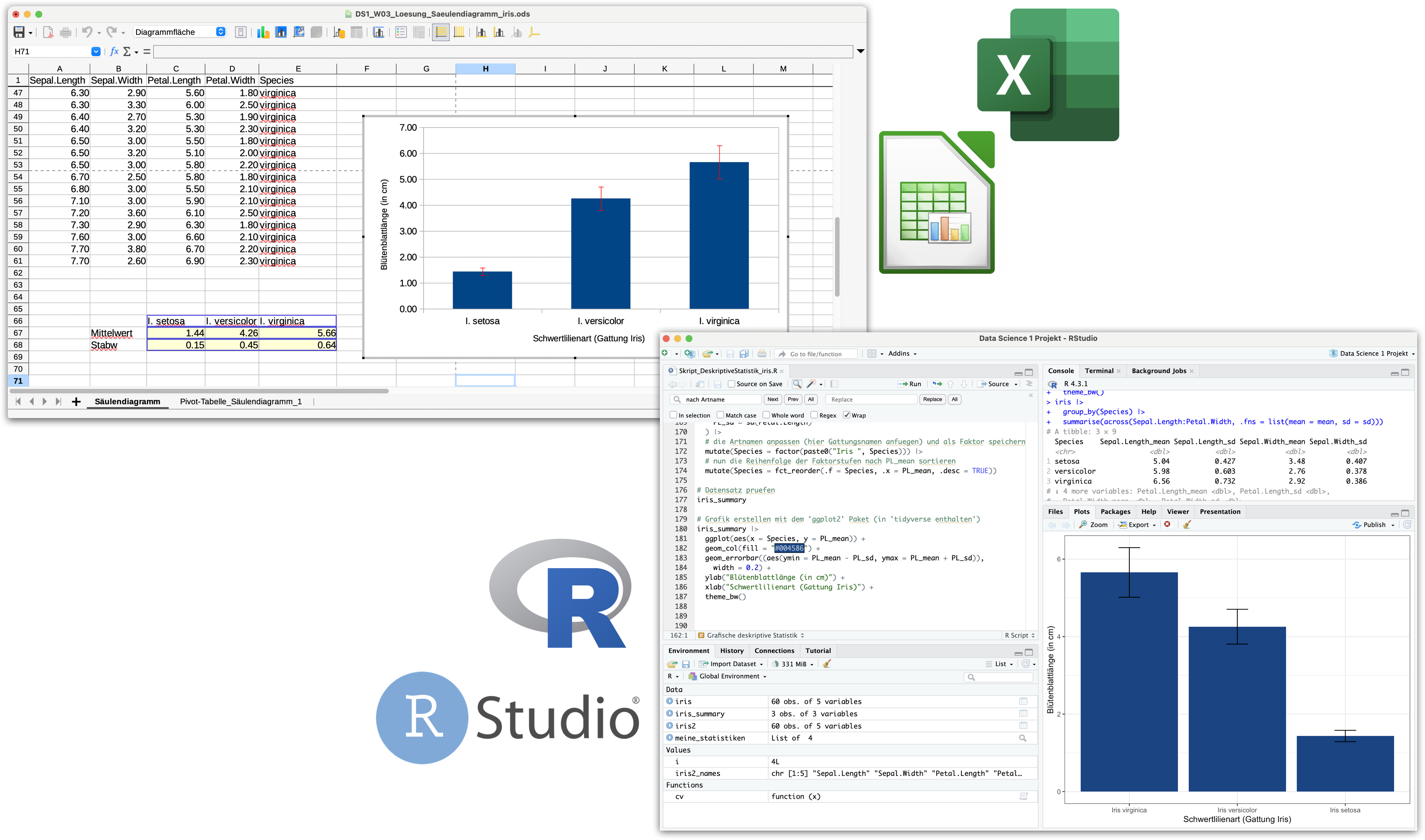
Was haben wir gerade gelernt?
# ------------------------------ Vorbereitung ----------------------------------
#### Laden von Paketen
library(tidyverse) # laedt 9 Pakete
# # (das gleiche wie alle Pakete einzeln zu laden)
# library(dplyr)
# library(forcats)
# library(ggplot2)
# library(lubridate)
# library(purrr)
# library(readr)
# library(stringr)
# library(tibble)
# library(tidyr)
#### Eigene Funktionen
# Variantionskoeffizient
cv <- function(x) {
sd(x)/mean(x)
}
# -------------------- Import und Datenaufbereitung ----------------------------
#### Import von CSV-Dateien
# (-> Swirl-Lektion L01 in DSB-03-Datenaufbereitung oder per Anleitung durchs Tidyversum)
# Import der ODS-Datei, welche in den Zeilen 66-68 noch Text enthaelt
iris <- readODS::read_ods("DS1_W03_Saeulendiagramm_mit_iris.ods")
#### Pruefung des Imports und Datensichtung
# (-> Swirl-Lektion L02 in DSB-03)
# Pruefung des Datentyps --> IMMER DIREKT NACH DEM IMPORT VERWENDEN!
str(iris) # str = structure
# Betrachtung des Inhalts
iris
# View(iris)
# Korrektur des Datentyps und der Zeilen
iris <- iris[1:60, ] |>
mutate(across(Sepal.Length:Petal.Width, as.numeric)) |>
mutate(Species = as.factor(Species))
str(iris)
# Welche Werte kommen in jeder Spalte vor?
lapply(iris, unique)
### Weitere Funktionen zur Sichtung einzelner Aspekte
head(iris) # zeigt erste 6 Zeilen (Kopfzeilen)
tail(iris) # zeigt letzte 6 Zeilen (Endzeilen)
class(iris) # Identifikation der Objektklasse (Vektor, Matrix, dataframe,..)
nrow(iris) # Anzahl Zeilen
ncol(iris) # Anzahl Spalten
dim(iris) # Anzahl aller Dimensionen
names(iris) # Spaltennamen
typeof(iris$Sepal.Length) # Datentyp von Spalte 'Sepal.Length'
typeof(iris$Species) # Datentyp von Spalte 'Species'
# ----------------------- Deskriptive Statistik --------------------------------
#### Berechnung mehrerer Statistiken für jede Spalte im data frame
summary(iris)
#### Berechnung versch. Statistiken der Kronblattlaenge, gruppiert nach Art
# (-> Lektion L01 in DSB-02-Datenexploration mit R)
# (-> Lektion L06-Gruppierte Aggregation in DSB-03)
iris_summary <- iris |>
group_by(Species) |>
summarise(
PL_mean = mean(Petal.Length), # Mittelwert
PL_median = median(Petal.Length), # Median
PL_var = var(Petal.Length), # Varianz
PL_sd = sd(Petal.Length), # Standardabweichung
PL_se = sd(Petal.Length)/sqrt(length(Petal.Length)), # Standardfehler
PL_cv = cv(Petal.Length) # Variationskoeffizient
) |>
# die Artnamen anpassen (hier Gattungsnamen anfuegen) und als Faktor speichern
mutate(Species = factor(paste0("Iris ", Species))) |>
# nun die Reihenfolge der Faktorstufen nach PL_mean sortieren
mutate(Species = fct_reorder(.f = Species, .x = PL_mean, .desc = TRUE))
# Zusammenfassung ansehen
iris_summary
#### Saeulendiagramm erstellen mit dem 'ggplot2' Paket
# (siehe auch swirl-Kurs DSB-04-Datenvisualisierung mit ggplot2)
iris_summary |>
ggplot(aes(x = Species, y = PL_mean)) + # initiert Plot
# die Saeulen hinzufuegen
geom_col(fill = "#004586") +
# die Fehlerbalken hinzufuegen
geom_errorbar((aes(ymin = PL_mean - PL_sd, ymax = PL_mean + PL_sd)),
width = 0.2) +
# Achsenbeschriftung anpassen
ylab("Kronblattlänge (in cm)") +
xlab("Schwertlilienart (Gattung Iris)") +
# das Layout anpassen
theme_bw()DS1_VL04b_Demo_DeskriptiveStatistik_iris.R
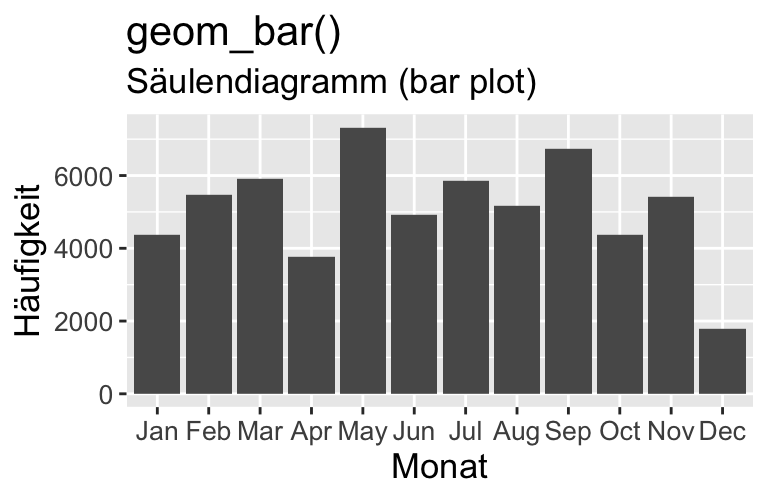
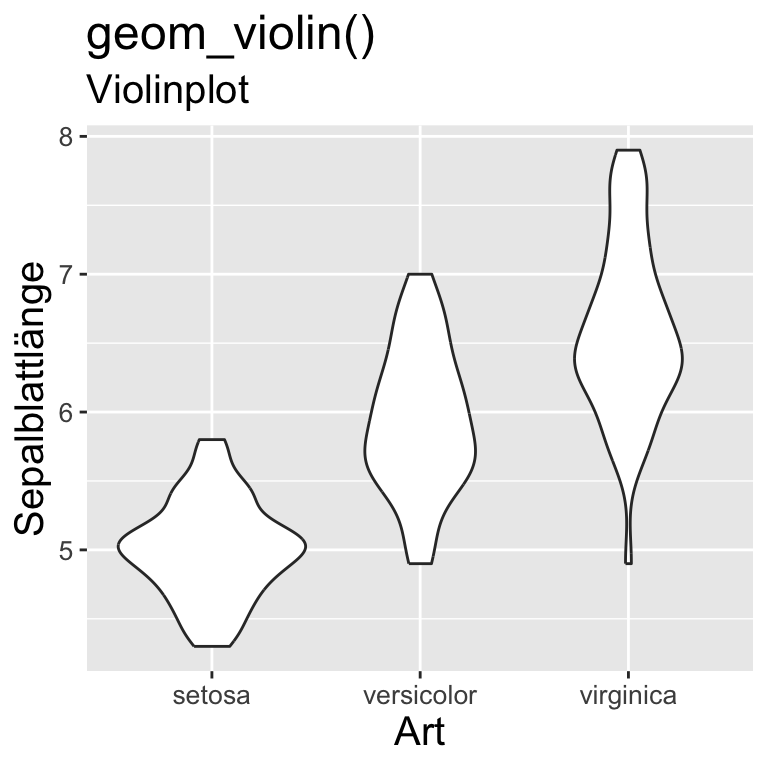
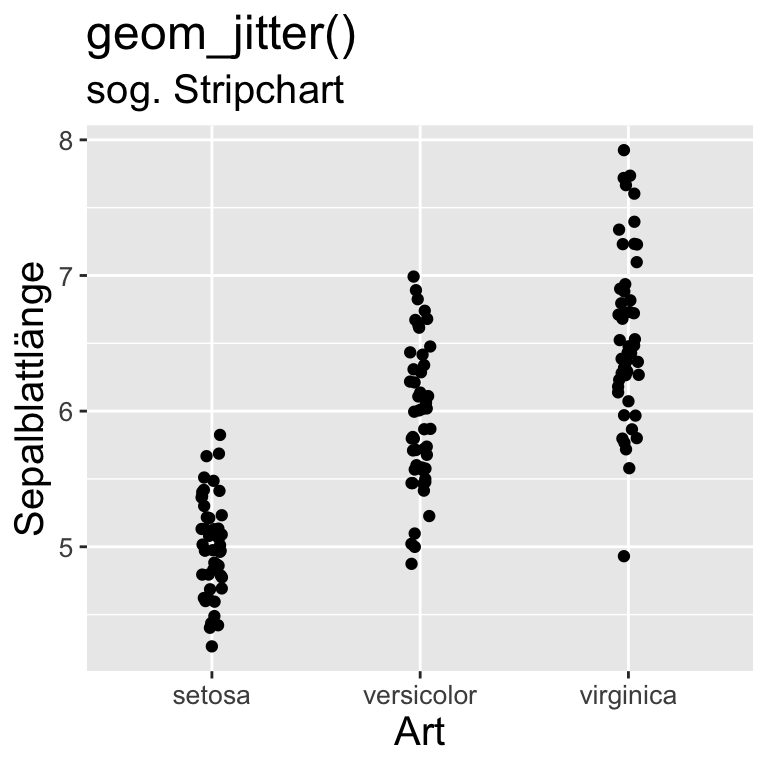
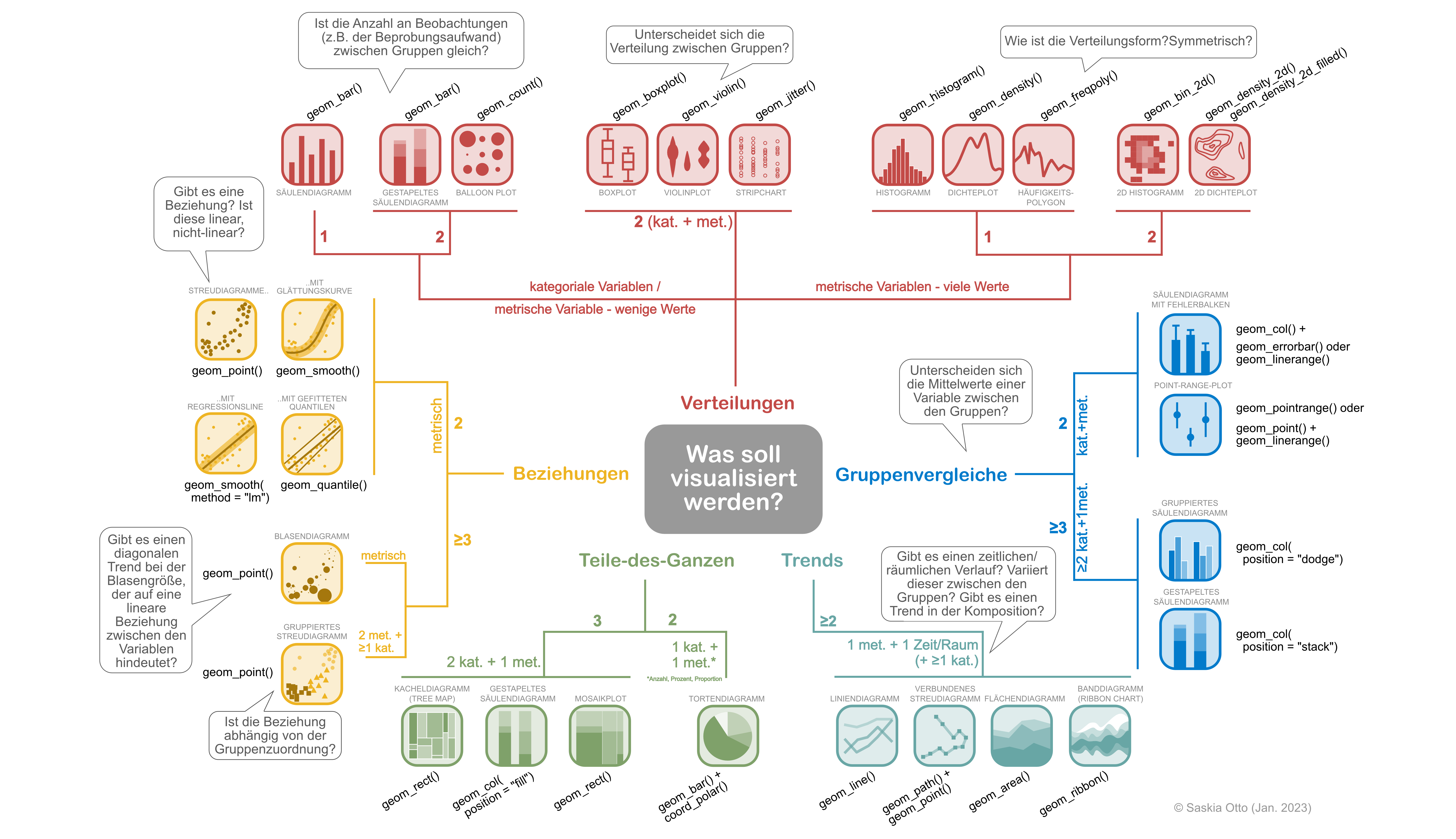
ggplot - geom_XXX() Funktionen
![]()
Wann wird welche Funktion benutzt?
Das ist abhängig von
- dem was dargestellt werden soll (die Frage/Fragekategorie)
- und den Daten-/Merkmalstypen bzw. dem Skalenniveau:
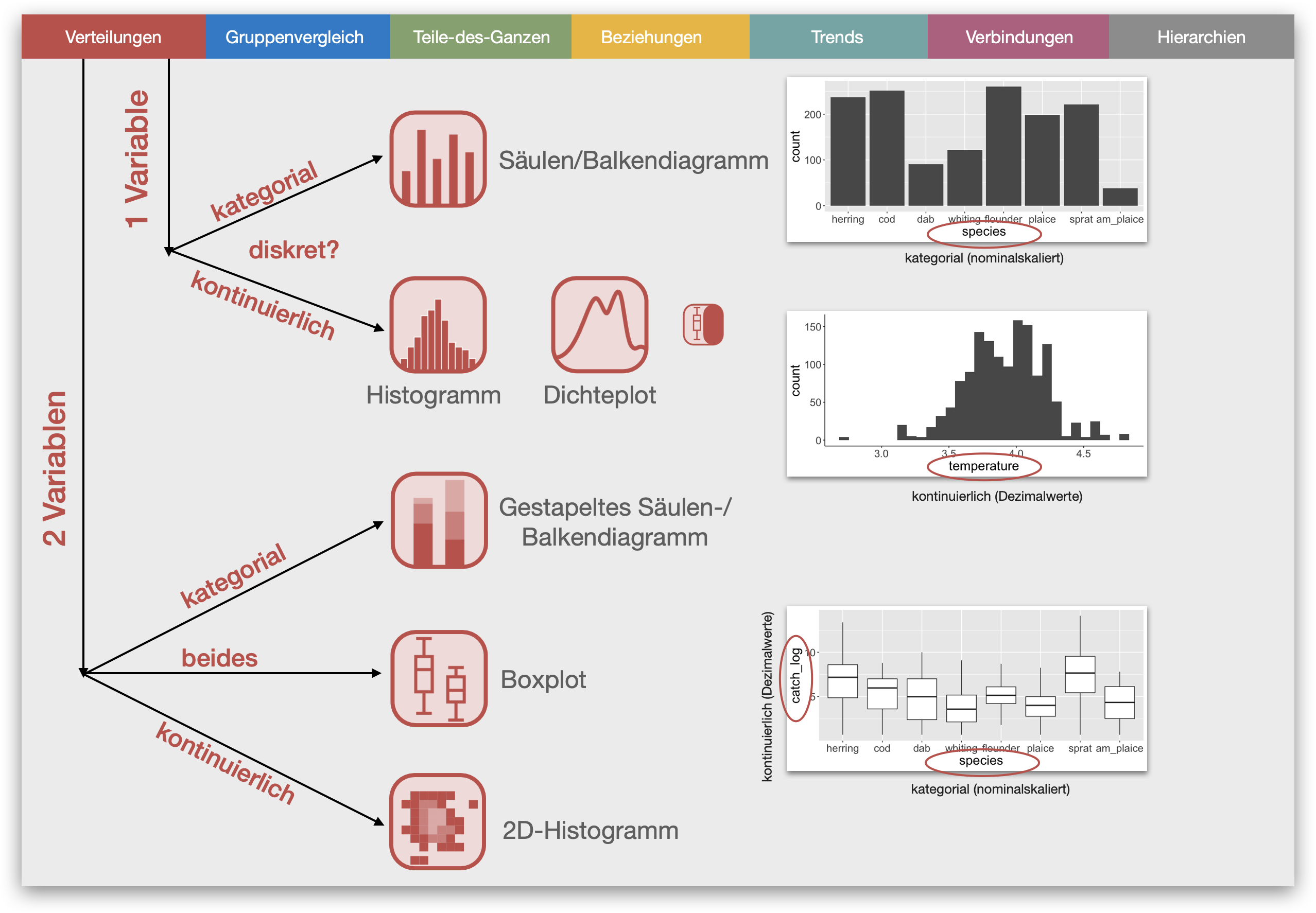
(Häufigkeits)verteilungen
Kategorial (metrisch) (1 VAR)
Metrisch (viele Werte) (1 VARIABLE)
Kategorial (metrisch) (2 VAR)
Code
bshydro15 |>
mutate(
fquarter = factor(lubridate::quarter(date_time)),
fwday = factor(lubridate::wday(date_time))
) |>
ggplot(aes(x=fquarter, y=fwday)) +
geom_count() +
scale_size_area(max_size = 7) +
labs(title = "geom_count()",
subtitle = "sog. Count Plot",
x="Jahresquartal", y="Wochentag") +
theme(text = element_text(size = 13),
axis.text = element_text(size = 10)) 
Metrisch (viele Werte) (2 VARIABLEN)
Code
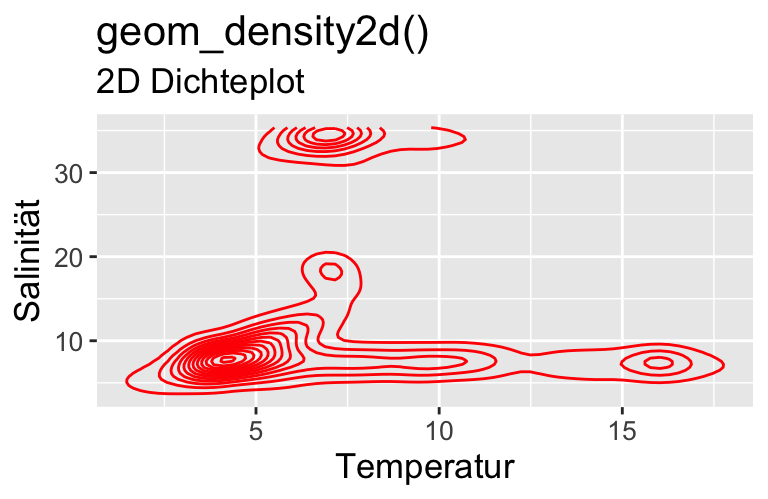
bshydro15 |>
drop_na() |>
ggplot(aes(x=temp, y=psal)) +
geom_bin2d() +
labs(title = "geom_bin2d()",
subtitle = "sog. Heatmap - 2d bin counts",
x = "Temperatur", y = "Salinität") +
scale_fill_gradient2(
trans = "log",
breaks = c(1, 10, 50, 250),
guide = guide_legend(
title = "Häufigkeit (log)")
) +
theme(text = element_text(size = 13),
axis.text = element_text(size = 10)) 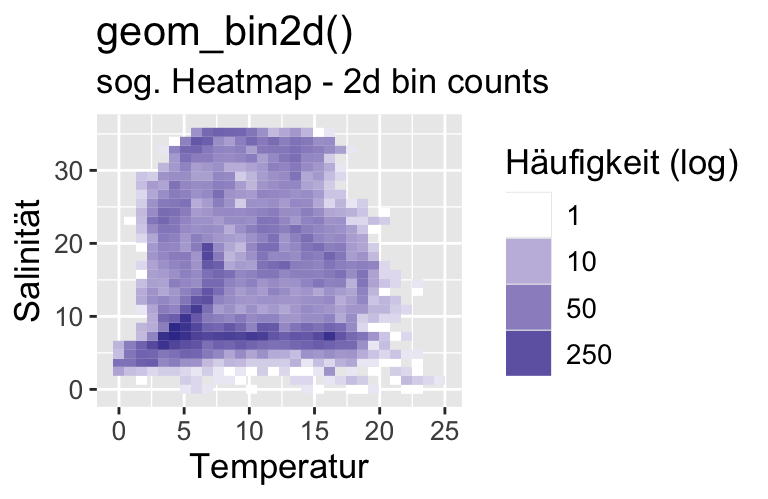
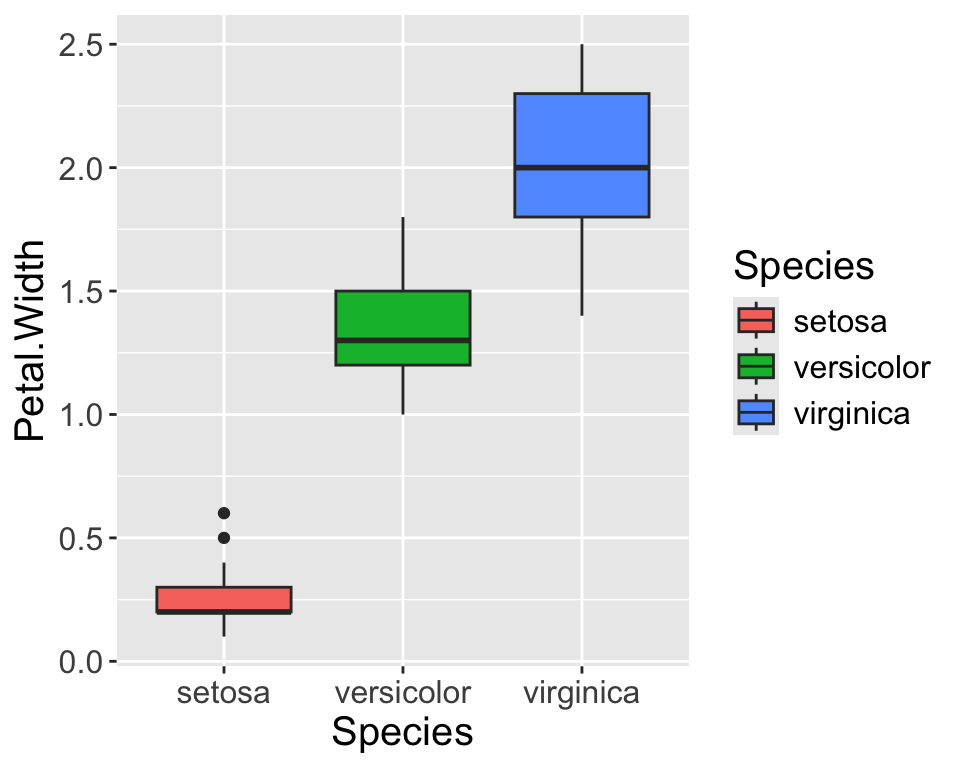
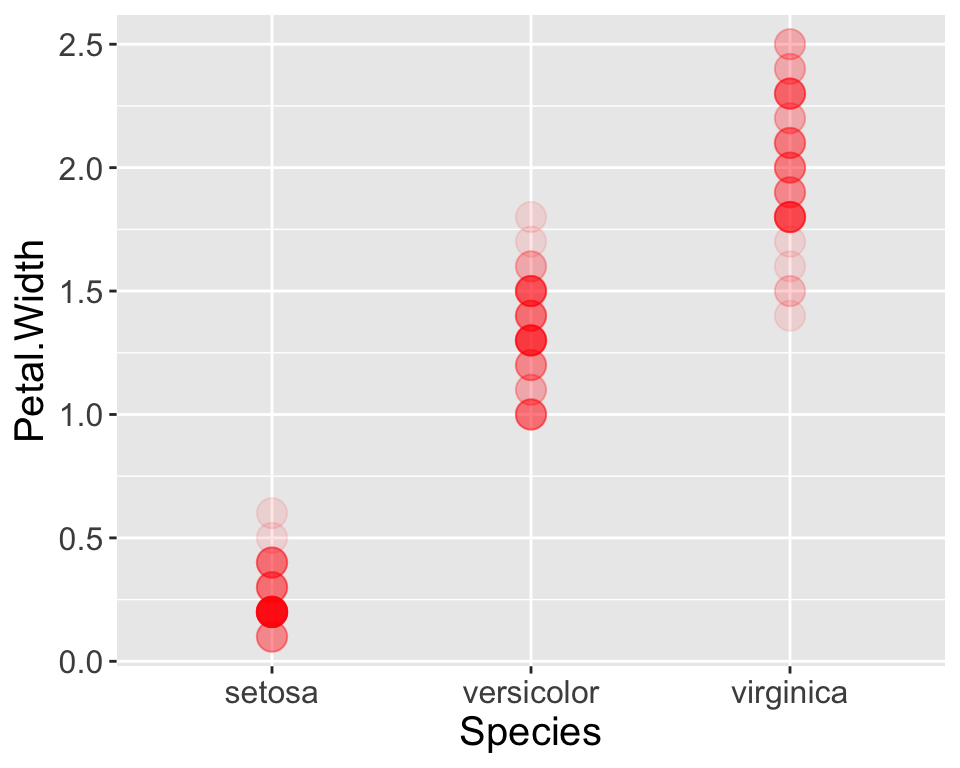
Verteilungen - ein Gruppenvergleich
Kategorial & Metrisch (2 VARIABLEN)
Gruppenvergleich (von Statistiken)
Kategorial & Metrisch (2 VARIABLEN)
Code
iris |>
group_by(Species) |>
summarise(
Mean = mean(Sepal.Length),
SD = sd(Sepal.Length)
) |>
ggplot(aes(x = Species, y = Mean,
fill = Species)) +
geom_col() +
geom_errorbar(aes(ymin = Mean-SD,
ymax = Mean+SD), width = 0.2) +
labs(
title = "geom_col() und geom_errorbar()",
subtitle = "Säulendiagramm mit Fehlerbalken",
x = "Art", y = "mittlere Sepalblattlänge"
) +
guides(fill = "none") +
theme(text = element_text(size = 13),
axis.text = element_text(size = 10)) 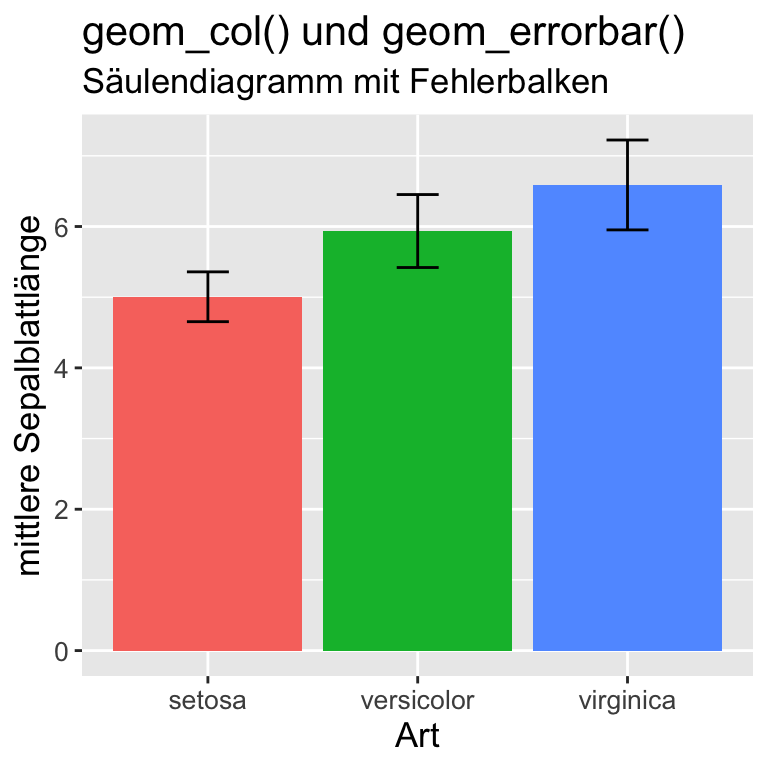
Code
iris |>
group_by(Species) |>
summarise(
Mean = mean(Sepal.Length),
SD = sd(Sepal.Length)
) |>
ggplot(aes(x = Species, y = Mean)) +
geom_pointrange(aes(ymin = Mean-SD,
ymax = Mean+SD)) +
labs(
title = "geom_pointrange()",
subtitle = "sog. Point-Range-Plot",
x = "Art", y = "mittlere Sepalblattlänge"
) +
theme(text = element_text(size = 13),
axis.text = element_text(size = 10)) 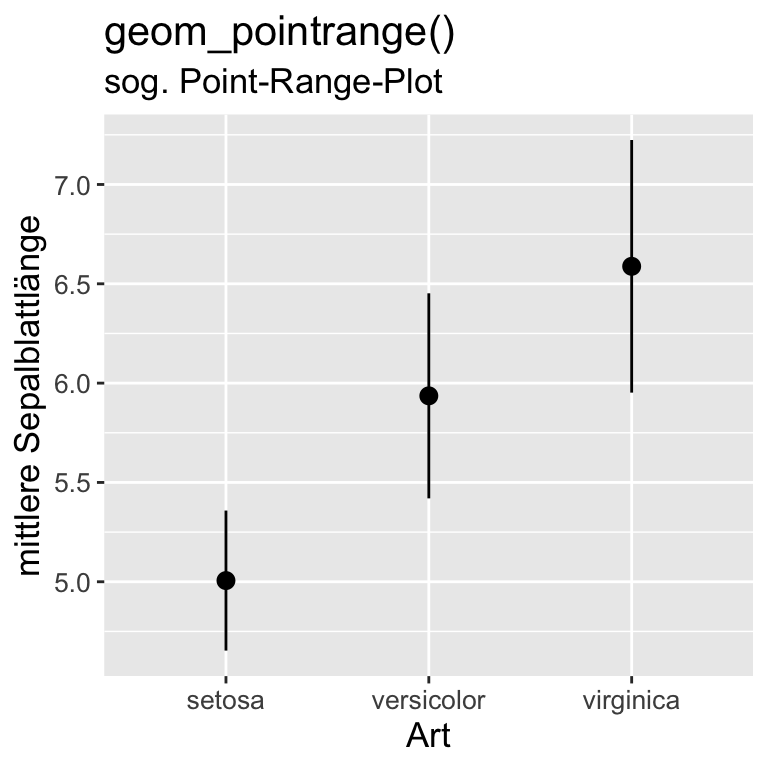
Kategorial & Metrisch (3 VAR)
Code
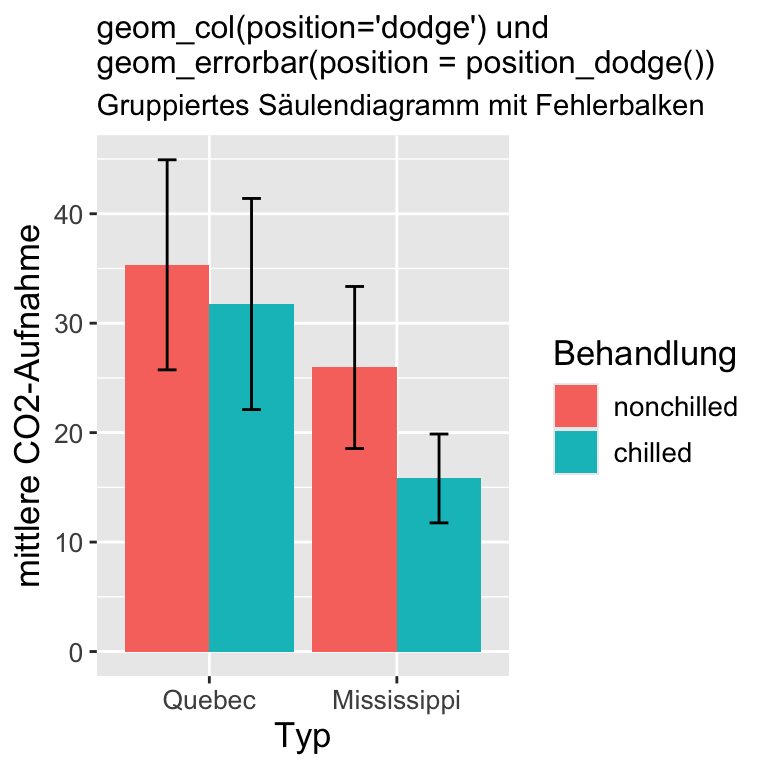
CO2 |>
group_by(Type, Treatment) |>
summarise(
Mean = mean(uptake),
SD = sd(uptake)
) |>
ggplot(aes(x = Type, y = Mean,
fill = Treatment)) +
geom_col(position = "dodge") +
geom_errorbar(aes(ymin = Mean-SD,
ymax = Mean+SD), width = 0.2,
position = position_dodge(.9)) +
labs(
title = "geom_col(position='dodge') und \ngeom_errorbar(position = position_dodge())",
subtitle = "Gruppiertes Säulendiagramm mit Fehlerbalken",
x = "Typ", y = "mittlere CO2-Aufnahme",
fill = "Behandlung"
) +
theme(text = element_text(size = 13),
axis.text = element_text(size = 10),
plot.title = element_text(size = 12),
plot.subtitle = element_text(size = 11)
)
Teile-des-Ganzen / Komposition
Kategorial (& Anteil) (1 bzw. 2 VAR)
Code
data(penguins, package = "palmerpenguins")
# Version 1: den Anteil selbst berechnen
# und dann geom_col() verwenden:
# penguins |>
# group_by(species, island) |>
# summarise(counts = n()) |>
# ggplot(aes(x = factor(1), y = counts,
# fill = species)) +
# geom_col(position = "fill", width = 1) +
# coord_polar(theta = "y") +
# labs(
# title = "geom_col() und coord_polar()",
# subtitle = "Tortendiagramm (pie chart)",
# fill = "Pinguinart"
# ) +
# theme_void()
# Version 2: mit geom_bar()
penguins |>
ggplot(aes(x = factor(1), fill = species)) +
geom_bar(width = 1) +
scale_fill_manual(values = c("#a16a7f", "#9ba16a", "#6a7fa1")) +
coord_polar(theta = "y") +
labs(
title = "geom_bar() und coord_polar()",
subtitle = "Tortendiagramm (pie chart)",
fill = "Pinguinart"
) +
theme_void() +
theme(axis.text = element_blank(),
text = element_text(size = 13))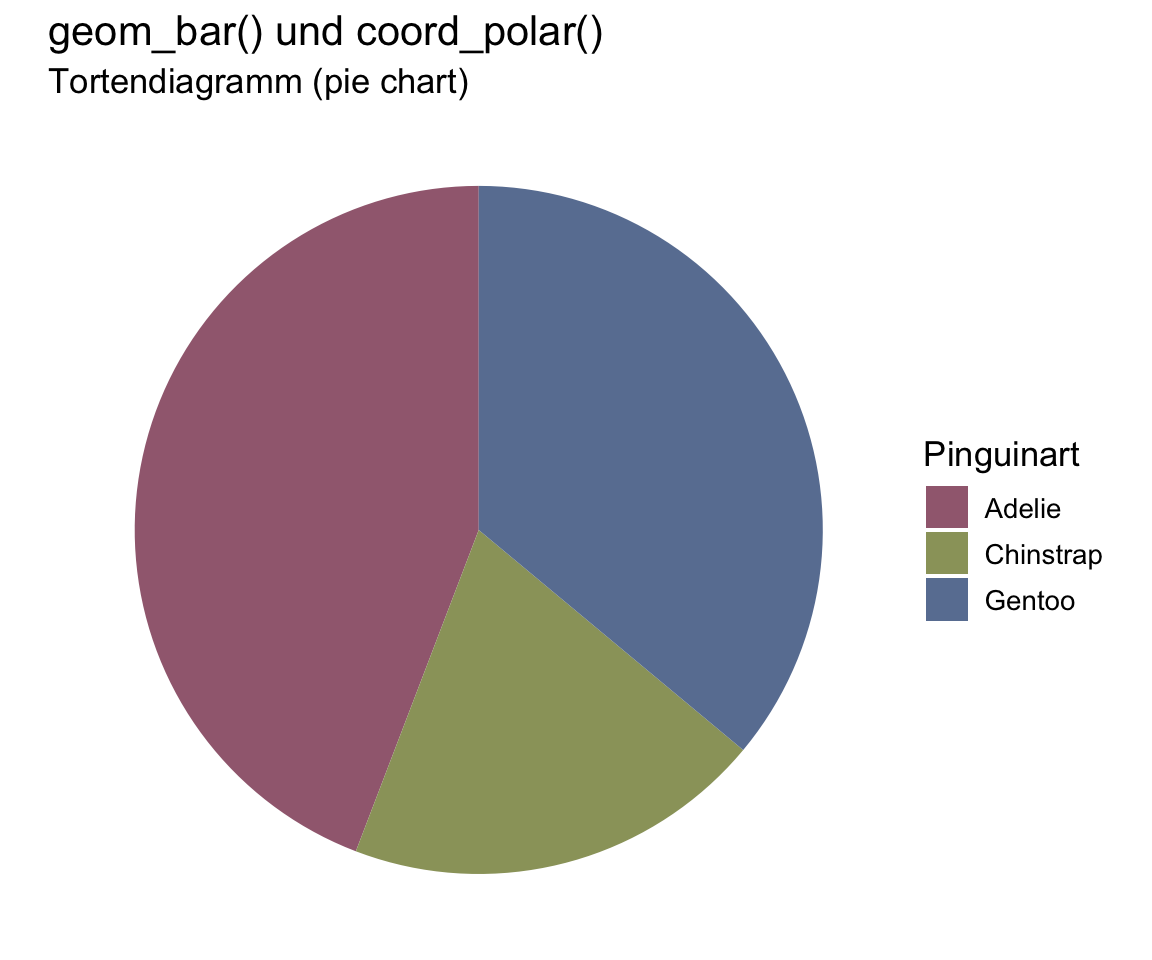
Kategorial (& Anteil) (2 bzw. 3 VAR)
Code
penguins |>
group_by(species, island) |>
summarise(counts = n()) |>
ggplot(aes(x = island, y = counts,
fill = species)) +
geom_col(position = "fill") +
scale_fill_manual(values =
c("#a16a7f", "#9ba16a", "#6a7fa1")) +
labs(
title = "geom_col(position = 'fill')",
subtitle = "Gestapeltes Säulendiagramm",
x = "Insel", y = "Anteil der Häufigkeit",
fill = "Pinguinart"
) +
theme(text = element_text(size = 13),
axis.text = element_text(size = 10))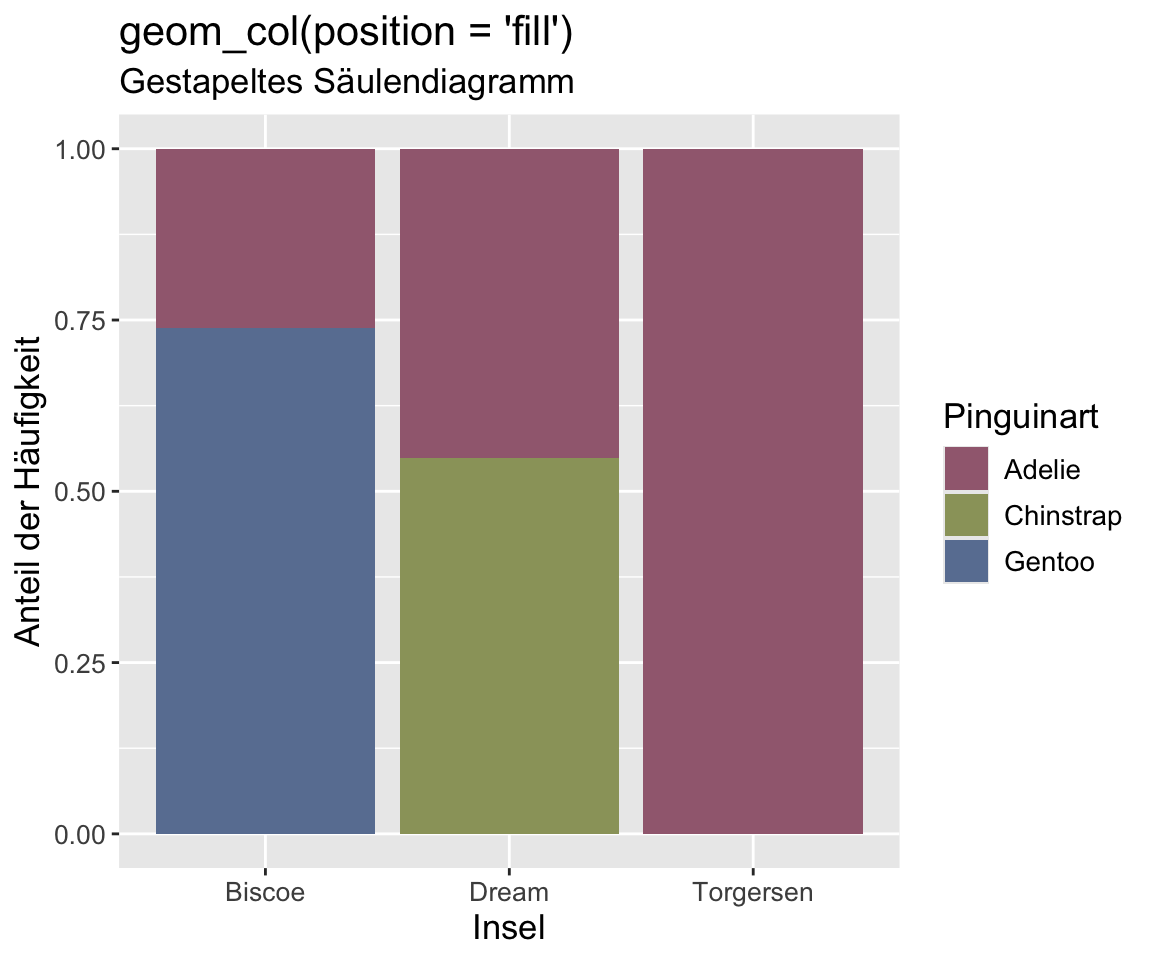
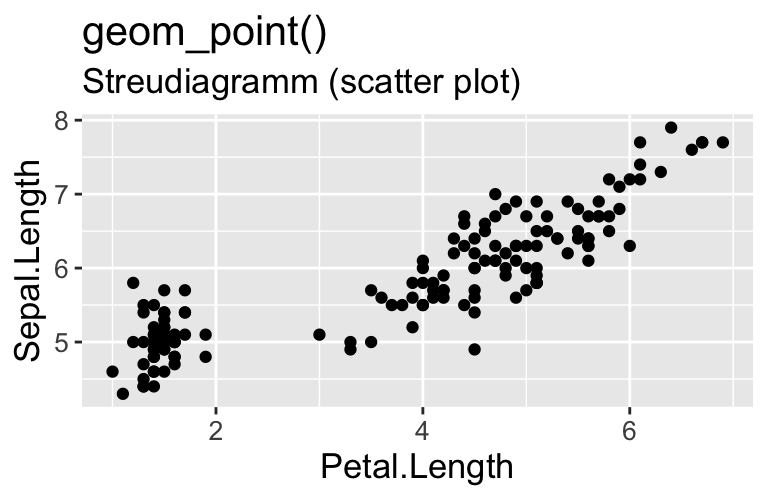
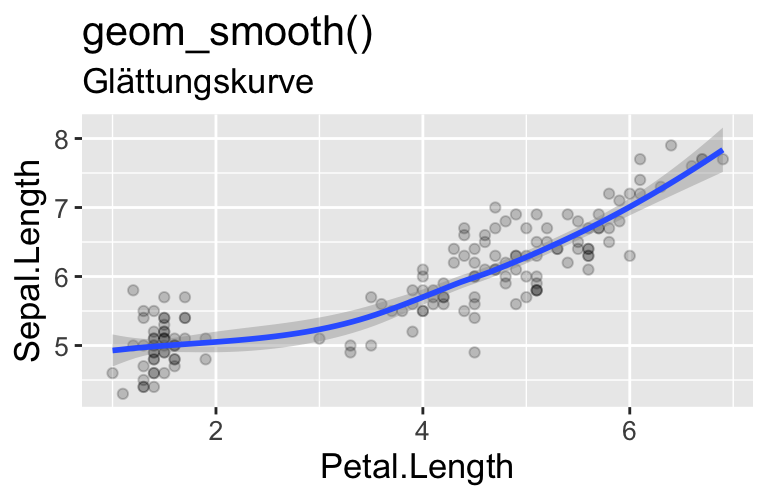
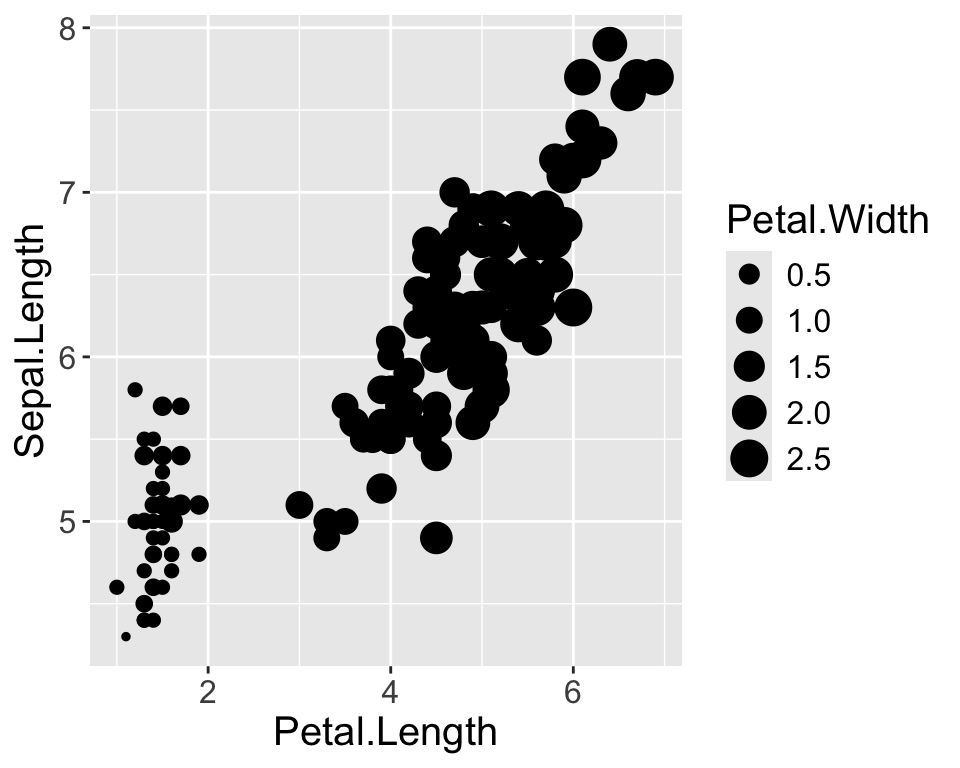
Beziehungen zwischen Variablen
Metrisch (2 VARIABLEN)
Metrisch (3 VARIABLEN X,Y,Z)
Metrisch (X,Y) & Kategorial (Z) (3 VARIABLEN)
Zeitliche Trends
Metrisch & Zeit (2 VARIABLEN)
Code
huron <- data.frame(year = 1875:1972,
level = as.vector(LakeHuron))
ggplot(huron, aes(year)) +
geom_ribbon(aes(ymin = level - 1,
ymax = level + 1), fill = "grey70") +
geom_line(aes(y = level), colour = "grey65") +
labs(
title = "geom_ribbon()",
subtitle = "Banddiagramm (ribbon chart)",
x = "Jahre", y = "Huronsee \nPegelstand (in Fuß)"
) +
theme(text = element_text(size = 15),
axis.text = element_text(size = 10)) 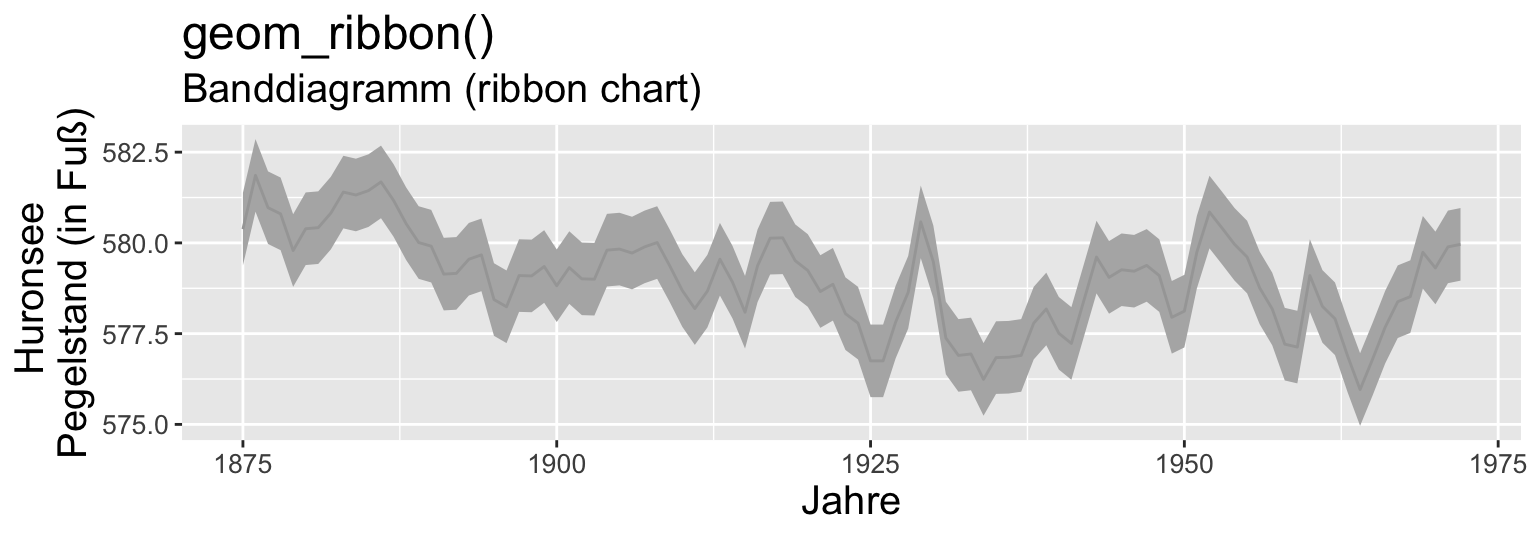
Metrisch, Zeit & Kategorial (3 V.)
Code
cod |>
ggplot(aes(x = year, y = ssb)) +
geom_area(aes(fill = area)) +
labs(
title = "geom_area()",
subtitle = "(Gestapeltes) Flächendiagramm",
x = "Jahr",
y = "Gesamtgröße Kabeljau-Bestand (in Tonnen)",
fill = "Gebiet"
) +
guides(fill = guide_legend(nrow = 3,
title.position = "bottom")) +
theme(text = element_text(size = 14),
axis.text = element_text(size = 10),
legend.position = "bottom",
legend.justification = "left",
legend.text = element_text(size = 9))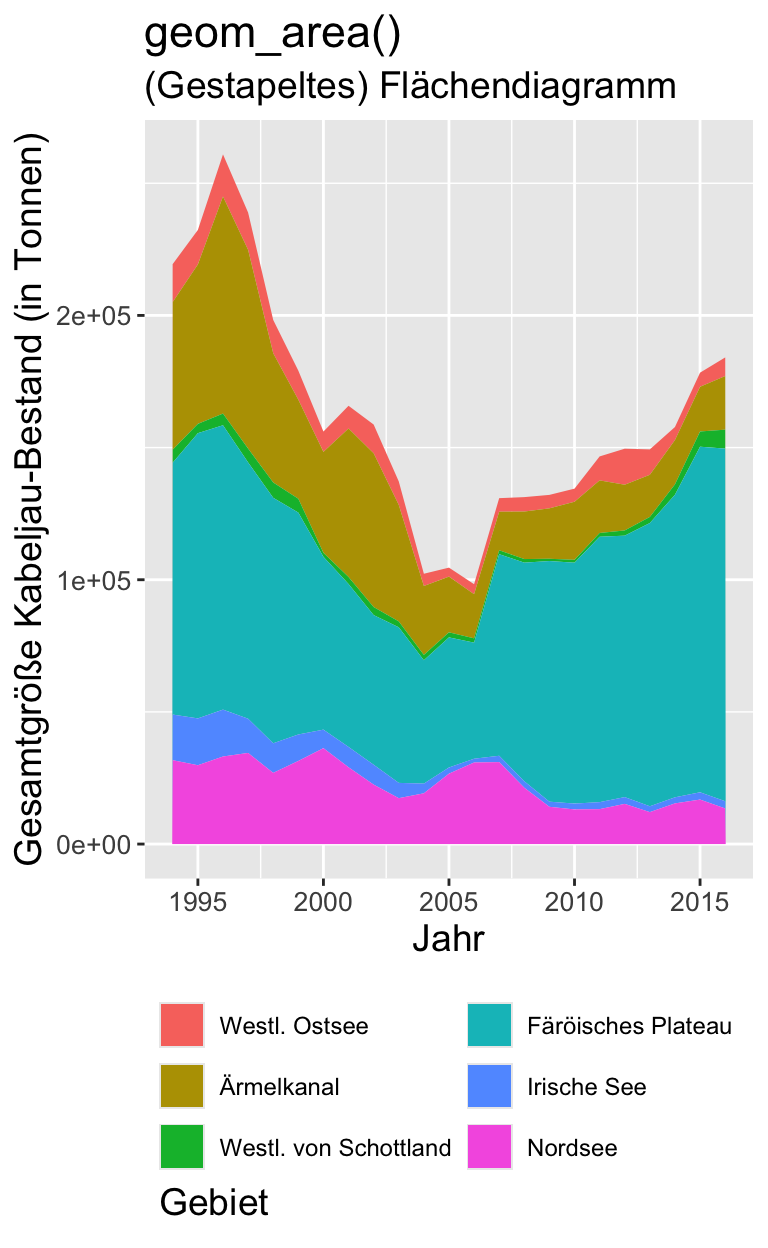
Your turn …
![]()
Quiz 1-5 | ggplots
![]()
![]()
Q1 | Häufigkeitsverteilung
Q2 | Gruppenvergleich
Q3 | Gruppenvergleich
Q4 | Beziehung
Q5 | Beziehung
Übungsaufgabe

Optionale swirl-Lektionen zur Vertiefung
![]()
Kurs DSB-02-Datenexploration mit R
- L02-Erste grafische Analyse
- L04-Basisgrafik Histogramm
- L05-Basisgrafik Boxplot
- L06-Basisgrafik Säulendiagramm
- L07-Basisgrafik Streudiagramm
- L08-Globale Einstellungen und Grafiken_exportieren
Kurs DSB-04-Datenvisualisierung mit ggplot2
- L01-Geschichtete Grammatik-Eine Einfuehrung in gglot2
- L02-geom_Funktionen für Verteilungen kategorialer Variablen
- L03-geom_Funktionen für Verteilungen kontinuierlicher Variablen
- L04-geom_Funktionen für Gruppenvergleiche von Verteilungen
- L05-geom_Funktionen für Gruppenvergleiche
- L06-geom_Funktionen für Beziehungen
- L07-geom_Funktionen für Trends
Übungsskripte
![]()
- Vervollständigung eines Lückenskripts zur Erstellung von Histogrammen, Boxplots, Säulen- und Streudiagrammen mit ‘ggplot2’: DS1_W09_Uebungsskript_VisualisierungMitR.R
Fallstudie

Sie sind jetzt so weit, …
..dass Sie mit der grafischen deskriptiven Statistik starten können! Sie können entweder beide Varianten ausprobieren oder nur ggplot2.
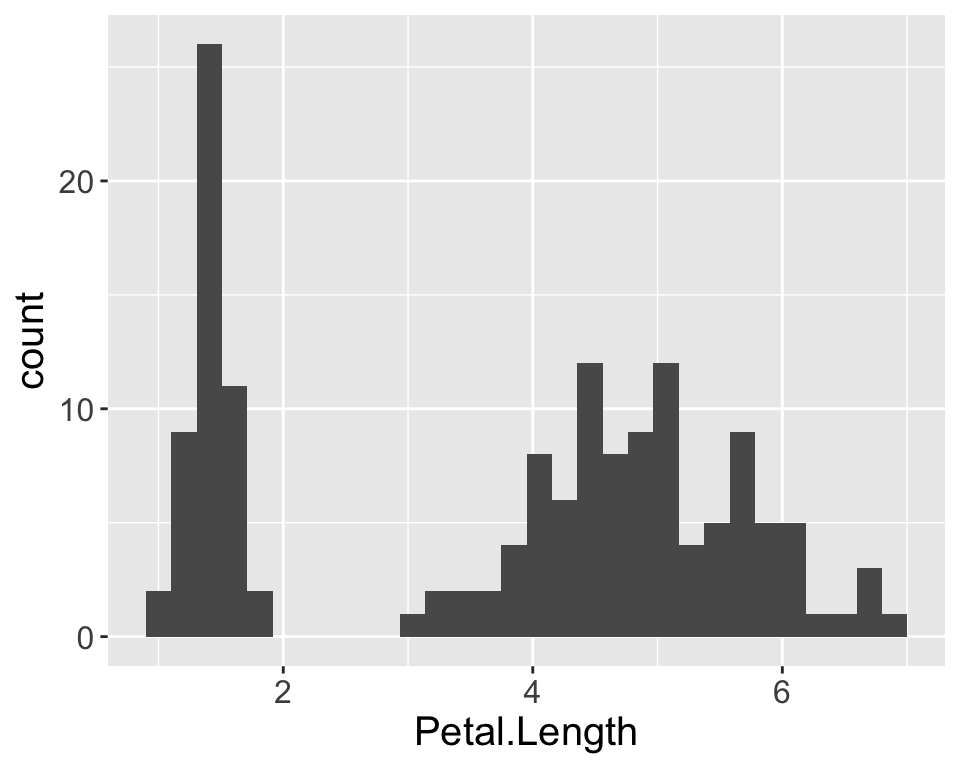
Optionale Challenge 1: Grafik erstellen
![]()
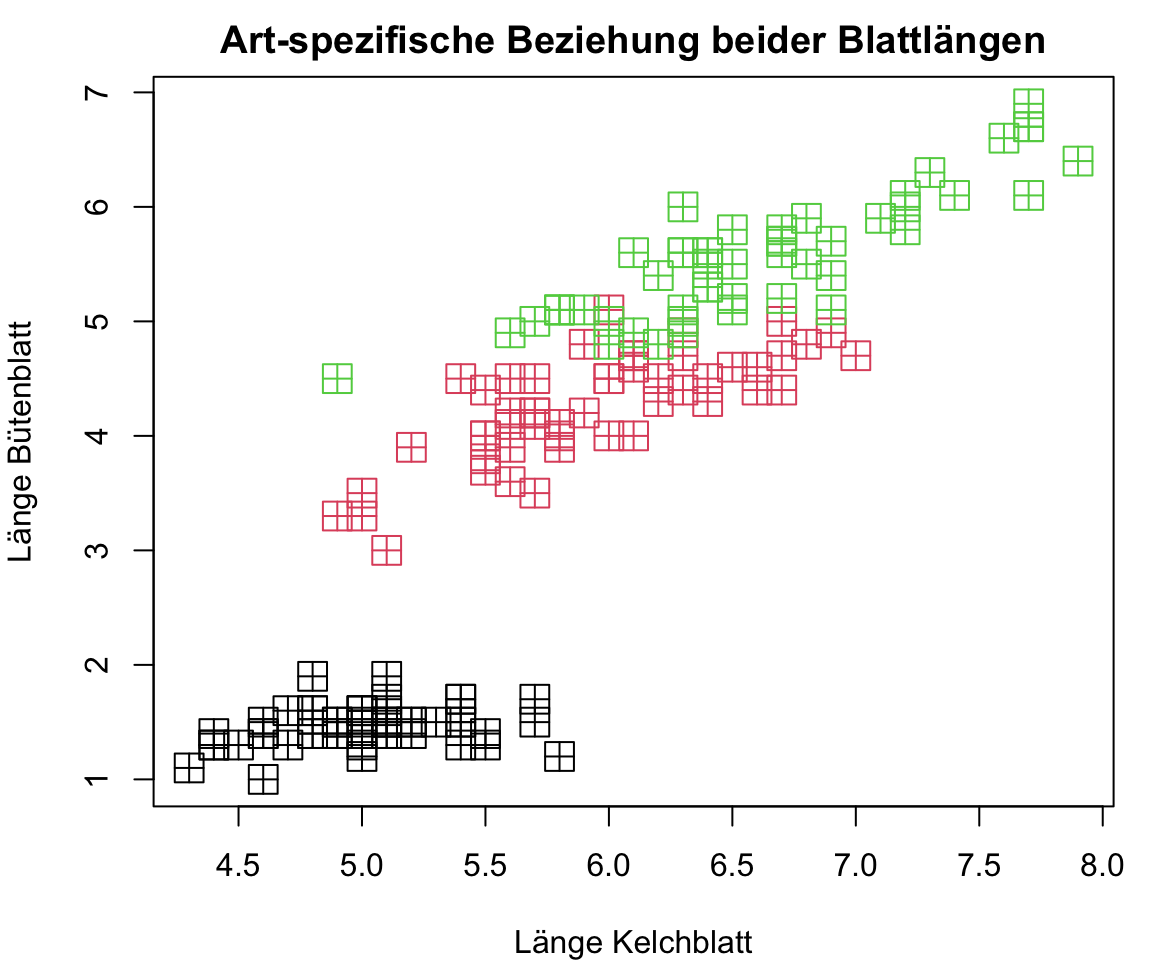
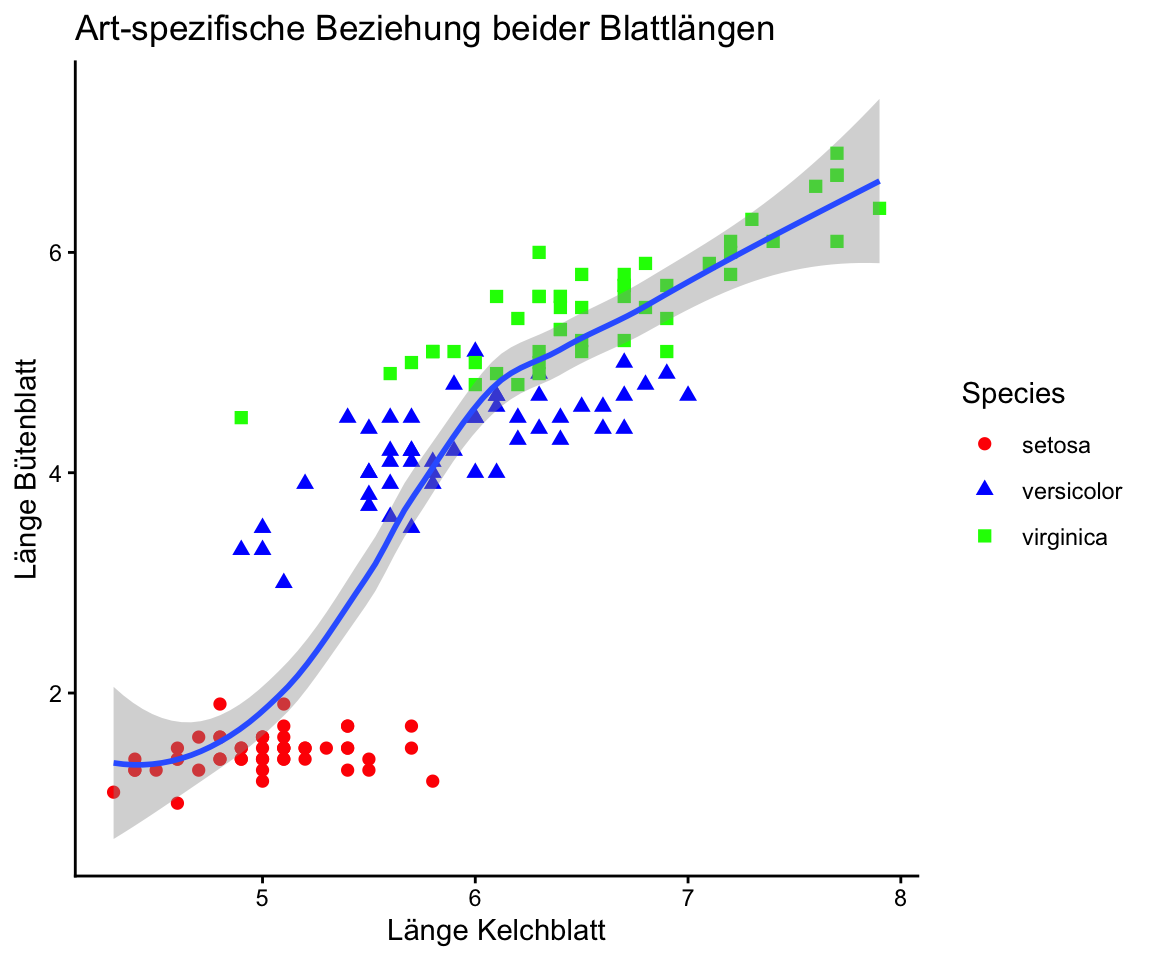
Versuchen Sie einen der beiden Plots mit dem iris Datensatz in base R bzw. ‘ggplot2’ zu reproduzieren:
Optionale Challenge 2: Grafik erstellen
und abspeichern
![]()
Versuchen Sie folgenden Plot mit dem iris Datensatz in base R zu reproduzieren und diesen als PDF abzuspeichern:
Wie fühlen Sie sich jetzt…?

Total konfus?

Dann schauen Sie sich doch mal…
- …folgende Weblinks zur Diagrammerstellung mit den built-in Funktionen für Basisplots an:
- das ggplot2 Cheatsheet genauer an.
- und lesen Sie Kapitel 3 Data visualisation über Datenvisualisierung mit ggplot2 in ‘R for Data Science’.
Posit Cheatsheet
Überblick an Funktionen im ggplot2 Paket

Cheatsheet zum ggplot2 Paket frei verfügbar unter diesem Link.
Total gelangweilt?

Dann testen Sie doch Ihr Wissen in folgendem Abschlussquiz und probieren Sie diese Challenges..
Abschlussquiz
![]()

Bei weiteren Fragen: saskia.otto(at)uni-hamburg.de

Diese Arbeit is lizenziert unter einer Creative Commons Attribution-ShareAlike 4.0 International License mit Ausnahme der entliehenen und mit Quellenangabe versehenen Abbildungen.
Kurswebseite: Data Science 1