Datenaufbereitung mit tidyverse:
Transformation und Anreicherung
Data Science 1 - Programmieren & Visualisieren
Saskia Otto & Monika Eberhard
Universität Hamburg, IMF
Wintersemester 2025/2026
Lernziele
![]()
Am Ende dieser VL- und Übungseinheit werden Sie
- … die grundlegenden Kenntnisse der Datensatztransformation, -aggregation und -kombination entsprechend einer Fragestellung beherrschen.
- Dazu gehört
- das Sortieren und Filtern von Zeilen,
- das Auswählen und die Neuerstellung von Spalten, sprich Variablen,
- das Aggregieren und die (Gruppen-spezifische) Erstellung von deskriptiven Statistiken und
- das Kombinieren von relationalen Daten.
Zur Erinnerung
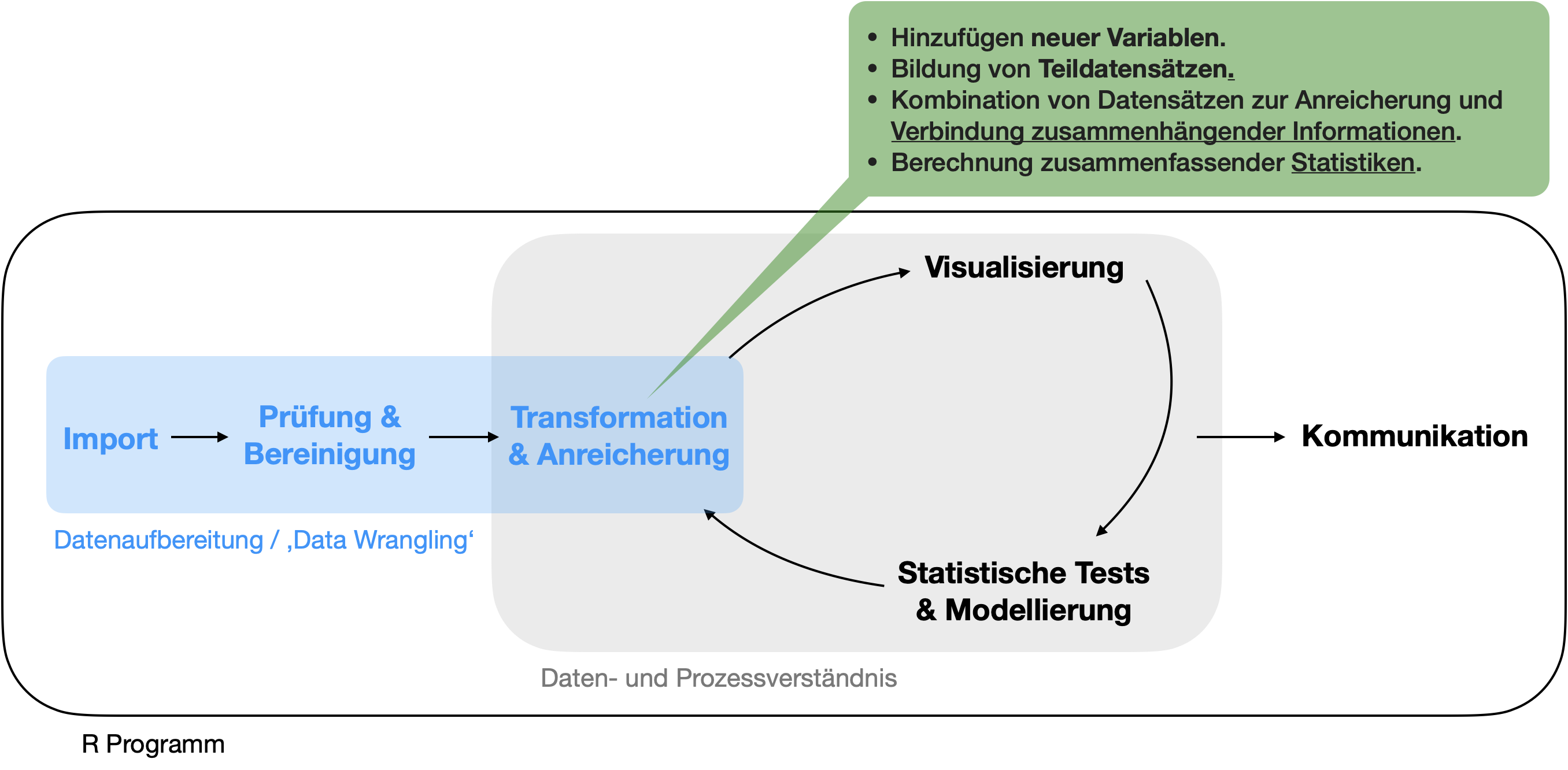
Die 3 Komponenten des ‘Data Wrangling’
Kernfunktionen von dplyr

| Typische Manipulationen | Kernfunktion in ‘dplyr’ |
|---|---|
| Manipuliere Beobachtungen (Zeilen) | filter(), arrange() |
| Manipuliere Variablen (Spalten) | select(), rename(), mutate(), transmute(), |
| Fasse Beobachtungen zusammen | summarise() |
| Gruppiere Beobachtungen | group_by(), ungroup() |
| Kombiniere Tabellen | bind_rows, bind_cols und join_ Funktionen |
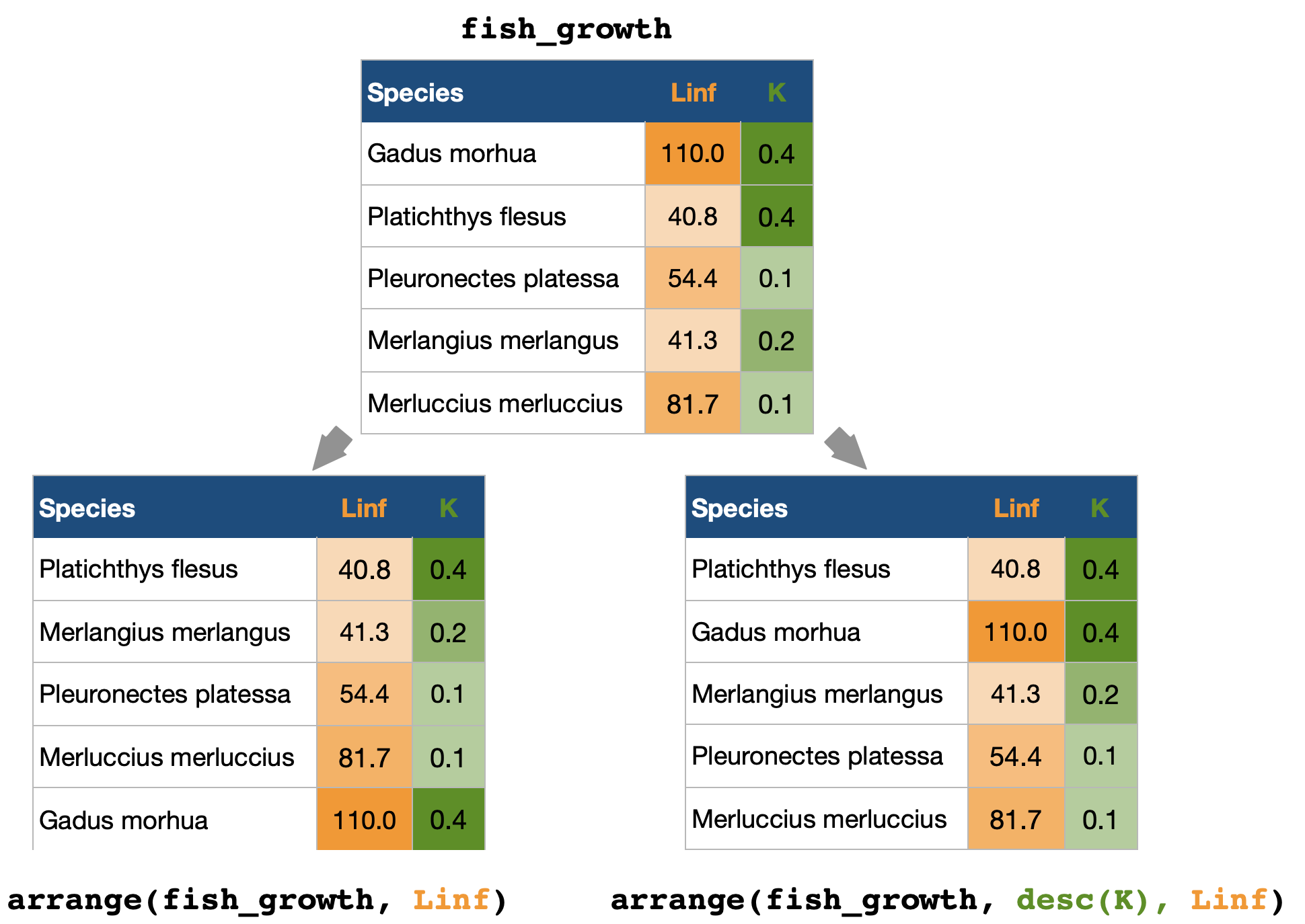
Eine Demonstration mit Wachstumsinformation von 5 Fischarten

(‘Linf’ = mittlere maximale Länge, ‘K’ = Rate in der sich der Fisch an ‘Linf’ annähert)
Mit freundlicher Genehmigung der Fotografen von fishbase.org (Konstantinos I. Stergiou, Jim Greenfield) und uwphoto.no(Rudolf Svensen).
Transformation: Zeilen- und Spaltenmanipulation

Zeilenmanipulation | Sortieren
arrange() → sortiert Zeilen (also Beobachtungen) nach spezifischen Variablen:
Zeilenmanipulation | Filtern
filter() → extrahiert Reihen, die ein logisches Kriterium erfüllen:

Zeilenmanipulation | Verknüpftes Filtern
Verknüpfte Abfragen in filter() werden mit Kommata aufgeführt (UND-Operator):

Spaltenmanipulation | Variablenauswahl
select() → extrahiert Spalten nach Namen oder mittels Helferfunktion:
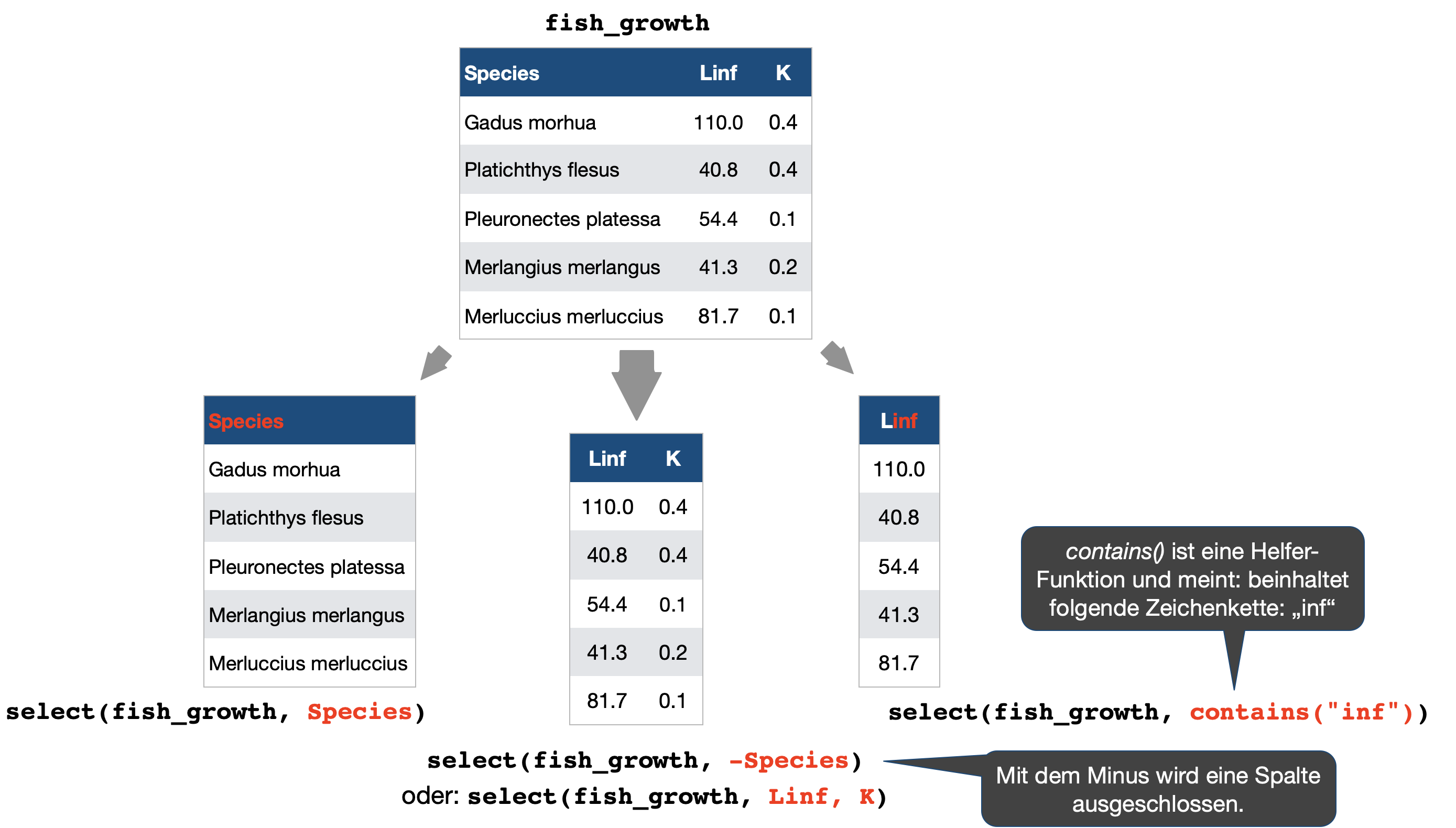
Spaltenmanipulation | Helferfunktion 1
Übersicht der Helferfunktionen für select()

Quelle: ältere Version des sog. cheatsheets
Data Transformation with dplyr (lizensiert unter CC-BY-SA).
Spaltenmanipulation | Namensänderung
rename() → ändert die Namen einzelner Variablen (neuerName = alterName):
Spaltenmanip. | Variablenerstellung
mutate() und transmute() → erstellen neue Variablen:

Spaltenmanipulation | Helferfunktion 2
Übersicht der Helferfunktionen für mutate() / transmute()
Sie können jede Berechnung mit einer Variablen machen, solange diese vektorisiert ist:

Quelle: ältere Version des sog. cheatsheets Data Transformation with dplyr (lizensiert unter CC-BY-SA).
Kombinieren von Befehlen
Hilfreiches Tool
Bevor wir jetzt in die Datenmanipulation starten, werden wir ein sehr hilfreiches Werkzeug für verknüpfte Operationen kennenlernen!!!
Der ‘pipe’ Operator
2 Varianten des ‘pipe’ Operators
Das Original: %>%
- Wurde zunächst durch das Paket ‘magritr’ zur Verfügung gestellt.
- Ist nun aber auch über alle ‘tidyverse’ Pakete zugänglich.
- → D.h., es muss erst eines dieser Pakete geladen werden, um den Operator nutzen zu können.

Neuer nativer Pipe Operator: |>
- Ab der R Version 4.1 gibt es diesen nützlichen Operator auch direkt in base R.
- → D.h., es muss KEIN Paket extra geladen werden.
- In diesem Modul (auch in den swirl-Kursen) werden wir NUR mit dem nativen Operator
|>arbeiten!
Wozu ein solcher Operator?
Vereinfacht Operationen:
- Mit
|>können diverse Funktionsaufrufe sequenziell miteinander verknüpfen werden, ohne Zwischenobjekte zu erstellen. |>leitet den Inhalt des Objekts auf der linken Seite in den Ausdruck auf der rechten Seite.- Weitere Schritte können einfach und überall der Sequenz an Operationen hinzugefügt, entfernt oder auskommentiert werden.
‘Piping’ mit |> | Wie?
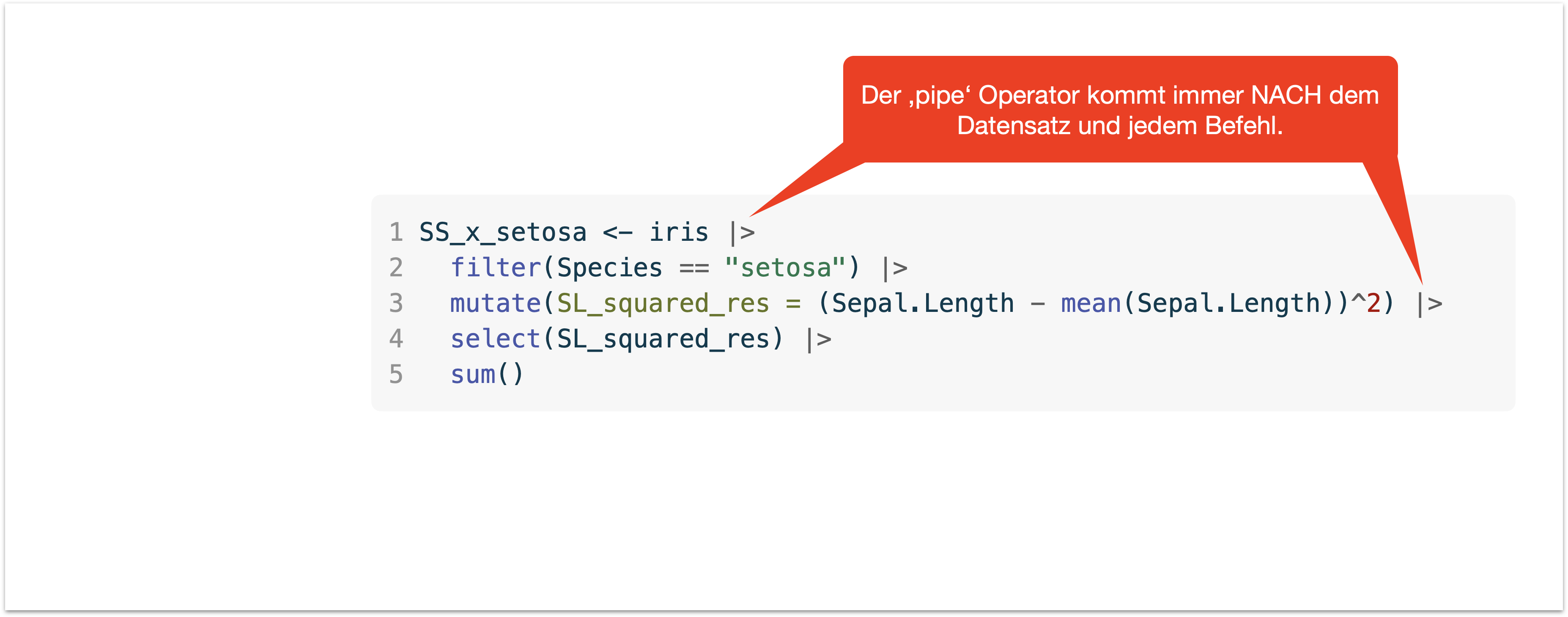
‘Piping’ mit |> | Interpretation

Erinnert doch fast an ein Kochrezept, oder?
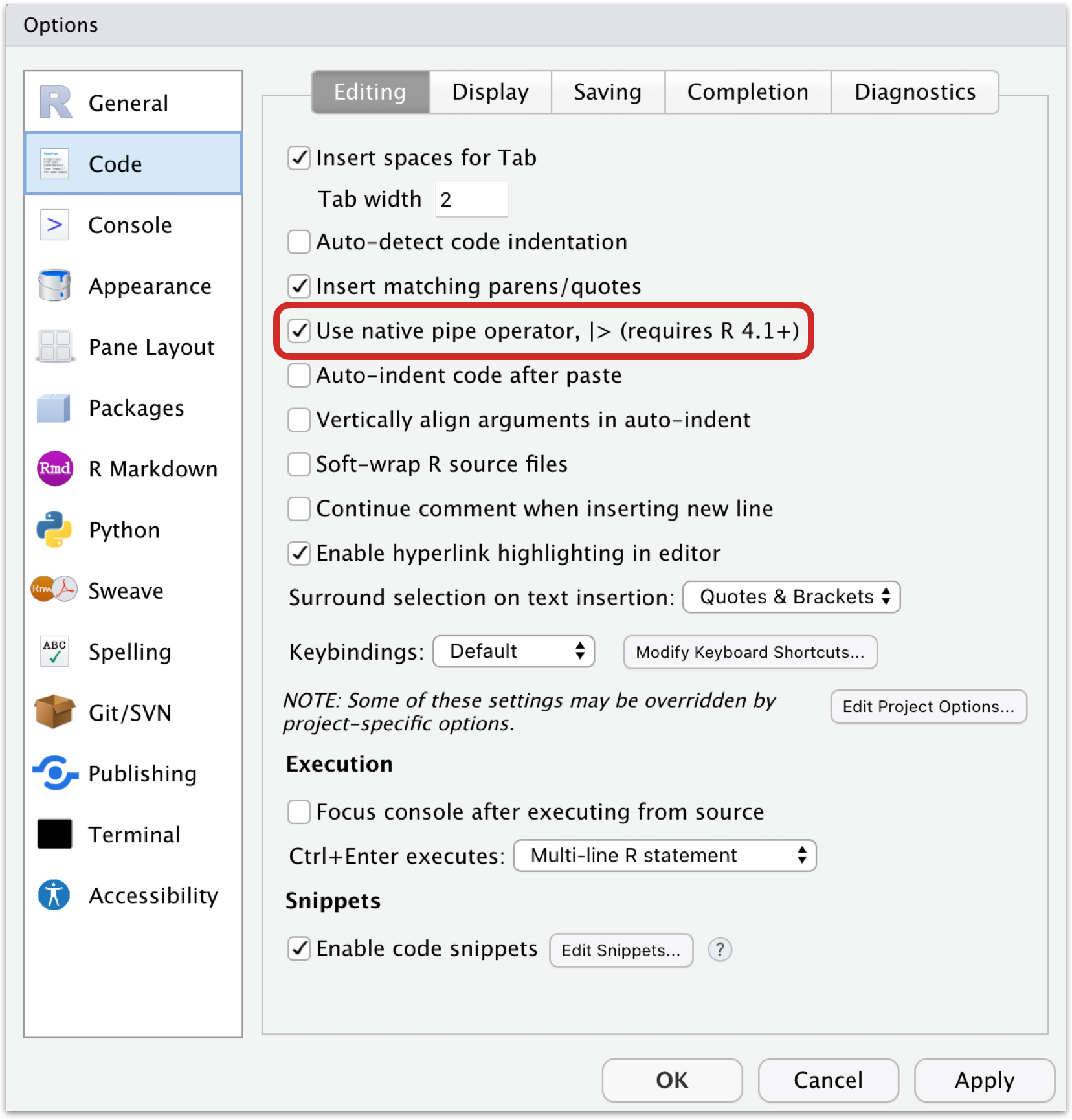
Shortcut für |>
Kleiner Tipp:
Der Pipe-Operator lässt sich mit dem Shortcut strg (bzw. cmd) + shift + m automatisch schreiben.
Um den nativen Operator standardmäßig zu nutzen, muss vorweg unter
- ‘Preferences > Code’ bzw.
- ‘Tools > Global Options..’ > Code
- bei ‘Use native pipe operator, |> (requires R 4.1+)’
ein Häkchen gesetzt werden!
Your turn …
![]()
05:00
Quiz 1-3 | Zeilen- und Spaltenmanipulation
![]()
![]()
Q1
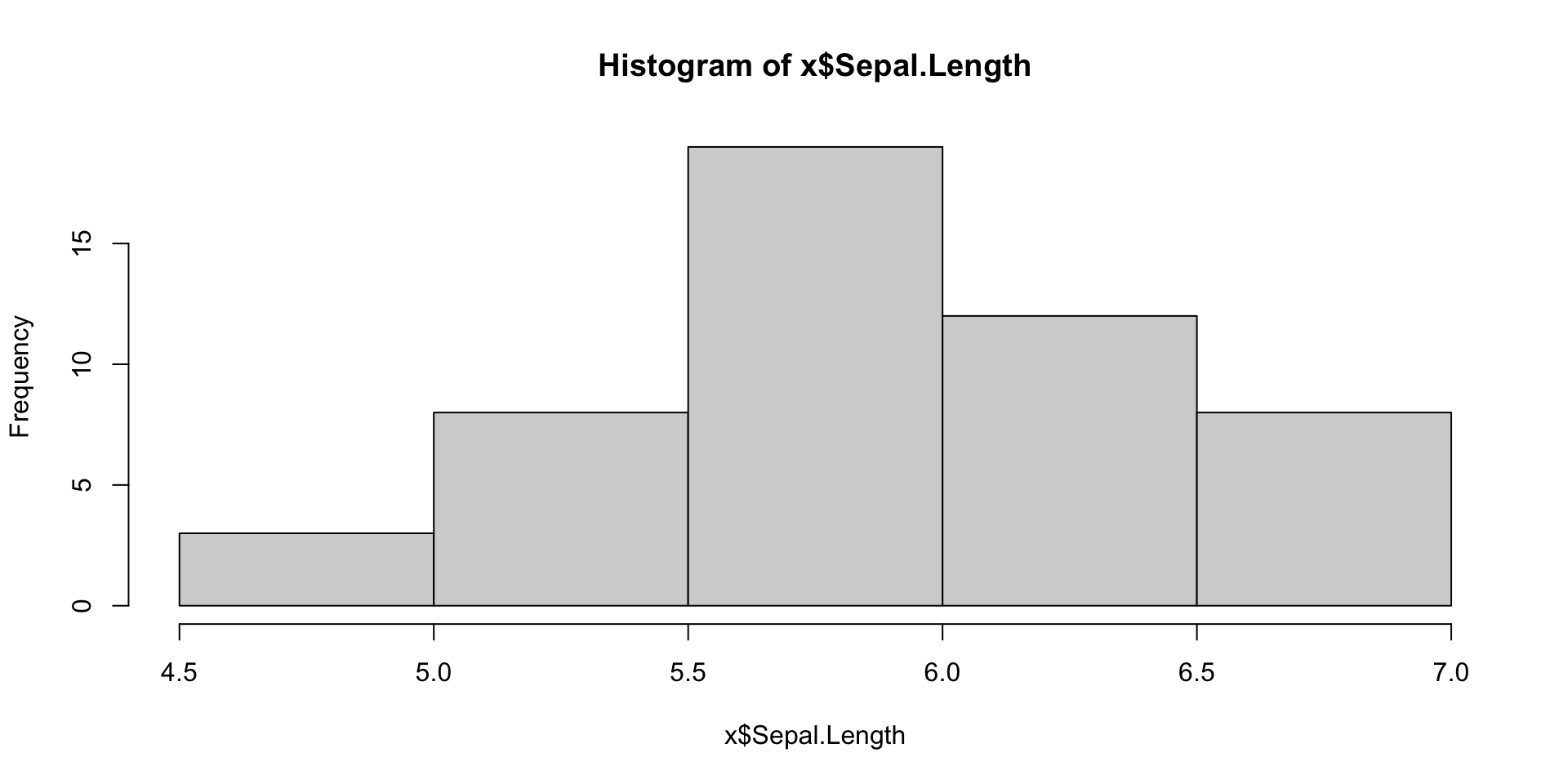
Wir wollen uns die Verteilung der Kelchblattlänge bei der Art Iris versicolor anschauen:
Q2
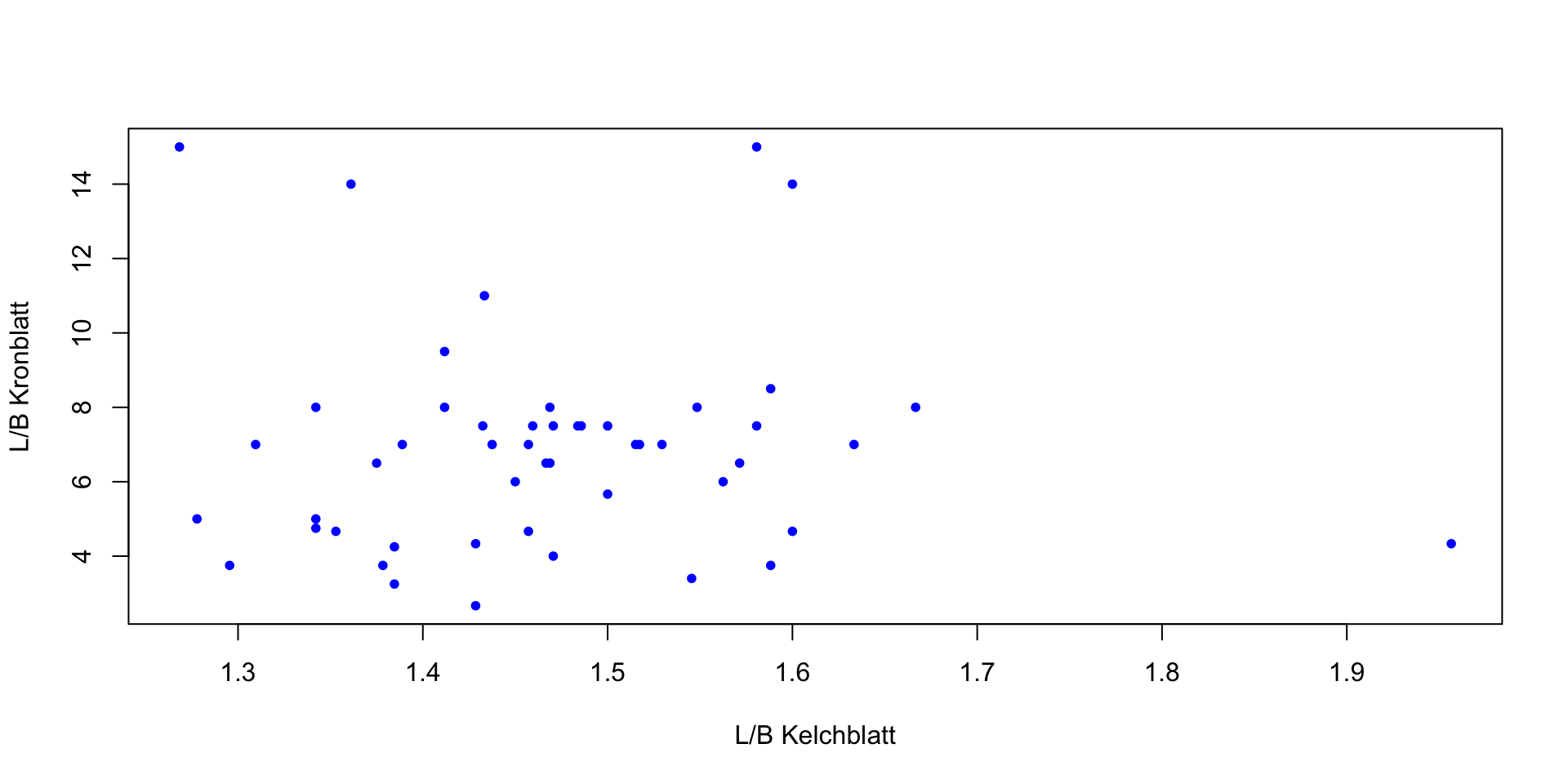
Gibt es eine Beziehung zwischen dem Längen/Breiten-Verhältnis bei Kron- und Kelchblättern der Art Iris setosa? Um dies grafisch untersuchen zu können, transformieren Sie den iris Datensatz:
Q3
Erstellen Sie einen ‘tibble’ basierend auf dem iris Datensatz, welcher nur die Spalte ‘Species’ enthält und eine neue (absteigend sortierte) Spalte mit dem Quotienten aus der Kelchblattlänge zu -breite. Der ‘tibble’ soll aber nur die Arten I. versicolor und I. virginica und Quotientenwerte größer 2 enthalten:
iris |>
mutate(sepal_lw = Sepal.Length/Sepal.Width) |>
filter(Species %in% c('versicolor', 'virginica'), sepal_lw > 2) |>
select(Species, sepal_lw) |>
arrange(desc(sepal_lw)) |>
as_tibble()# A tibble: 79 × 2
Species sepal_lw
<fct> <dbl>
1 virginica 2.96
2 versicolor 2.82
3 virginica 2.75
4 versicolor 2.74
5 versicolor 2.73
6 virginica 2.73
7 virginica 2.68
8 virginica 2.64
9 virginica 2.57
10 virginica 2.53
# ℹ 69 more rowsTransformation: Daten aggregieren

Datenaggregation | Zusammenfassung
summarise() → reduziert Variablen zu Einzelwerten:

Datenaggregation | Helferfunktion
Hilfreiche Funktionen, die zusammenfassen
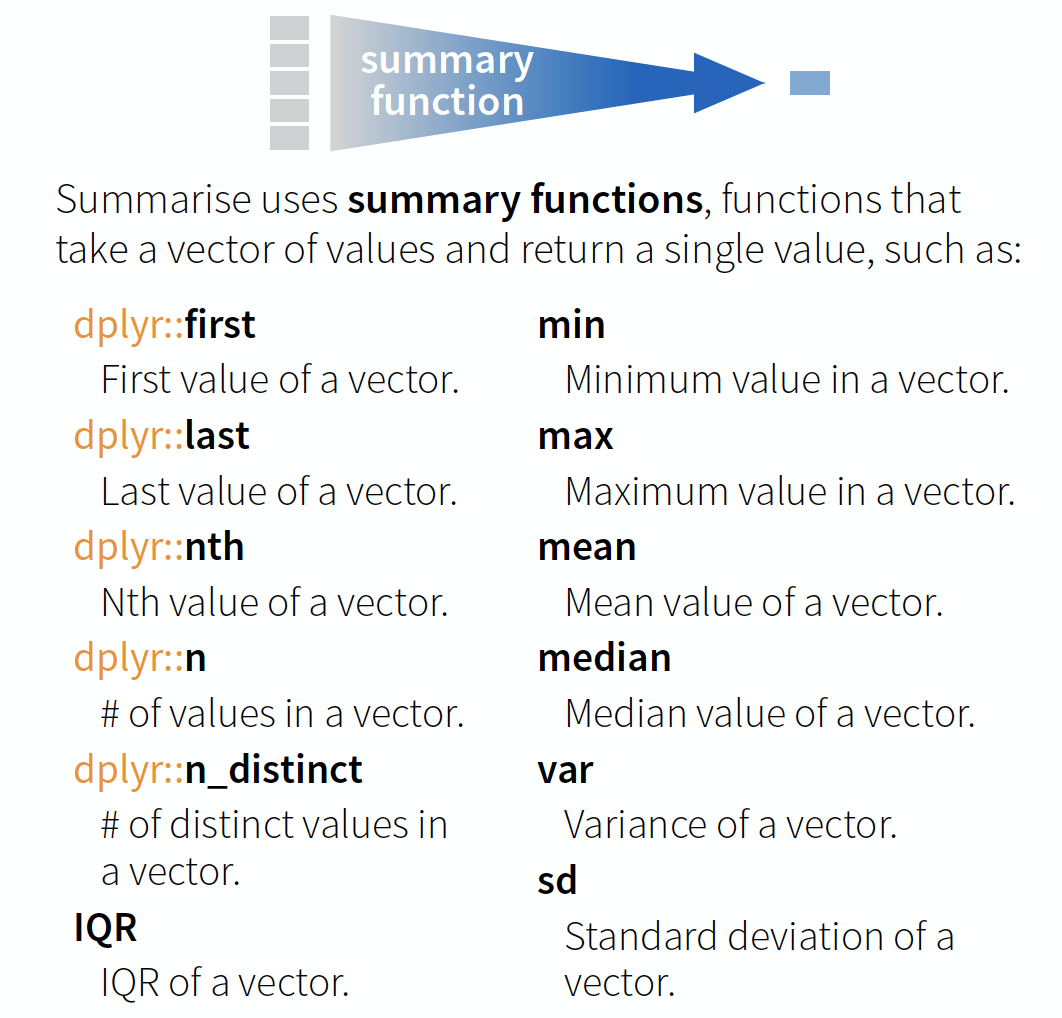
Quelle: ältere Version des sog. cheatsheets
Data Transformation with dplyr (lizensiert unter CC-BY-SA).
Datenaggregation | ‘Shortcuts’
![]()
Es gibt 2 sehr nützliche Funktionen, die man anstelle von summarise() nutzen kann:
count()→ zur Berechnung der Anzahl an Zeilendistinct()→ zum Entfernen von Duplikaten (Zeilen mit komplett gleichem Inhalt in den jeweiligen Spalten)
distinct()
# A tibble: 15 × 2
Species Sepal.Length
<fct> <dbl>
1 setosa 5.1
2 setosa 4.9
3 setosa 4.7
4 setosa 4.6
5 setosa 5
6 setosa 5.4
7 setosa 4.4
8 setosa 4.8
9 setosa 4.3
10 setosa 5.8
11 setosa 5.7
12 setosa 5.2
13 setosa 5.5
14 setosa 4.5
15 setosa 5.3→ Wie Sie sehen, wurden die 50 Zeilen auf 15 reduziert.
Datenaggregation | Beispiel iris 1
Berechnung einer Statistik für mehrere Variablen gleichzeitig:
iris |>
summarise(
SL_mean = mean(Sepal.Length),
SW_mean = mean(Sepal.Width),
PL_mean = mean(Petal.Length),
PW_mean = mean(Petal.Width)
) SL_mean SW_mean PL_mean PW_mean
1 5.843333 3.057333 3.758 1.199333→ Viel Schreibarbeit wenn mehr als nur der Mittelwert berechnet werden soll…
Lösung: Die Helferfunktion across()
Berechnung einer Statistik für mehrere Variablen gleichzeitig:
Lösung: Die Helferfunktion across() | 2
Berechnung mehrerer deskriptiver Statistiken für alle kontinuierlichen Variablen → hier muss eine Liste dem .fns Argument übergeben werden:
iris |>
summarise(
across(
.cols = Sepal.Length:Petal.Width,
.fns = list(mean = mean, median = median, sd = sd)
)
) Sepal.Length_mean Sepal.Length_median Sepal.Length_sd Sepal.Width_mean
1 5.843333 5.8 0.8280661 3.057333
Sepal.Width_median Sepal.Width_sd Petal.Length_mean Petal.Length_median
1 3 0.4358663 3.758 4.35
Petal.Length_sd Petal.Width_mean Petal.Width_median Petal.Width_sd
1 1.765298 1.199333 1.3 0.7622377Aber moment mal…
![]() …was ist, wenn wir die Statistiken für bestimmte Gruppen (wie hier die Arten) getrennt bestimmen wollen????
…was ist, wenn wir die Statistiken für bestimmte Gruppen (wie hier die Arten) getrennt bestimmen wollen????
Lösung: Operationen gruppenweise durchführen
group_by()nimmt eine existierende Tabelle und konvertiert sie in eine gruppierte Tabelle, in der Operationen gruppenweise durchgeführt werden können.ungroup()entfernt Gruppierungen (beisummarise()nicht nötig, aber bei z.B.count()).
Prinzipien der gruppenweisen Operationen
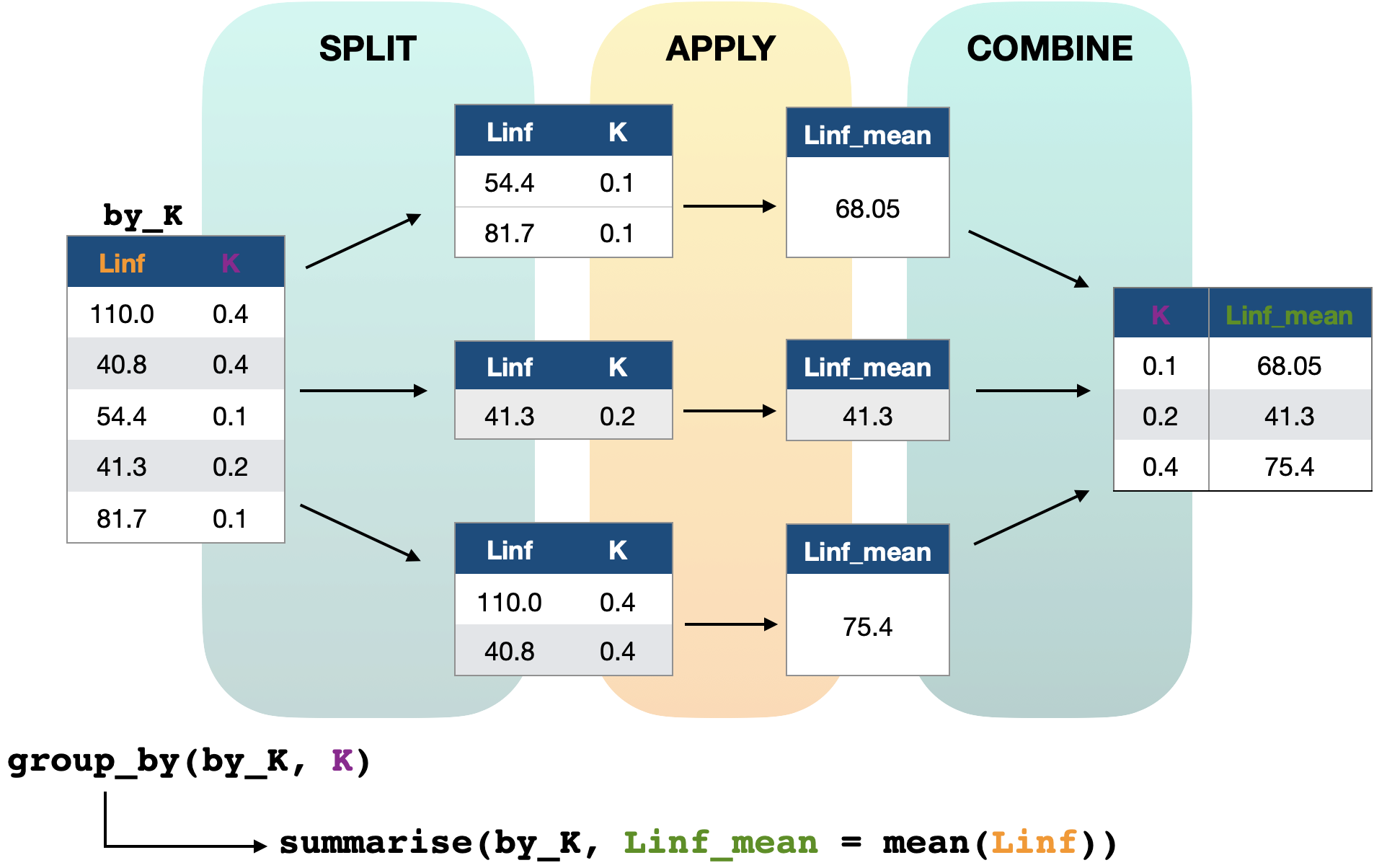
Gruppierte Datenaggregation | iris
iris |>
group_by(Species) |> # einfach diesen Befehl einschieben
summarise(
across(
.cols = Sepal.Length:Petal.Width,
.fns = mean
)
)# A tibble: 3 × 5
Species Sepal.Length Sepal.Width Petal.Length Petal.Width
<fct> <dbl> <dbl> <dbl> <dbl>
1 setosa 5.01 3.43 1.46 0.246
2 versicolor 5.94 2.77 4.26 1.33
3 virginica 6.59 2.97 5.55 2.03 Gruppierung zeitweise entfernen | iris
Kontrollparameter einbauen | iris
- Kontrollieren Sie jeden Zwischenschritt zwei-oder dreimal!
- Überprüfen Sie bei Aggregationen und numerischen Zusammenfassungen immer, ob die richtige Anzahl an Datenpunkten auch eingeflossen ist. Denn dies kann ein Hinweis auf fehlerhaftes Aggregieren sein:
Your turn …
![]()
05:00
Quiz 4-5 | Datenaggregation
![]()
![]()
Q4
Berechnen Sie pro Art: Median, Mittelwert, Standardabweichung und Minimalwert für die Kronblattlänge:
iris |>
group_by(Species) |>
summarise(
pl_median = median(Petal.Length),
pl_mean = mean(Petal.Length),
pl_sd = sd(Petal.Length),
pl_min = min(Petal.Length)
)# A tibble: 3 × 5
Species pl_median pl_mean pl_sd pl_min
<fct> <dbl> <dbl> <dbl> <dbl>
1 setosa 1.5 1.46 0.174 1
2 versicolor 4.35 4.26 0.470 3
3 virginica 5.55 5.55 0.552 4.5Q5
Berechnen Sie die Stichprobengröße (= Zeilenanzahl) pro Ernährungsgruppe im ChickWeight Datensatz:
Rows: 578
Columns: 4
$ weight <dbl> 42, 51, 59, 64, 76, 93, 106, 125, 149, 171, 199, 205, 40, 49, 5…
$ Time <dbl> 0, 2, 4, 6, 8, 10, 12, 14, 16, 18, 20, 21, 0, 2, 4, 6, 8, 10, 1…
$ Chick <ord> 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 2, 2, 2, 2, 2, 2, 2, 2, 2, …
$ Diet <fct> 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, …Transformation: Datensätze kombinieren

Kombination von Tabellen | 1
Zeilenweise nach Position
bind_rows() → hängt 2 oder mehr Tabellen zeilenweise aneinander:

- Spalten werden nach Namen abgeglichen und fehlende Spalten werden mit NA aufgefüllt.
- Weitere Funktionen die zeilenweise verbinden:
intersect(),union(),setdiff()
Kombination von Tabellen | 2
Spaltenweise nach Position
bind_cols() → hängt 2 oder mehr Tabellen spaltenweise aneinander:
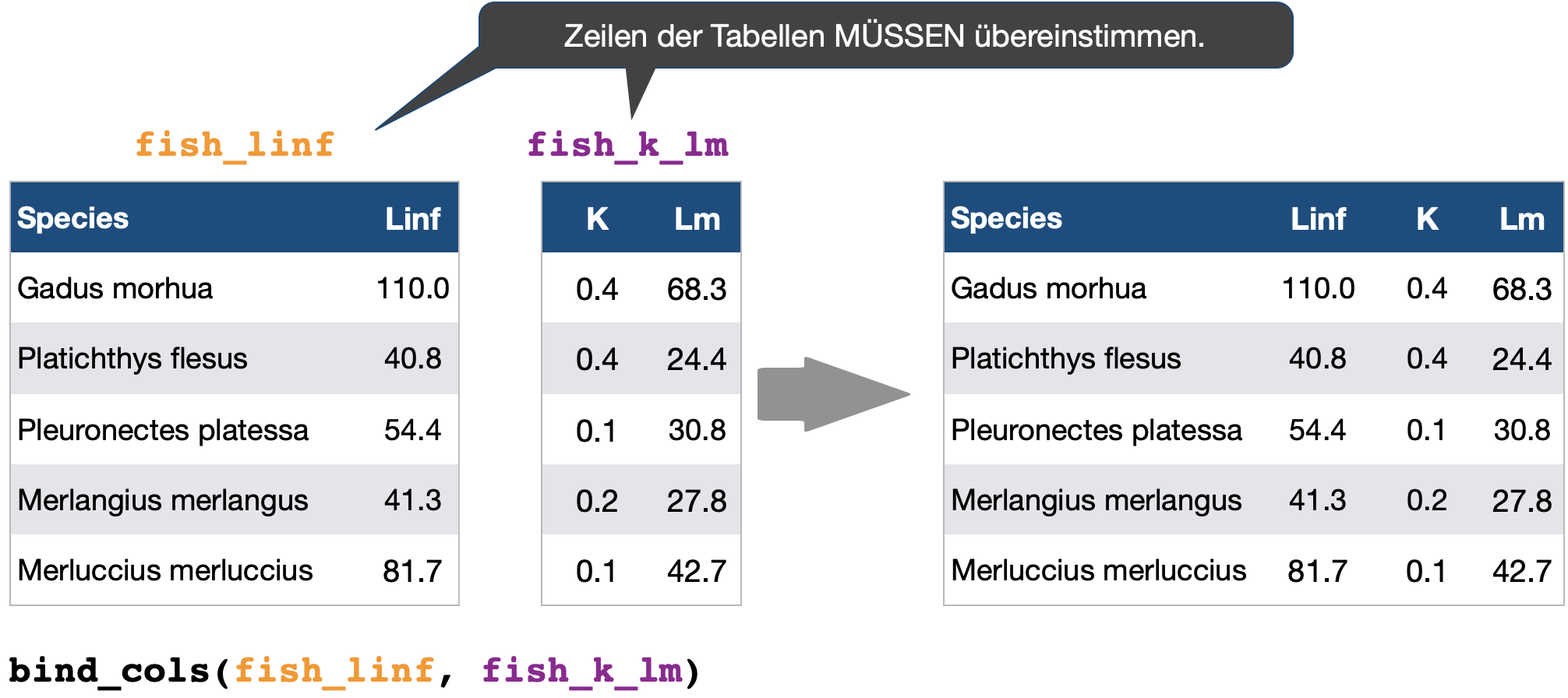
- Zeilen werden hier entsprechend ihrer Position verknüpft → für ein Verknüpfung über die Werte nutzen Sie eine der folgenden
XXX_join()Funktionen!
Kombination von Tabellen | 3
Zusammenfügung über Werte
XXX_join() → Die Funktionsgruppe verbindet Tabellen basierend auf gleichen Werten in übereinstimmenden Spalten:
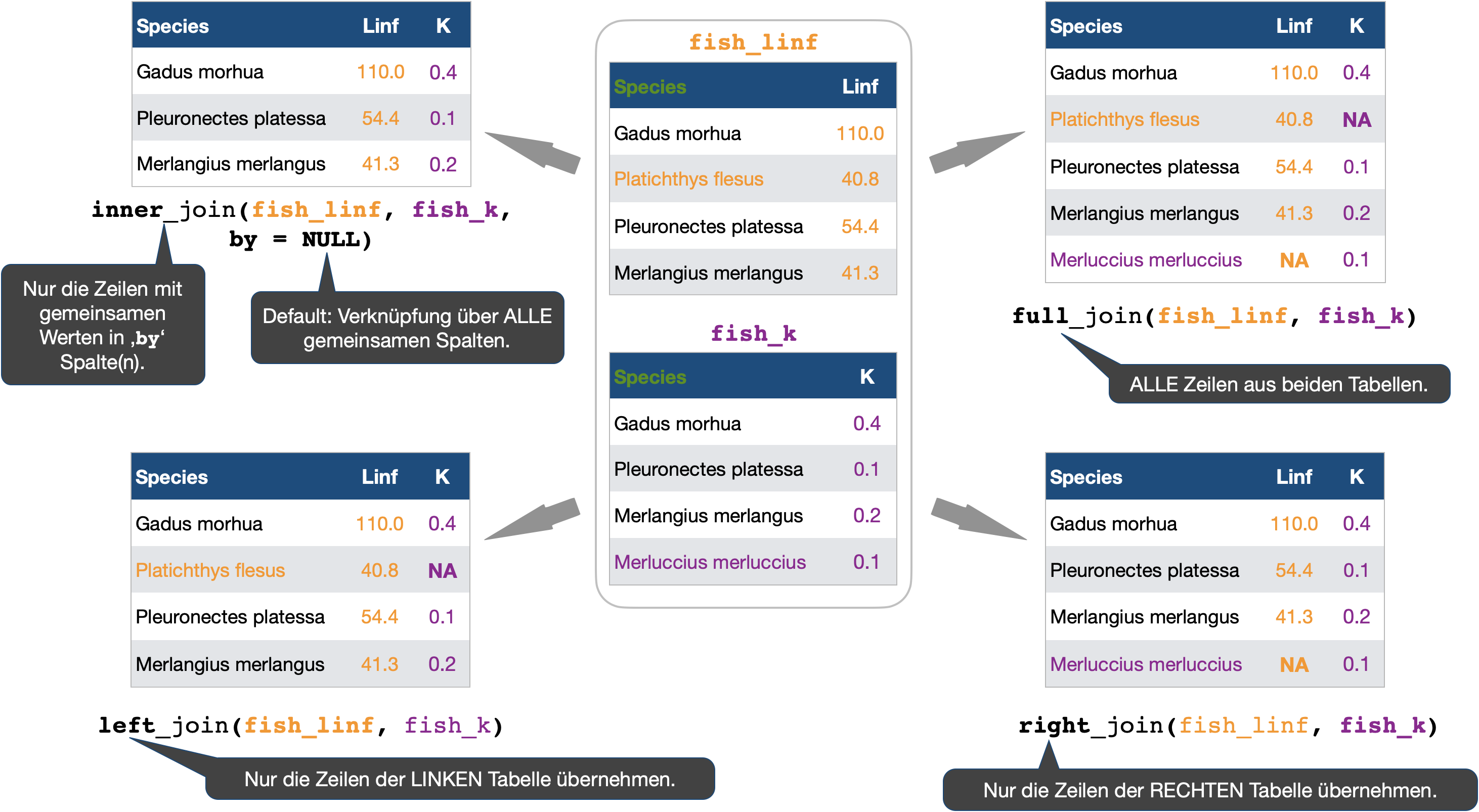
- Je nach übereinstimmenden Tabelleninhalten können dabei nur Spalten, nur Zeilen oder beides aneinandergehängt werden.
Zurück zu Demo B..

Von Calc zu R wechseln | Demo B
Deskriptive Statistik mit iris
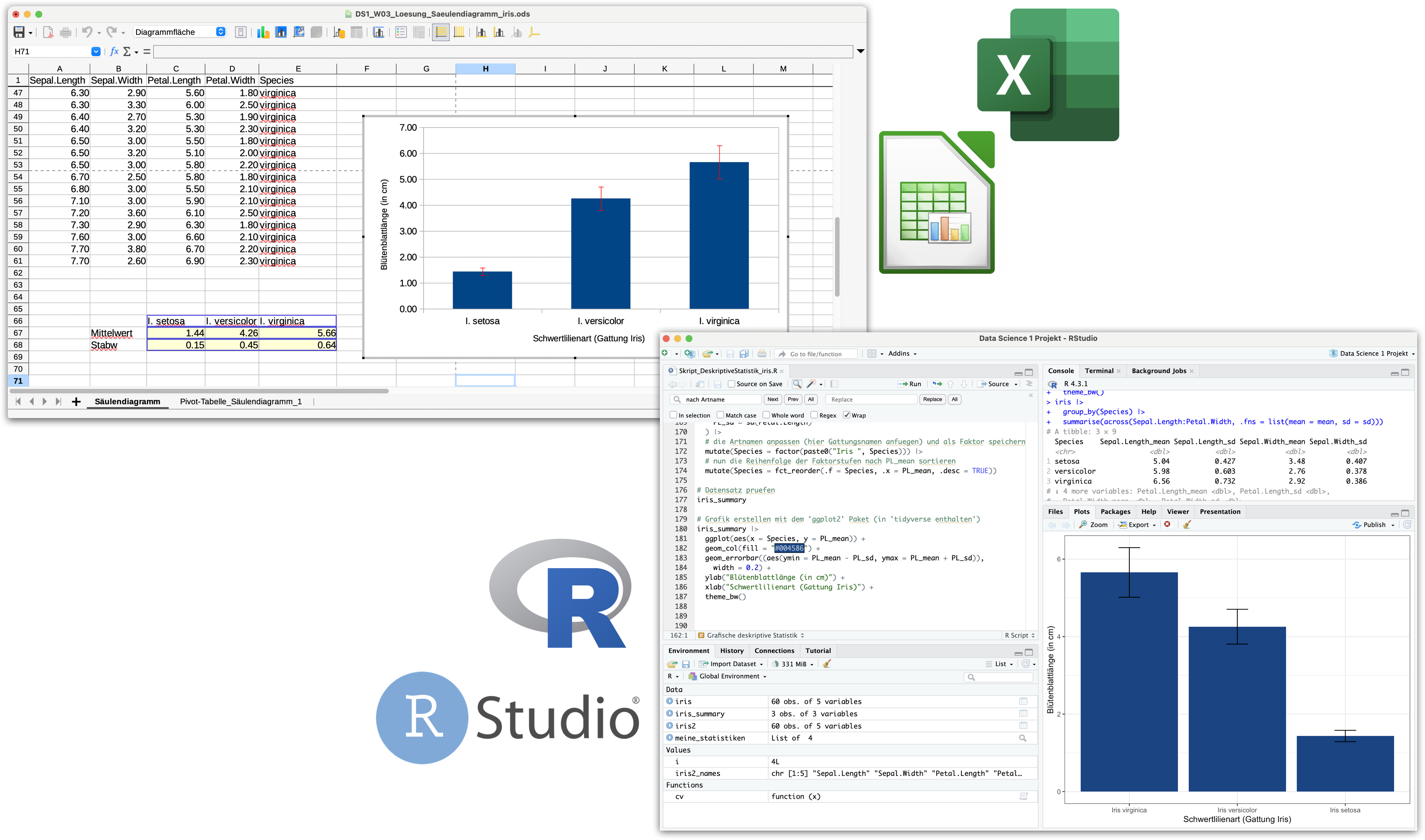
Was haben wir gerade gelernt?
# ------------------------------ Vorbereitung ----------------------------------
#### Laden von Paketen
library(tidyverse) # laedt 9 Pakete
# # (das gleiche wie alle Pakete einzeln zu laden)
# library(dplyr)
# library(forcats)
# library(ggplot2)
# library(lubridate)
# library(purrr)
# library(readr)
# library(stringr)
# library(tibble)
# library(tidyr)
#### Eigene Funktionen
# Variantionskoeffizient
cv <- function(x) {
sd(x)/mean(x)
}
# -------------------- Import und Datenaufbereitung ----------------------------
#### Import von CSV-Dateien
# (-> Swirl-Lektion L01 in DSB-03-Datenaufbereitung oder per Anleitung durchs Tidyversum)
# Import der ODS-Datei, welche in den Zeilen 66-68 noch Text enthaelt
iris <- readODS::read_ods("DS1_W03_Saeulendiagramm_mit_iris.ods")
#### Pruefung des Imports und Datensichtung
# (-> Swirl-Lektion L02 in DSB-03)
# Pruefung des Datentyps --> IMMER DIREKT NACH DEM IMPORT VERWENDEN!
str(iris) # str = structure
# Betrachtung des Inhalts
iris
# View(iris)
# Korrektur des Datentyps und der Zeilen
iris <- iris[1:60, ] |>
mutate(across(Sepal.Length:Petal.Width, as.numeric)) |>
mutate(Species = as.factor(Species))
str(iris)
# Welche Werte kommen in jeder Spalte vor?
lapply(iris, unique)
### Weitere Funktionen zur Sichtung einzelner Aspekte
head(iris) # zeigt erste 6 Zeilen (Kopfzeilen)
tail(iris) # zeigt letzte 6 Zeilen (Endzeilen)
class(iris) # Identifikation der Objektklasse (Vektor, Matrix, dataframe,..)
nrow(iris) # Anzahl Zeilen
ncol(iris) # Anzahl Spalten
dim(iris) # Anzahl aller Dimensionen
names(iris) # Spaltennamen
typeof(iris$Sepal.Length) # Datentyp von Spalte 'Sepal.Length'
typeof(iris$Species) # Datentyp von Spalte 'Species'
# ----------------------- Deskriptive Statistik --------------------------------
#### Berechnung mehrerer Statistiken für jede Spalte im data frame
summary(iris)
#### Berechnung versch. Statistiken der Kronblattlaenge, gruppiert nach Art
# (-> Lektion L01 in DSB-02-Datenexploration mit R)
# (-> Lektion L06-Gruppierte Aggregation in DSB-03)
iris_summary <- iris |>
group_by(Species) |>
summarise(
PL_mean = mean(Petal.Length), # Mittelwert
PL_median = median(Petal.Length), # Median
PL_var = var(Petal.Length), # Varianz
PL_sd = sd(Petal.Length), # Standardabweichung
PL_se = sd(Petal.Length)/sqrt(length(Petal.Length)), # Standardfehler
PL_cv = cv(Petal.Length) # Variationskoeffizient
) |>
# die Artnamen anpassen (hier Gattungsnamen anfuegen) und als Faktor speichern
mutate(Species = factor(paste0("Iris ", Species))) |>
# nun die Reihenfolge der Faktorstufen nach PL_mean sortieren
mutate(Species = fct_reorder(.f = Species, .x = PL_mean, .desc = TRUE))
# Zusammenfassung ansehen
iris_summary
#### Saeulendiagramm erstellen mit dem 'ggplot2' Paket
# (siehe auch swirl-Kurs DSB-04-Datenvisualisierung mit ggplot2)
iris_summary |>
ggplot(aes(x = Species, y = PL_mean)) + # initiert Plot
# die Saeulen hinzufuegen
geom_col(fill = "#004586") +
# die Fehlerbalken hinzufuegen
geom_errorbar((aes(ymin = PL_mean - PL_sd, ymax = PL_mean + PL_sd)),
width = 0.2) +
# Achsenbeschriftung anpassen
ylab("Kronblattlänge (in cm)") +
xlab("Schwertlilienart (Gattung Iris)") +
# das Layout anpassen
theme_bw()DS1_VL04b_Demo_DeskriptiveStatistik_iris.R
Übungsaufgabe

Optionale swirl-Lektionen zur Vertiefung
![]()
Kurs DS1-03-Datenaufbereitung oder per Anleitung durchs Tidyversum
- L05-Transformation mit dplyr: Manipulation von Zeilen und Spalten
- L06-Transformation mit dplyr: Gruppierte Aggregation
- L07-Transformation mit dplyr: Datensätze kombinieren
Übungsskript
![]()
- Vervollständigung eines Lückenskripts zur Erkundung und Aggregation des internen Datensatz
starwars: DS1_W08_Uebungsskript_Datentransformation.R
Fallstudie

Sie sind jetzt so weit, …
- ..dass Sie - falls nötig - ihre einzelnen Fallstudiendateien über eine gemeinsame Spalte mittels einer der
inner_XXX()Funktionen aus dem dplyr Paket zusammenführen könnten. - Sie können anschließend die numerische Statistik der aggregierten Datensätze durchführen.
Wie fühlen Sie sich jetzt…?

Total konfus?

Lesetipps
- Kapitel 5 Data transformation zur Datentransformation und
- Kapitel 18 Pipes zum ‘pipe’ Operator in ‘R for Data Science’
- Posit und DSB Cheatsheets (s. nächste Folien)
Posit Cheatsheet
Überblick an Funktionen im dplyr Paket

Cheatsheet zum tidyr Paket frei verfügbar unter diesem Link.
DSB Cheatsheet: Basic R functions

Enthält wichtigste Funktionen der Datenaufbereitung
Download Link (alternativ auch im Moodle-Kurs)
Total gelangweilt?

Dann testen Sie doch Ihr Wissen in folgendem Abschlussquiz…
Abschlussquiz
![]()

Bei weiteren Fragen: saskia.otto(at)uni-hamburg.de

Diese Arbeit is lizenziert unter einer Creative Commons Attribution-ShareAlike 4.0 International License mit Ausnahme der entliehenen und mit Quellenangabe versehenen Abbildungen.
Kurswebseite: Data Science 1