Datenaufbereitung mit tidyverse:
Import/Export und Bereinigung
Data Science 1 - Programmieren & Visualisieren
Saskia Otto & Monika Eberhard
Universität Hamburg, IMF
Wintersemester 2025/2026
Lernziele
![]()
Am Ende dieser VL- und Übungseinheit werden Sie
- Daten in R importieren und auch exportieren können.
- den Unterschied zwischen dem Export von CSV- oder TXT-Dateien und den sog. Rdata-Dateien kennen.
- die Grundprinzipien der Datenaufbereitung kennen.
- wissen, wie die Datenprüfung und -bereinigung in R mithilfe einiger Grund- und ‘tidyverse’-Funktionen erfolgt.
- einen weiteren Objekttypen kennengelernt haben: den sog. ‘tibble’
Was können Sie bis jetzt?

Aber wie kommen wir …
… von hier…
nach hier?

Die Datenaufbereitung
Die Datenaufbereitung | Was ist das?
- bezeichnet den Prozess der Bereinigung, Umwandlung und Zuordnung von Daten von einer “rohen” Datenform in ein anderes Format mit der Absicht, sie für Analysen geeigneter und wertvoller zu machen.
- ist eine wichtige Voraussetzung, um die Qualität der Daten zu gewährleisten und einen Zusammenhang zwischen den Daten herstellen zu können.
- Datenanalysten verbringen einen großen Teil ihrer Zeit mit der Datenaufbereitung im Vergleich zur eigentlichen Analyse der Daten.
- Idealerweise werden Daten in R so geladen und umgeformt, dass damit [leicht die anschließende Visualisierung und Modellierung erfolgen kann.
- Im Englischen spricht man auch von ‘Data Wrangling’ oder ’Data Munging’.
Die Datenaufbereitung | Komponenten
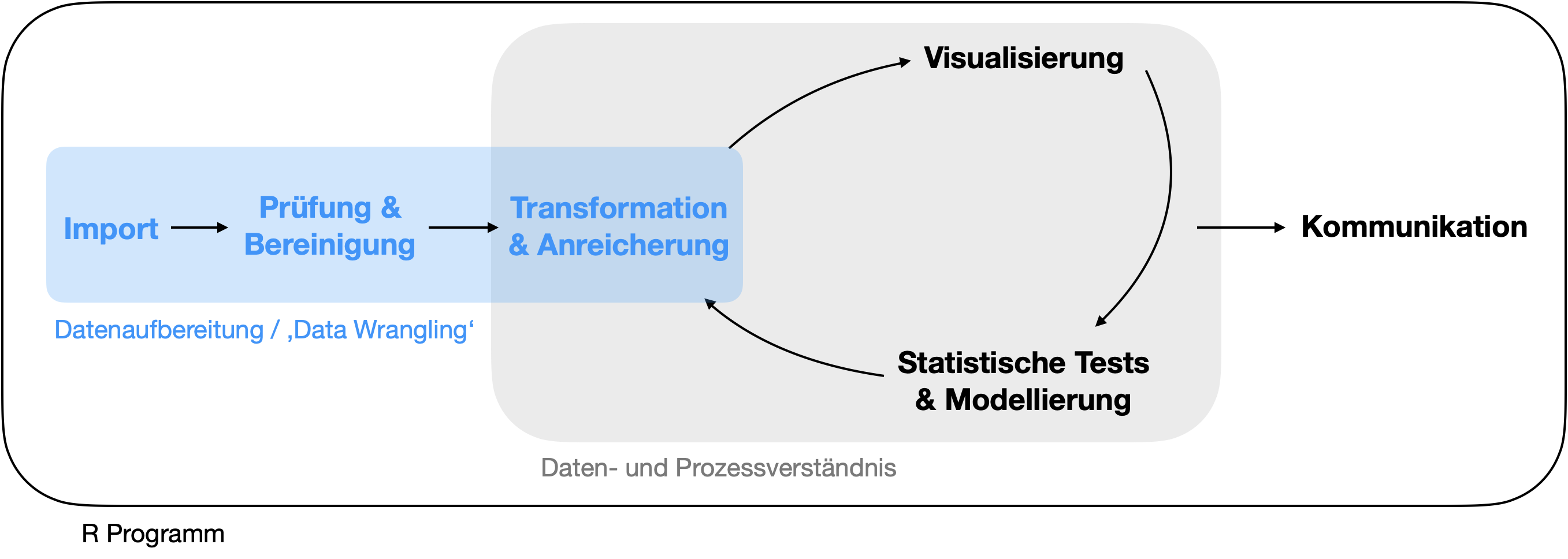
Modifiziert von: R for Data Science von Wickam & Grolemund, 2017 (lizensiert unter CC-BY-NC-ND 3.0 US).
Komponenten im Detail
Import
- Extraktion der Daten in Rohform aus der Datenquelle.
- ‚Parsen‘ der Daten in vordefinierte Datenstrukturen (in R meist data frames).
Prüfung/Bereinigung
- Berichtigung von Informationen.
- Entfernung von irrelevanten Daten und Ausreißern.
- Handling fehlender Werte.
- Anpassung der Daten an ein standardisiertes Muster:
- Jede Spalte repräsentiert eine Variable.
- Jede Zeile repräsentiert eine Beobachtung.
- Maskierung privater oder sensibler Dateneinträge.
Transformation
- Hinzufügen neuer Variablen.
- Bildung von Teildatensätzen.
- Kombination von Datensätzen zur Anreicherung und Verbindung zusammenhängender Informationen.
- Berechnung zusammenfassender Statistiken.
Der Import (und Export)

Datenquellen
- Komma-separierte Werte (.csv)
- Textdateien (.txt)
- Excel Dateien (.xls / .xlsx)
- Calc Dateien (.ods)
- NetCDF (Network Common Data Form)
- Relationale Datenbanken (MySQL, PostgreSQL, etc.)
- Webseiten
Note
Meistens werden flache Dateien in R importiert (sprich ohne interne Hierarchien and Beziehungen wie in Datenbanken).
Import aus Calc oder Excel
Basisfunktionen zum Importieren
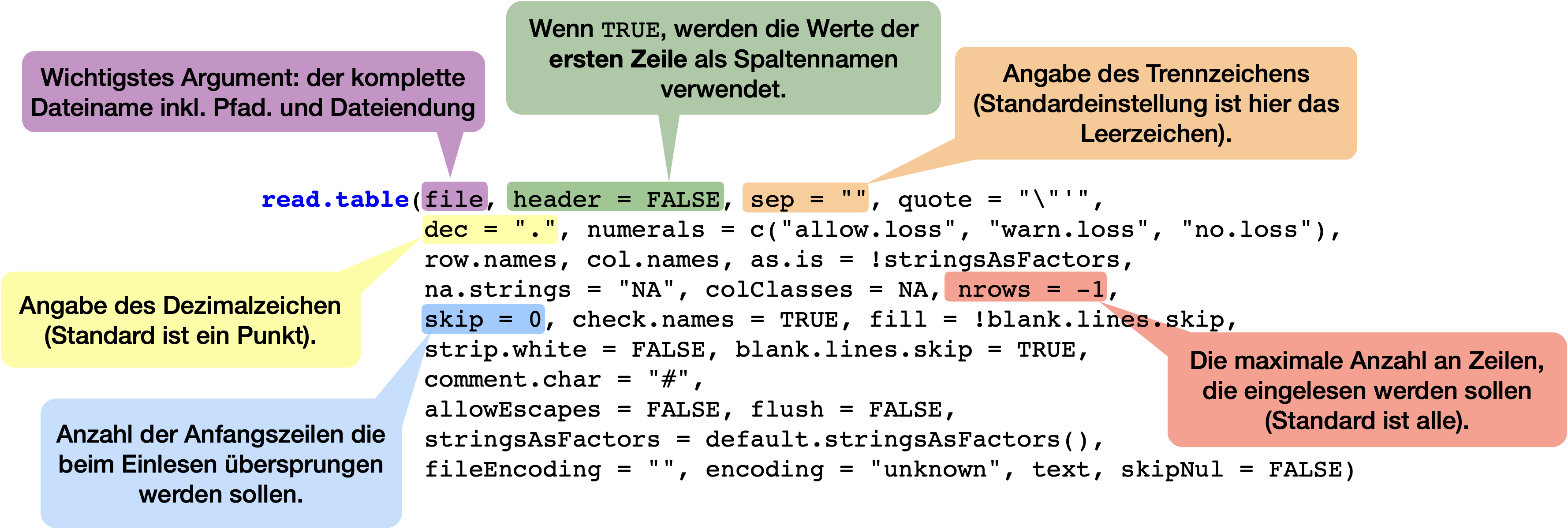
Beispiel read.table()
→ Liest eine Datei im Tabellenformat ein und erstellt daraus einen data frame:
Übersicht der Basis-Importfunktionen
Import zu data frames
| Funktion | Details |
|---|---|
read.table(file, header = FALSE, sep = "", dec = ".") |
Importiert Datei im Tabellenformat (Leerzeichen-separiert) |
read.csv(file, header = TRUE, sep = ",", dec = ".") |
Importiert CSV-Datei (Komma-separiert) |
read.csv2(file, header = TRUE, sep = ";", dec = ",") |
Importiert CSV-Datei (Semikolon-separiert, Komma als Dezimalzeichen) |
read.delim(file, header = TRUE, sep = "\\t", dec = ".") |
Importiert Tab-separierte Textdatei |
read.delim2(file, header = TRUE, sep = "\\t", dec = ",") |
Importiert Tab-separierte Textdatei (mit Komma als Dezimalzeichen) |
Roadmap für den Import

Beispiel | Import LOKALER CSV-Datei
Fütterungsversuch beim Kabeljau
![]()
- Tabellenblatt in der ODS Datei (aus der Calc Übungswoche 3) → als saubere CSV-Datei abgespeichert.
- Jetzt Import mit
read.csv():
- Überprüfung des Imports mit
str(),head()/tail(),View()undsummary():
'data.frame': 11 obs. of 2 variables:
$ Verzehr : num 1868 140 256 719 1714 ...
$ Wachstum: num 482.2 -90.5 -40.3 236 512.7 ... Verzehr Wachstum
1 1868.0 482.2
2 140.3 -90.5
3 255.6 -40.3
4 718.8 236.0
5 1714.4 512.7
6 1149.1 296.6# View(kabeljau) # kompletter Inhalt
summary(kabeljau) # deskriptive Statistik -> komische Extremwerte? Verzehr Wachstum
Min. : 140.3 Min. :-90.50
1st Qu.: 323.8 1st Qu.:-15.05
Median : 718.8 Median :198.00
Mean : 856.2 Mean :180.02
3rd Qu.:1323.5 3rd Qu.:349.80
Max. :1868.0 Max. :512.70 Beispiel | Import ENTFERNTER CSV-Datei
Digitalisiertes Fangprotokoll
# Angabe der URL im Dateinamen
fang <- read.csv(file = "https://saskiaotto.github.io/uham-bio-data-science-1/Fangprotokoll.csv")
str(fang)'data.frame': 8 obs. of 22 variables:
$ Schiff : chr "Alkor" "Alkor" "Alkor" "Alkor" ...
$ Reise : int 551 551 551 551 551 551 551 551
$ Station : chr "43-3" "43-3" "43-3" "43-3" ...
$ Hol : int 9 9 9 9 9 9 9 9
$ Datum : chr "10.03.2021" "10.03.2021" "10.03.2021" "10.03.2021" ...
$ Startzeit : chr "11:36:00" "11:36:00" "11:36:00" "11:36:00" ...
$ Endzeit : chr "12:21:00" "12:21:00" "12:21:00" "12:21:00" ...
$ Schleppdauer : int 45 45 45 45 45 45 45 45
$ Fangbeginn_Breite : num 54.5 54.5 54.5 54.5 54.5 ...
$ Fangbeginn_Laenge : num 11.3 11.3 11.3 11.3 11.3 ...
$ Hieven_Breite : num 54.5 54.5 54.5 54.5 54.5 ...
$ Hieven_Laenge : num 11.4 11.4 11.4 11.4 11.4 ...
$ Fanggeraet : chr "JFT" "JFT" "JFT" "JFT" ...
$ Wassertiefe : int 30 30 30 30 30 30 30 30
$ Fangtiefe : num 26.5 26.5 26.5 26.5 26.5 26.5 26.5 26.5
$ Netzoeffnung : num 3.5 3.5 3.5 3.5 3.5 3.5 3.5 3.5
$ Grundtau_Boden : int 0 0 0 0 0 0 0 0
$ Schleppgeschw : num 2.6 2.6 2.6 2.6 2.6 2.6 2.6 2.6
$ Globalstrahlung : int 380 380 380 380 380 380 380 380
$ Gesamtgewicht_Hol_kg: num 61.5 61.5 61.5 61.5 61.5 ...
$ Art : chr "Hering" "Sprotte" "Dorsch" "Wittling" ...
$ Gesamtzahl : int 50 6565 3 2 1 149 49 1Andere Datentypen
Für den Import weiterer Datentypen können folgende Pakete nützlich sein:
Export von Daten
Wenn Sie Ihre Daten exportieren wollen, um sie dann mit anderen Programmen zu bearbeiten, verwenden Sie am besten das gleiche Format wie beim Import.
Die meisten Importfunktionen haben ein Äquivalent zum Exportieren:
write.table(),write.csv(),write.csv2()
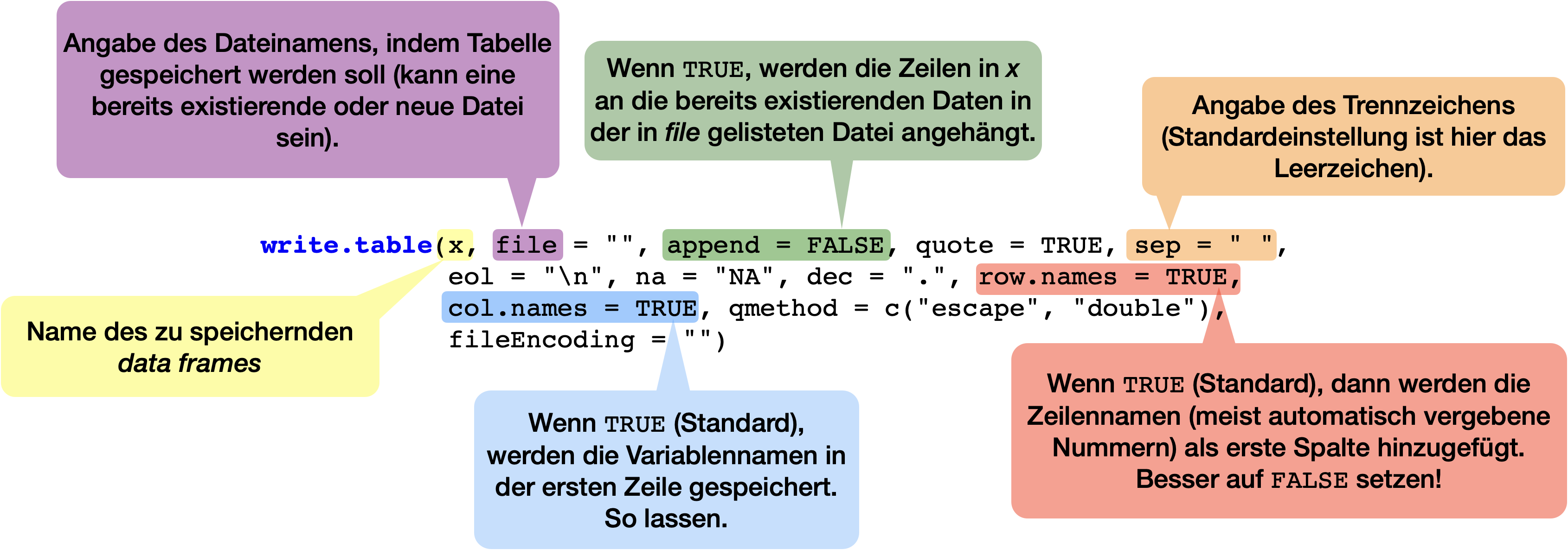
Beispiel | Export
Fütterungsversuch beim Kabeljau
Verzehr Wachstum Ratio
1 1868.0 482.2 0.2581370
2 140.3 -90.5 -0.6450463
3 255.6 -40.3 -0.1576682Nach Veränderung → im gleichen CSV-Format abspeichern und später in z.B. Calc öffnen:
Daten als R Objekte speichern und laden
‘data frame(s)’ können als .R oder .Rdata Objekt mit
save(dataframe1, dataframe2,.., file = "filename.Rdata")abgespeichert
- und jederzeit wieder mit
load(file = "filename.Rdata")geladen werden. - Vorteil:
- Es können mehrere Objekte in einer Datei abgespeichert werden.
- Auch Arrays, Listen und Funktionen lassen sich damit speichern!
Zurück zu unseren Demos..

Von Calc zu R wechseln | Demo A
Lineare Regression zum
Fütterungsversuch beim Kabeljau
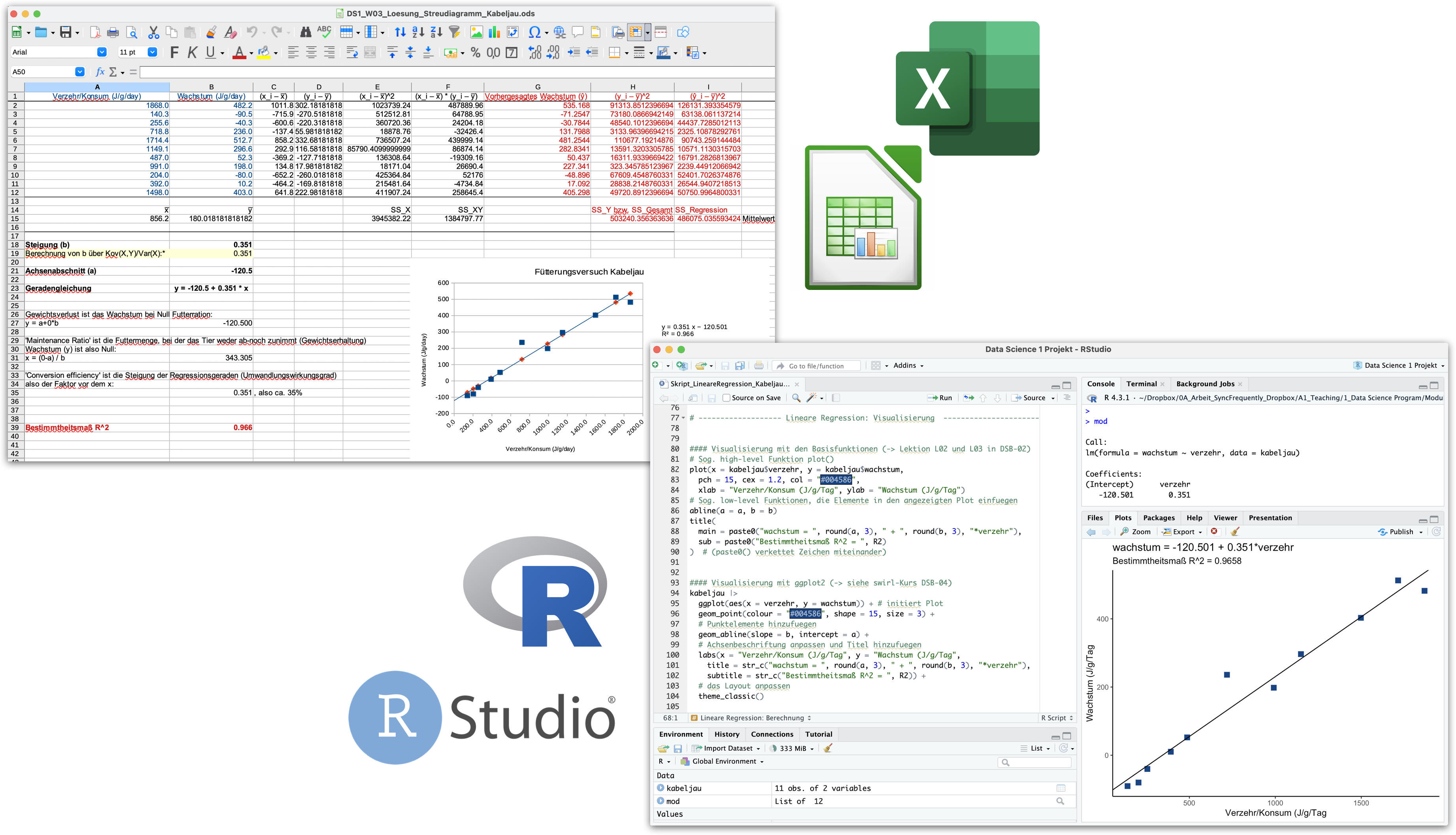
Was haben wir gerade gelernt?
# ------------------------------ Vorbereitung ----------------------------------
#### Laden von Paketen
library(tidyverse)
# library(readODS)
# -------------------- Import und Datenaufbereitung ----------------------------
#### Import einer Textdatei im CSV-Format (das gaengigste Format)
kabeljau_csv <- read.csv(file = "Wachstum-Futter.csv")
str(kabeljau_csv) # --> output ist ein data frame
#### Import von Calc- und Excel-Dateien (ODS- und XLSX-Format)
# Import einer ODS-Datei mit dem 'readODS' Paket
kabeljau <- readODS::read_ods("DS1_W03_Streudiagramm_Kabeljau.ods",
sheet = "Daten_Visualisierung")
str(kabeljau) # --> output ist ein tibble
# Import einer XLSX-Datei mit z.B. dem 'readxl' Paket
kabeljau_xlsx <- readxl::read_excel("DS1_W03_Streudiagramm_Kabeljau.xlsx",
sheet = "Daten_Visualisierung")
str(kabeljau_xlsx) # --> output ist ein tibble
#### Datensichtung und -transformation
# Anpassen der Spaltennamen
names(kabeljau) <- c("verzehr", "wachstum")
str(kabeljau)
# Wertebereich pruefen
summary(kabeljau)
# ---------------------- Lineare Regression: Berechnung ------------------------
# (-> Swirl-Lektion L03 in DSB-02-Datenexploration mit R)
#### Manuelle Berechnung
# Um Tipparbeit zu sparen, speichern wir die Spalten als einzelne Vektoren
x <- kabeljau$verzehr
y <- kabeljau$wachstum
# Steigungsparameter b berechnen
b <- cov(x = x, y = y)/ var(x) # der shortcut mit der Kovarianz und Varianz
b
# Achsenabschnitt a berechnen
a <- mean(y) - b*mean(x)
a
# Das Bestimmtheitsmass R^2 berechnen
y_obs <- a + b*x # die vorhergesagten Werte
ss_gesamt <- sum( (y - mean(y))^2 ) # Summenquadrate Gesamt
ss_regression <- sum( (y_obs - mean(y))^2 ) # Summenquadrate der Regression
R2 <- round(ss_regression/ss_gesamt, 4)
R2
##### Zum Vergleich die Regression automatisch berechnen mit lm()
# Erstellung des Modells
mod <- lm(formula = wachstum ~ verzehr, data = kabeljau)
mod
# Ausgabe nur der beiden Koeffizienten
coef(mod)
# Ausgabe aller wichtigen Statistiken des Modells, inklusive von R^2
# (mehr dazu in Data Science 2)
summary(mod)
# ------------------- Lineare Regression: Visualisierung ----------------------
#### Visualisierung mit den Basisfunktionen (-> Lektion L02 und L03 in DSB-02)
# Sog. high-level Funktion plot()
plot(x = kabeljau$verzehr, y = kabeljau$wachstum,
pch = 15, cex = 1.2, col = "#004586",
xlab = "Verzehr/Konsum (J/g/Tag", ylab = "Wachstum (J/g/Tag")
# Sog. low-level Funktionen, die Elemente in den angezeigten Plot einfuegen
abline(a = a, b = b)
title(
main = paste0("wachstum = ", round(a, 3), " + ", round(b, 3), "*verzehr"),
sub = paste0("Bestimmtheitsmaß R^2 = ", R2)
) # (paste0() verkettet Zeichen miteinander)
#### Visualisierung mit ggplot2 (-> siehe swirl-Kurs DSB-04)
kabeljau |>
ggplot(aes(x = verzehr, y = wachstum)) + # initiert Plot
geom_point(colour = "#004586", shape = 15, size = 3) +
# Punktelemente hinzufuegen
geom_abline(slope = b, intercept = a) +
# Achsenbeschriftung anpassen und Titel hinzufuegen
labs(x = "Verzehr/Konsum (J/g/Tag", y = "Wachstum (J/g/Tag",
title = str_c("wachstum = ", round(a, 3), " + ", round(b, 3), "*verzehr"),
subtitle = str_c("Bestimmtheitsmaß R^2 = ", R2)) +
# das Layout anpassen
theme_bw()DS1_VL04a_Demo_LineareRegression_Kabeljau.R
Von Calc zu R wechseln | Demo B
Deskriptive Statistik mit iris
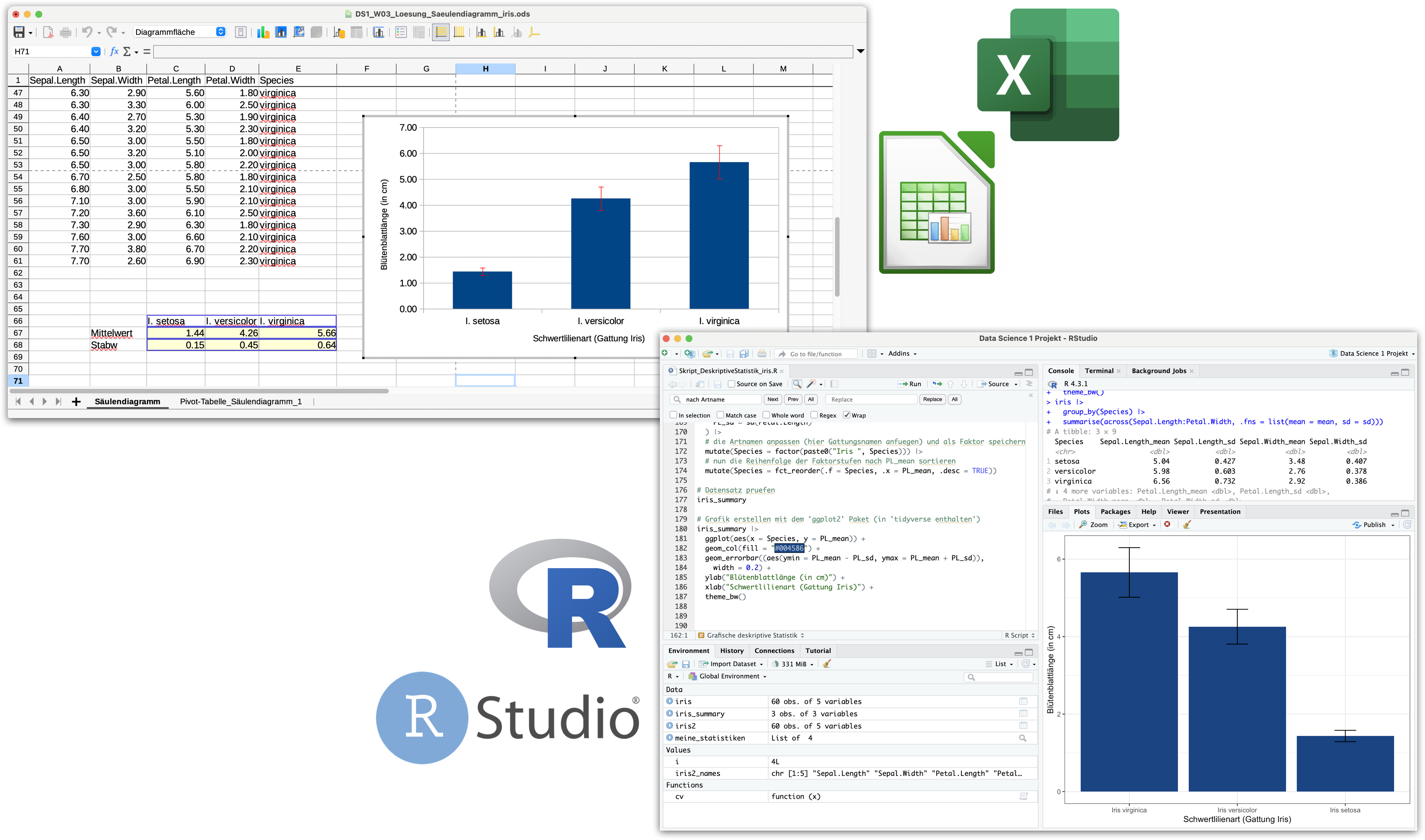
Was haben wir gerade gelernt?
# ------------------------------ Vorbereitung ----------------------------------
#### Laden von Paketen
library(tidyverse) # laedt 9 Pakete
# # (das gleiche wie alle Pakete einzeln zu laden)
# library(dplyr)
# library(forcats)
# library(ggplot2)
# library(lubridate)
# library(purrr)
# library(readr)
# library(stringr)
# library(tibble)
# library(tidyr)
#### Eigene Funktionen
# Variantionskoeffizient
cv <- function(x) {
sd(x)/mean(x)
}
# -------------------- Import und Datenaufbereitung ----------------------------
#### Import von CSV-Dateien
# (-> Swirl-Lektion L01 in DSB-03-Datenaufbereitung oder per Anleitung durchs Tidyversum)
# Import der ODS-Datei, welche in den Zeilen 66-68 noch Text enthaelt
iris <- readODS::read_ods("DS1_W03_Saeulendiagramm_mit_iris.ods")
#### Pruefung des Imports und Datensichtung
# (-> Swirl-Lektion L02 in DSB-03)
# Pruefung des Datentyps --> IMMER DIREKT NACH DEM IMPORT VERWENDEN!
str(iris) # str = structure
# Betrachtung des Inhalts
iris
# View(iris)
# Korrektur des Datentyps und der Zeilen
iris <- iris[1:60, ] |>
mutate(across(Sepal.Length:Petal.Width, as.numeric)) |>
mutate(Species = as.factor(Species))
str(iris)
# Welche Werte kommen in jeder Spalte vor?
lapply(iris, unique)
### Weitere Funktionen zur Sichtung einzelner Aspekte
head(iris) # zeigt erste 6 Zeilen (Kopfzeilen)
tail(iris) # zeigt letzte 6 Zeilen (Endzeilen)
class(iris) # Identifikation der Objektklasse (Vektor, Matrix, dataframe,..)
nrow(iris) # Anzahl Zeilen
ncol(iris) # Anzahl Spalten
dim(iris) # Anzahl aller Dimensionen
names(iris) # Spaltennamen
typeof(iris$Sepal.Length) # Datentyp von Spalte 'Sepal.Length'
typeof(iris$Species) # Datentyp von Spalte 'Species'
# ----------------------- Deskriptive Statistik --------------------------------
#### Berechnung mehrerer Statistiken für jede Spalte im data frame
summary(iris)
#### Berechnung versch. Statistiken der Kronblattlaenge, gruppiert nach Art
# (-> Lektion L01 in DSB-02-Datenexploration mit R)
# (-> Lektion L06-Gruppierte Aggregation in DSB-03)
iris_summary <- iris |>
group_by(Species) |>
summarise(
PL_mean = mean(Petal.Length), # Mittelwert
PL_median = median(Petal.Length), # Median
PL_var = var(Petal.Length), # Varianz
PL_sd = sd(Petal.Length), # Standardabweichung
PL_se = sd(Petal.Length)/sqrt(length(Petal.Length)), # Standardfehler
PL_cv = cv(Petal.Length) # Variationskoeffizient
) |>
# die Artnamen anpassen (hier Gattungsnamen anfuegen) und als Faktor speichern
mutate(Species = factor(paste0("Iris ", Species))) |>
# nun die Reihenfolge der Faktorstufen nach PL_mean sortieren
mutate(Species = fct_reorder(.f = Species, .x = PL_mean, .desc = TRUE))
# Zusammenfassung ansehen
iris_summary
#### Saeulendiagramm erstellen mit dem 'ggplot2' Paket
# (siehe auch swirl-Kurs DSB-04-Datenvisualisierung mit ggplot2)
iris_summary |>
ggplot(aes(x = Species, y = PL_mean)) + # initiert Plot
# die Saeulen hinzufuegen
geom_col(fill = "#004586") +
# die Fehlerbalken hinzufuegen
geom_errorbar((aes(ymin = PL_mean - PL_sd, ymax = PL_mean + PL_sd)),
width = 0.2) +
# Achsenbeschriftung anpassen
ylab("Kronblattlänge (in cm)") +
xlab("Schwertlilienart (Gattung Iris)") +
# das Layout anpassen
theme_bw()DS1_VL04b_Demo_DeskriptiveStatistik_iris.R
So, dann auf zur Datenprüfung und -bereinigung
… aber dazu müssen wir einige neue R Pakete kennenlernen!
‘Tidy (uni)verse’
Ist eine Sammlung von R Paketen, die die gleiche Philosophie teilen und aufeinander abgestimmt sind:
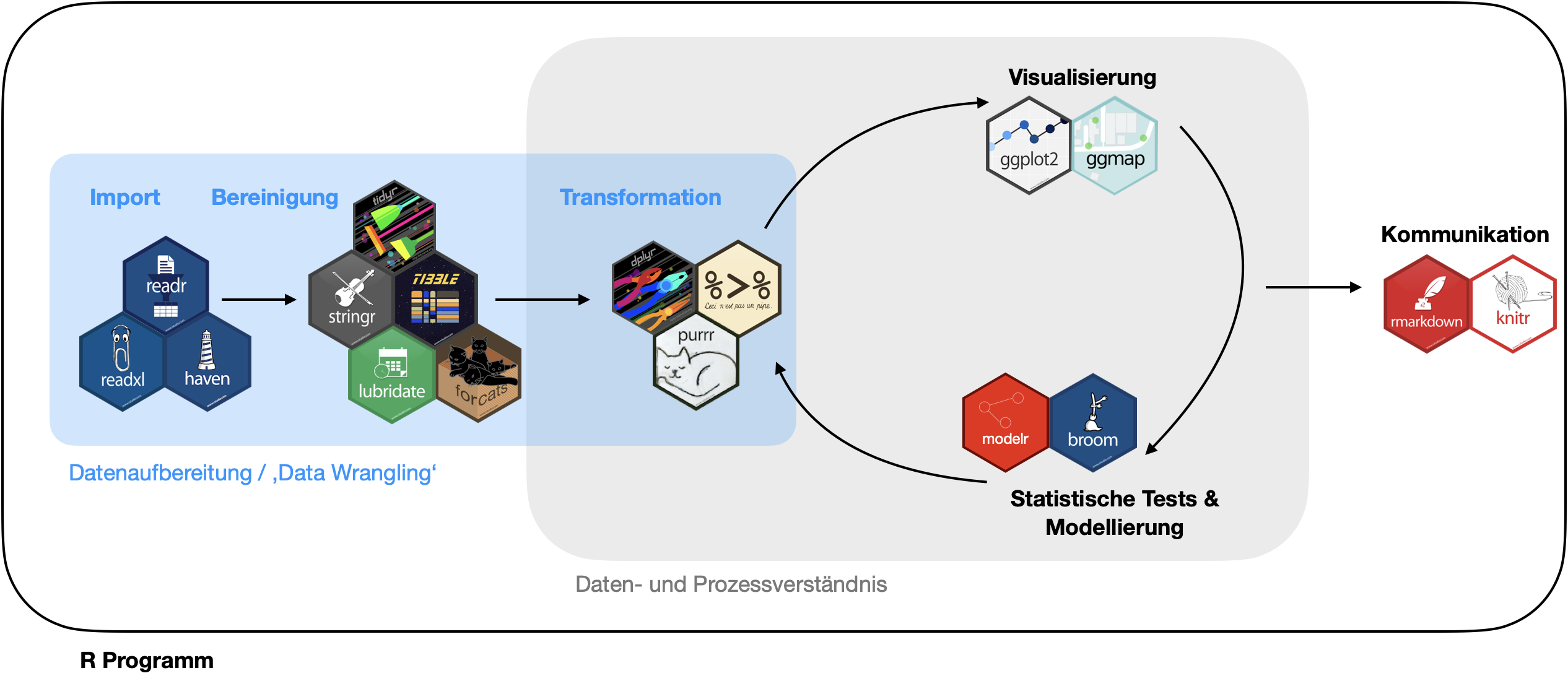
Modifiziert von: R for Data Science von Wickam & Grolemund, 2017 (lizensiert unter CC-BY-NC-ND 3.0 US).
‘Tidy (uni)verse’ | Installation
Am einfachsten ist die Installation (und das spätere Laden) eines einzigen Paketes, welches weitere Pakete installiert und später lädt, mit:
Warum ‘tidyverse’?
- Konsistenz, z.B.
- alle ‘stringr’ Funktionen nehmen eine Zeichenreihenfolge als erstes Argument
- die meisten Funktionen nehmen den ‘data frame’ als erstes Argument (‘piping’)
- Es zwingt zu einer sorgfältigen Vorgehensweise
- Synergien zwischen verschiedenen Paketen und Werkzeugen
- Implementiert einfache Lösungen zu allgemeinen Problemen
- Smartere Voreinstellungen
- z.B.
write.csv(row.names = FALSE),readr::write_csv()
- z.B.
- Funktionen sind oft wesentlich schneller (viele sind mit Rcpp umgesetzt)
- Zunahme an Paketen die das ‘tidyverse’ Konzept implementieren
Moment … ein kurzer Abstecher zu Paketen

Paket laden (JEDE SITZUNG)
- Ein Paket muss bei JEDER Sitzung neu geladen werden, um auf die Funktionen zugreifen zu können.
- Dazu gibt es
library()undrequire(). - R überprüft dann erstmal, ob dieses Paket auch installiert ist.
- Der Unterschied zwischen beiden Funktionen liegt nur in dem was passiert, wenn das Paket nicht vorhanden ist:
Der Suchpfad
![]()
Wenn ein spezifisches Paket geladen wird, wird es als ‘default’ zum Suchpfad hinzugefügt:
Reihenfolge der Suchumgebungen

- Um eine Funktion aufrufen zu können, muss R sie erstmal finden. Dafür sucht R als erstes in der globalen Umgebung.
- Anschließend sucht R im Suchpfad, also der Liste aller geladenen Pakete, und zwar in einer spezifischen Reihenfolge.
- Wenn R mehrere Funktionen mit dem gleichen Namen findet, verwendet es die Funktion des zuletzt geladenen Pakets.
Modifiziert von: Advanced R von H. Wickam, 2014
Suchpfad anzeigen
Sie können sich den Suchpfad explizit anzeigen lassen mit search().
[1] ".GlobalEnv" "tools:rstudio" "package:stats"
[4] "package:graphics" "package:grDevices" "package:utils"
[7] "package:datasets" "package:methods" "Autoloads"
[10] "package:base" Nach dem Laden von tidyverse werden 8 weitere ‘tidyverse’ Pakete mit geladen (es wird dabei auch ein Konflikt mit Funktionsnamen angezeigt - filter() und lag() existieren in 2 Paketen):
── Attaching packages ─────────────────────── tidyverse 1.3.2 ──
✔ ggplot2 3.4.0 ✔ purrr 0.3.5
✔ tibble 3.1.8 ✔ dplyr 1.0.10
✔ tidyr 1.2.1 ✔ stringr 1.4.1
✔ readr 2.1.3 ✔ forcats 0.5.2
── Conflicts ────────────────────────── tidyverse_conflicts() ──
✖ dplyr::filter() masks stats::filter()
✖ dplyr::lag() masks stats::lag()Suchpfad nach dem Laden von ‘tidyverse’
Wir lassen uns den Suchpfad noch mal anzeigen:
[1] ".GlobalEnv" "package:forcats" "package:stringr"
[4] "package:dplyr" "package:purrr" "package:readr"
[7] "package:tidyr" "package:tibble" "package:ggplot2"
[10] "package:tidyverse" "tools:rstudio" "package:stats"
[13] "package:graphics" "package:grDevices" "package:utils"
[16] "package:datasets" "package:methods" "Autoloads"
[19] "package:base" 9 Pakete wurden insgesamt hinzugefügt (direkt nach der globalen Umgebung). Neben dem Paket tidyverse noch die Pakete: forcats, stringr, dplyr, purrr, readr, tidyr, tibble und ggplot2
Wie kann man Pakete aus der Sitzung deaktivieren?
Pakete werden aus dem Suchpfad einfach mit detach() entfernt
Oder indem die Box neben dem Paketnamen im ‘Packages’ Fenster entfernt wird:

Information zu Paketen
- Wenn Sie
?packagename(z.B.?tidyverse) aufrufen, erhalten Sie weitere Informationen zu den Kernaufgaben des Pakets und den verfügbaren Funktionen. Manchmal gibt es auch Weblinks für weitere Infos.
Hilfreiche Vignetten
Your turn …
![]()
00:20
Quiz 1 | Funktionskonflikte
![]()
Datenbereinigung mit ‘tidyr’

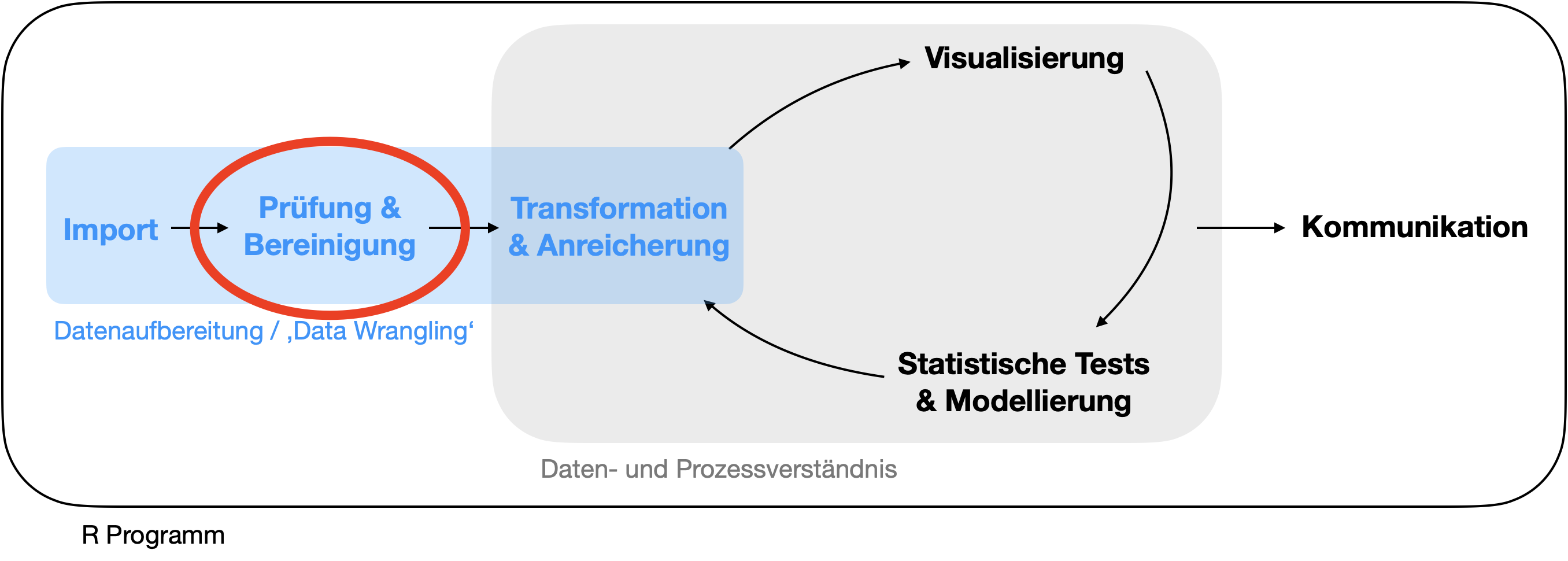
Komponenten im Detail
Import
Datenprüfung/Datenbereinigung
- Berichtigung von Informationen.
- Entfernung von irrelevanten Daten und Ausreißern.
- Handling fehlender Werte.
- Anpassung der Daten an ein standardisiertes Muster:
- Jede Spalte repräsentiert eine Variable.
- Jede Zeile repräsentiert eine Beobachtung.
- Maskierung privater oder sensibler Dateneinträge.
Transformation
Handling fehlender Werte
3 hilfreiche Funktionen für den Umgang mit NAs

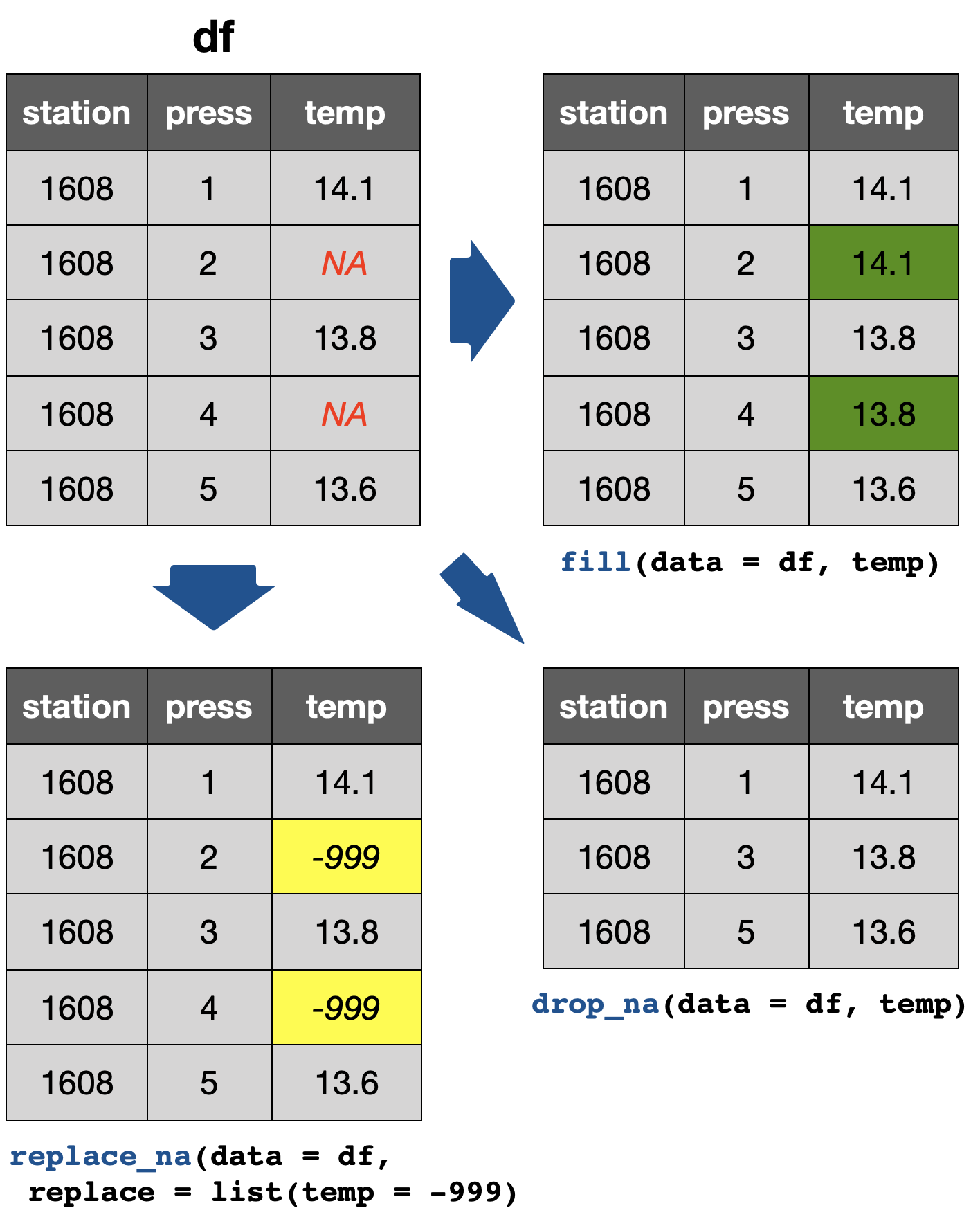
tidyr::drop_na(data, ...): Lässt ganze Reihen, die fehlende Werte enthält, entfallen.tidyr::fill(data, ..., .direction = c("down", "up")): Füllt fehlende Werte mit den vorherigen (direction = “down”) oder nachfolgenden (direction = “up”) Einträgen auf.tidyr::replace_na(data, replace = list(), ...): Ersetzt fehlende Werte mit einem spezifischen Wert für jede Spalte.
Your turn …
![]()
05:00
Quiz 2-4 | Handling fehlender Werte
![]()
![]()
Der Datensatz
Gegeben sind folgende hydrografische Messungen der Ostsee-Station 1321 vom August 2015 :
station lat long depth pres temp psal doxy
1 1321 55 13.3 47 0.2 17.93 8.167 NA
2 1321 55 13.3 47 1.0 NA NA NA
3 1321 55 13.3 47 5.0 NA 8.328 4.60
4 1321 55 13.3 47 10.0 17.49 8.349 4.50
5 1321 55 13.3 47 15.0 17.44 NA 4.50
6 1321 55 13.3 47 20.0 17.04 8.340 4.50
7 1321 55 13.3 47 25.0 NA 8.303 4.60
8 1321 55 13.3 47 30.0 15.56 NA 4.60
9 1321 55 13.3 47 35.0 11.29 7.915 4.78
10 1321 55 13.3 47 40.0 NA 8.417 NA
11 1321 55 13.3 47 45.0 11.19 NA NApres = Druck, temp = Temperatur, psal = Salinität, doxy = gelöster Sauerstoff
2 | Zeilen mit NAs entfernen
3 | NAs ersetzen
pres temp psal doxy
1 0.2 17.93 8.167 4.60
2 1.0 17.93 8.167 4.60
3 5.0 17.93 8.328 4.60
4 10.0 17.49 8.349 4.50
5 15.0 17.44 8.349 4.50
6 20.0 17.04 8.340 4.50
7 25.0 17.04 8.303 4.60
8 30.0 15.56 8.303 4.60
9 35.0 11.29 7.915 4.78
10 40.0 11.29 8.417 4.78
11 45.0 11.19 8.417 4.784 | NAs austauschen
sample1_mod3 <- replace_na(sample1, list(
temp = median(sample1$temp, na.rm = TRUE),
psal = median(sample1$psal, na.rm = TRUE),
doxy = median(sample1$doxy, na.rm = TRUE) )
)
sample1_mod3[ ,5:8] pres temp psal doxy
1 0.2 17.93 8.167 4.60
2 1.0 17.04 8.328 4.60
3 5.0 17.04 8.328 4.60
4 10.0 17.49 8.349 4.50
5 15.0 17.44 8.328 4.50
6 20.0 17.04 8.340 4.50
7 25.0 17.04 8.303 4.60
8 30.0 15.56 8.328 4.60
9 35.0 11.29 7.915 4.78
10 40.0 17.04 8.417 4.60
11 45.0 11.19 8.328 4.60Anpassung der Daten an ein standardisiertes Muster
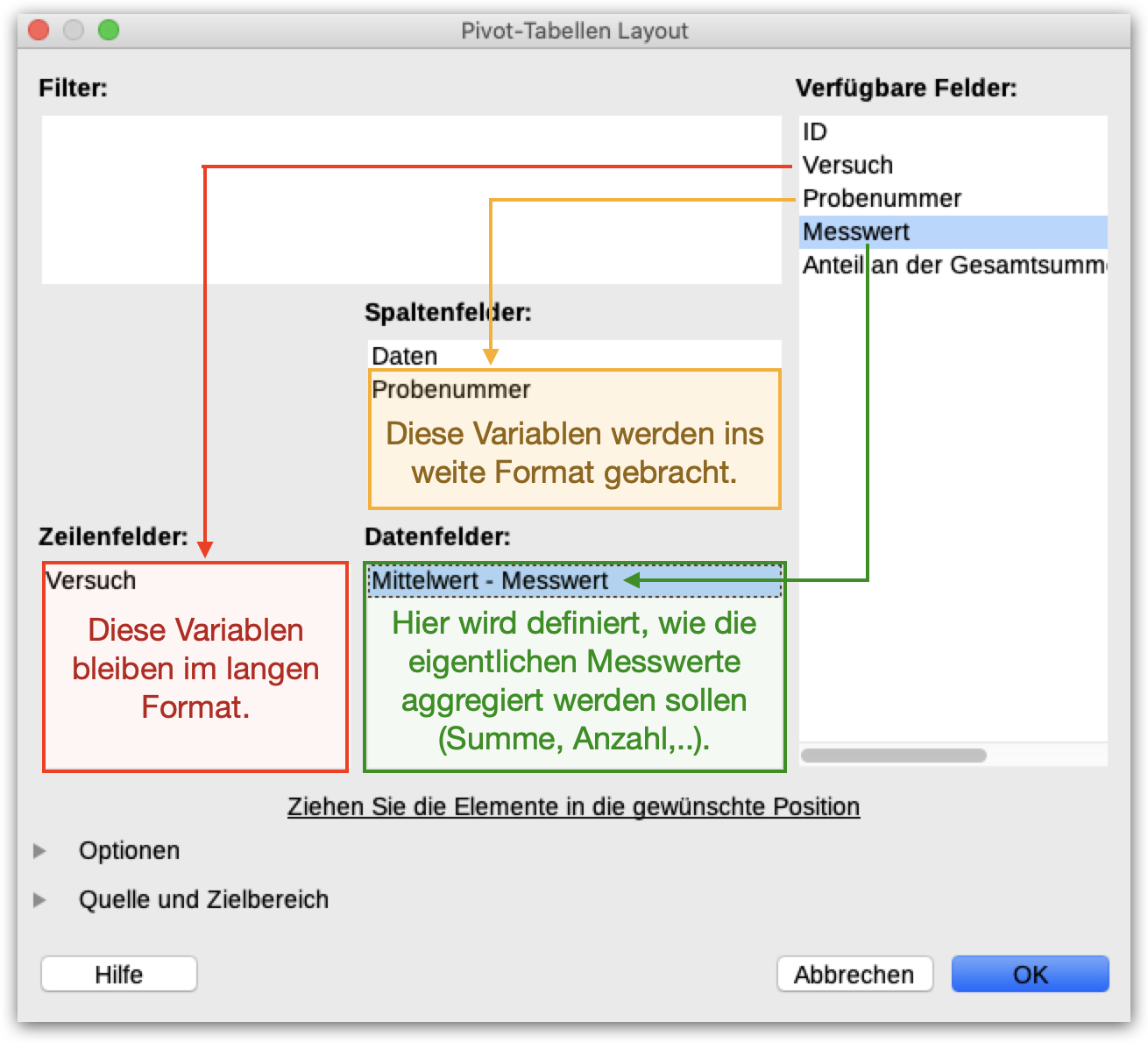
Wechsel zwischen dem langen und weiten Format

Pivot-Tabellenmanager in Calc
Funktionen im R Paket tidyr
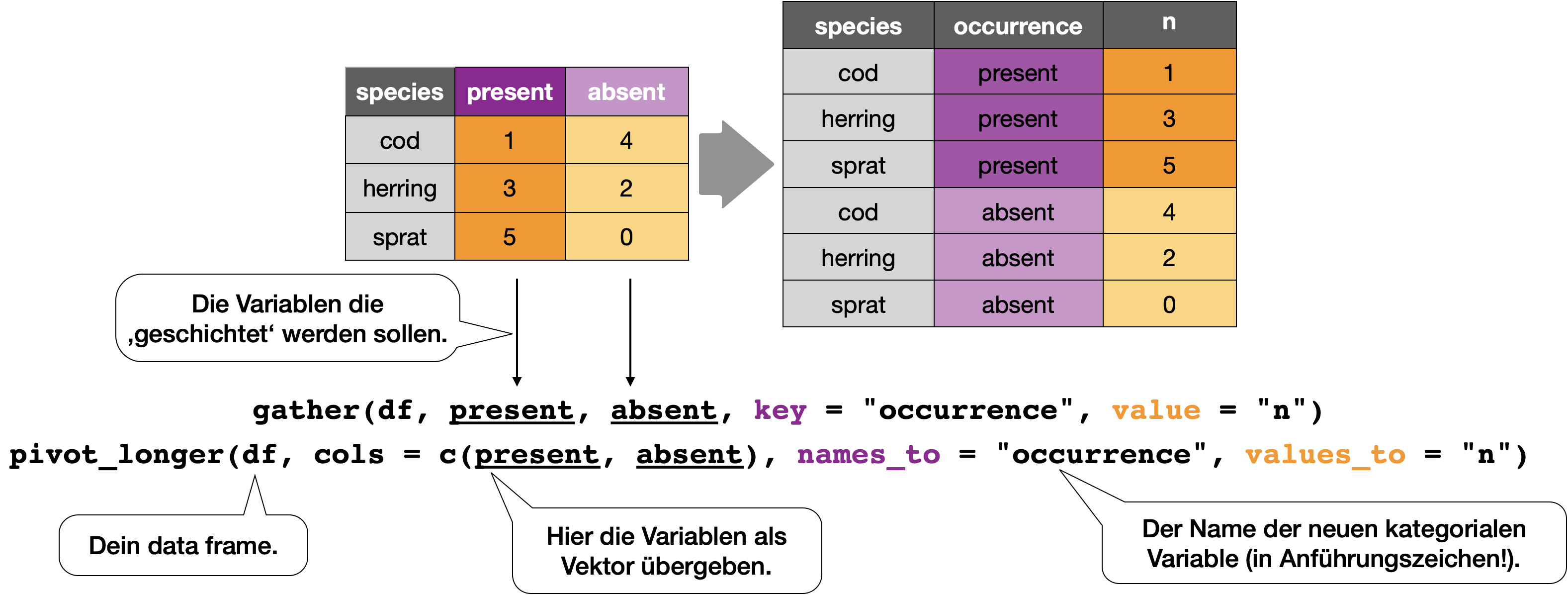
Das ‘tidyr’ Paket bietet zwei Funktionen für den Wechsel des Formats:
- Von weit zu lang:
pivot_longer()(ehemalsgather()) - Von lang zu weit:
pivot_wider()(ehemalsspread())
Wechsel von weitem zum langen Format | 1
pivot_longer() → bewegt Spaltennamen in eine Schlüsselspalte und rafft die Werte der Spalten in eine einzelne Spalte zusammen.
Wechsel von weitem zum langen Format | 2
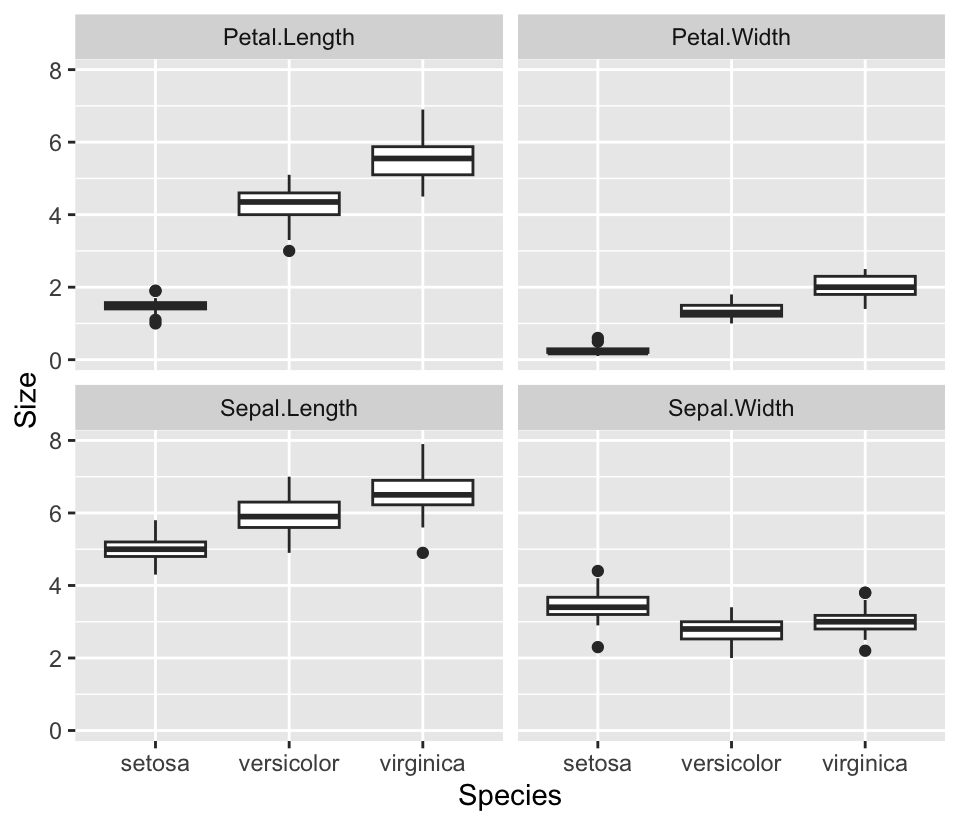
Beispiel iris
Uups, unser Datensatz ist jetzt ein tibble geworden????? ➟ Dazu gleich mehr!
# A tibble: 600 × 3
Species Leaf.Param Size
<fct> <chr> <dbl>
1 setosa Sepal.Length 5.1
2 setosa Sepal.Width 3.5
3 setosa Petal.Length 1.4
4 setosa Petal.Width 0.2
5 setosa Sepal.Length 4.9
6 setosa Sepal.Width 3
7 setosa Petal.Length 1.4
8 setosa Petal.Width 0.2
9 setosa Sepal.Length 4.7
10 setosa Sepal.Width 3.2
11 setosa Petal.Length 1.3
12 setosa Petal.Width 0.2
13 setosa Sepal.Length 4.6
14 setosa Sepal.Width 3.1
15 setosa Petal.Length 1.5
16 setosa Petal.Width 0.2
17 setosa Sepal.Length 5
18 setosa Sepal.Width 3.6
19 setosa Petal.Length 1.4
20 setosa Petal.Width 0.2
21 setosa Sepal.Length 5.4
22 setosa Sepal.Width 3.9
23 setosa Petal.Length 1.7
24 setosa Petal.Width 0.4
25 setosa Sepal.Length 4.6
26 setosa Sepal.Width 3.4
27 setosa Petal.Length 1.4
28 setosa Petal.Width 0.3
29 setosa Sepal.Length 5
30 setosa Sepal.Width 3.4
31 setosa Petal.Length 1.5
32 setosa Petal.Width 0.2
33 setosa Sepal.Length 4.4
34 setosa Sepal.Width 2.9
35 setosa Petal.Length 1.4
36 setosa Petal.Width 0.2
37 setosa Sepal.Length 4.9
38 setosa Sepal.Width 3.1
39 setosa Petal.Length 1.5
40 setosa Petal.Width 0.1
41 setosa Sepal.Length 5.4
42 setosa Sepal.Width 3.7
43 setosa Petal.Length 1.5
44 setosa Petal.Width 0.2
45 setosa Sepal.Length 4.8
46 setosa Sepal.Width 3.4
47 setosa Petal.Length 1.6
48 setosa Petal.Width 0.2
49 setosa Sepal.Length 4.8
50 setosa Sepal.Width 3
51 setosa Petal.Length 1.4
52 setosa Petal.Width 0.1
53 setosa Sepal.Length 4.3
54 setosa Sepal.Width 3
55 setosa Petal.Length 1.1
56 setosa Petal.Width 0.1
57 setosa Sepal.Length 5.8
58 setosa Sepal.Width 4
59 setosa Petal.Length 1.2
60 setosa Petal.Width 0.2
61 setosa Sepal.Length 5.7
62 setosa Sepal.Width 4.4
63 setosa Petal.Length 1.5
64 setosa Petal.Width 0.4
65 setosa Sepal.Length 5.4
66 setosa Sepal.Width 3.9
67 setosa Petal.Length 1.3
68 setosa Petal.Width 0.4
69 setosa Sepal.Length 5.1
70 setosa Sepal.Width 3.5
71 setosa Petal.Length 1.4
72 setosa Petal.Width 0.3
73 setosa Sepal.Length 5.7
74 setosa Sepal.Width 3.8
75 setosa Petal.Length 1.7
76 setosa Petal.Width 0.3
77 setosa Sepal.Length 5.1
78 setosa Sepal.Width 3.8
79 setosa Petal.Length 1.5
80 setosa Petal.Width 0.3
81 setosa Sepal.Length 5.4
82 setosa Sepal.Width 3.4
83 setosa Petal.Length 1.7
84 setosa Petal.Width 0.2
85 setosa Sepal.Length 5.1
86 setosa Sepal.Width 3.7
87 setosa Petal.Length 1.5
88 setosa Petal.Width 0.4
89 setosa Sepal.Length 4.6
90 setosa Sepal.Width 3.6
91 setosa Petal.Length 1
92 setosa Petal.Width 0.2
93 setosa Sepal.Length 5.1
94 setosa Sepal.Width 3.3
95 setosa Petal.Length 1.7
96 setosa Petal.Width 0.5
97 setosa Sepal.Length 4.8
98 setosa Sepal.Width 3.4
99 setosa Petal.Length 1.9
100 setosa Petal.Width 0.2
# ℹ 500 more rowsWechsel vom langem zum weiten Format
pivot_wider() → bewegt einzigartige Werte der Schlüsselspalte in die Spaltennamen und verbreitet die Werte der Spalte auf neue Spalten.
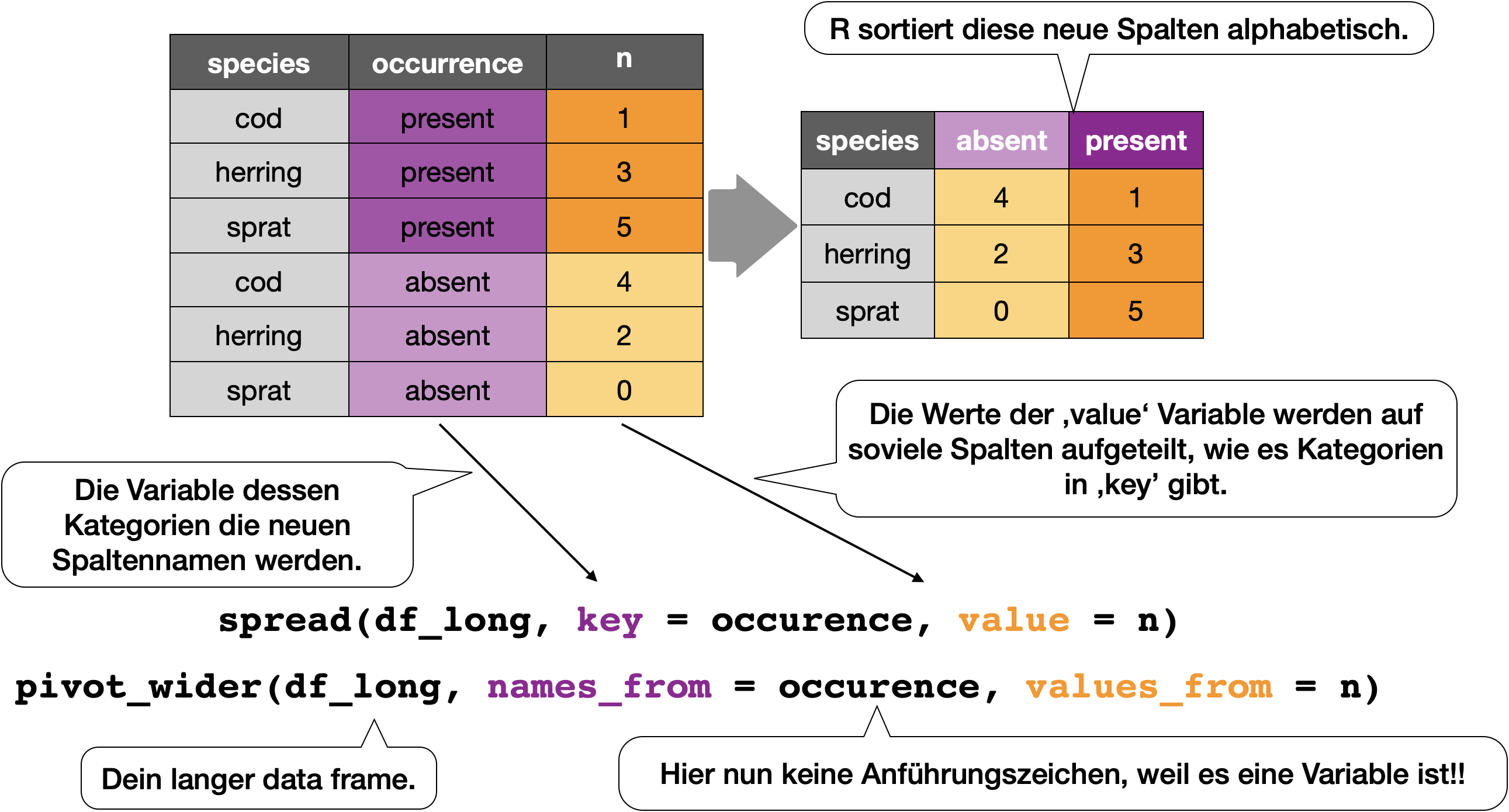
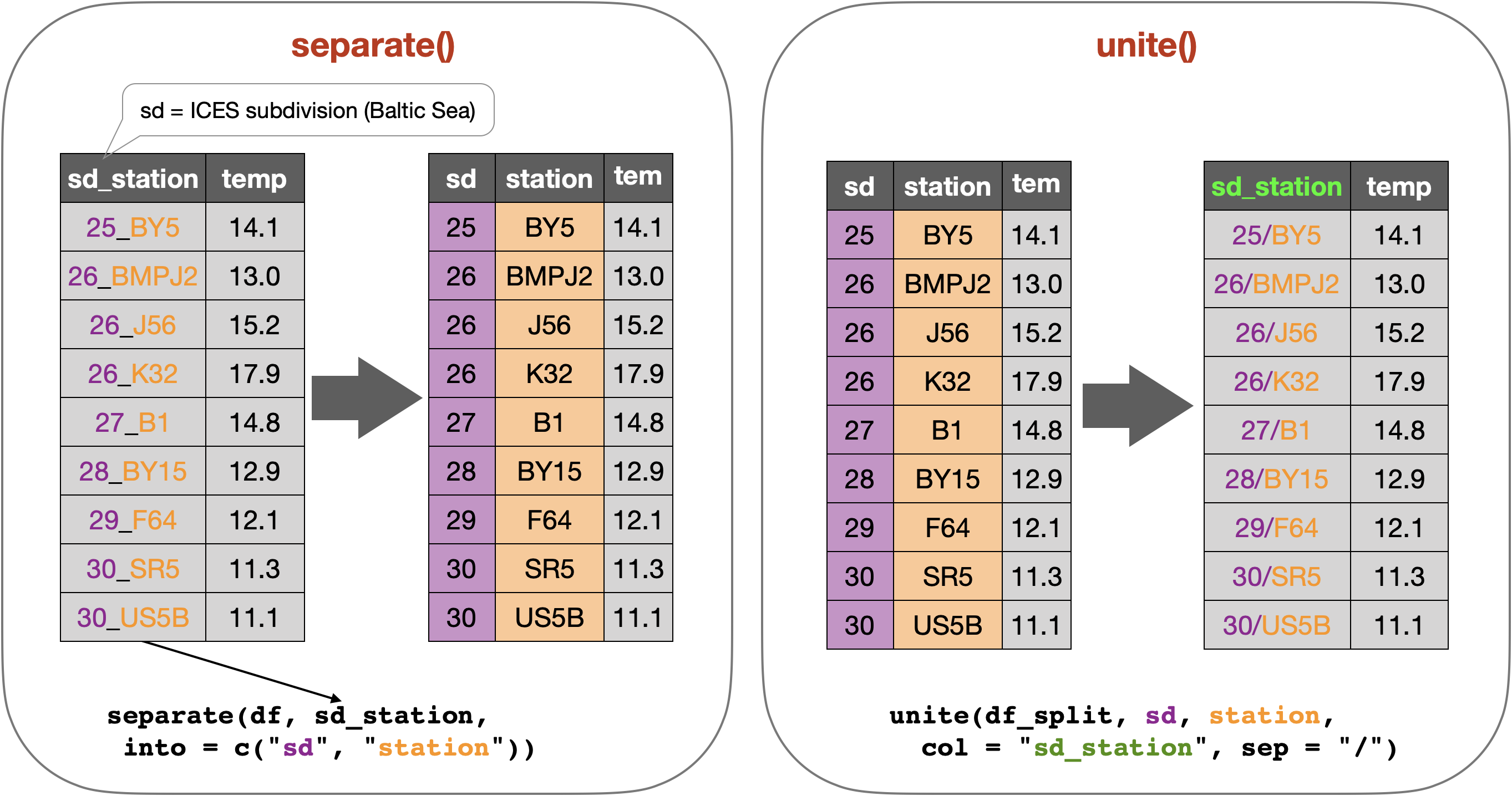
Trennen und verbinden von Spalten bzw. Informationen
Your turn …
![]()
05:00
Quiz 5-7 | Datenformat ändern
![]()
![]()
Ein Vergleich
Warum sind pivot_longer() und pivot_wider() nicht perfekt symmetrisch? Begutachten Sie sorgfältig folgenden Datensatz (hier wieder ein tibble):
# A tibble: 9 × 3
year quarter stock_return
<dbl> <int> <dbl>
1 2014 2 0.97
2 2014 3 1.13
3 2014 4 0.77
4 2015 1 0.61
5 2015 2 0.77
6 2015 3 1
7 2015 4 0.95
8 2016 1 0.79
9 2016 2 1.365 | Von lang zu weit
Vervollständigen Sie den Code, um den Datensatz in folgendes Format zu bringen:
# A tibble: 3 × 5
year `2` `3` `4` `1`
<dbl> <dbl> <dbl> <dbl> <dbl>
1 2014 0.97 1.13 0.77 NA
2 2015 0.77 1 0.95 0.61
3 2016 1.36 NA NA 0.796 | Von weit zu lang
Vervollständigen Sie den Code, um den Datensatz zurück ins ursprüngliche Format zu bringen.
stocks_mod2 <- pivot_longer(
stocks_mod1,
cols = `1`:`2`,
names_to = 'quarter',
values_to = 'stock_return'
)
stocks_mod2# A tibble: 12 × 3
year quarter stock_return
<dbl> <chr> <dbl>
1 2014 1 NA
2 2014 4 0.77
3 2014 3 1.13
4 2014 2 0.97
5 2015 1 0.61
6 2015 4 0.95
7 2015 3 1
8 2015 2 0.77
9 2016 1 0.79
10 2016 4 NA
11 2016 3 NA
12 2016 2 1.367 | Unterschied
Zurück zur Eingangsfrage: Warum sind pivot_longer() und pivot_wider() nicht perfekt symmetrisch?
Quiz 8 | Trennen von Spalten
![]()
# A tibble: 9 × 2
time stock_return
<chr> <dbl>
1 2/2014 0.97
2 3/2014 1.13
3 4/2014 0.77
4 1/2015 0.61
5 2/2015 0.77
6 3/2015 1
7 4/2015 0.95
8 1/2016 0.79
9 2/2016 1.36# A tibble: 9 × 3
quarter year stock_return
<chr> <chr> <dbl>
1 2 2014 0.97
2 3 2014 1.13
3 4 2014 0.77
4 1 2015 0.61
5 2 2015 0.77
6 3 2015 1
7 4 2015 0.95
8 1 2016 0.79
9 2 2016 1.36tibbles

Was sind eigentlich ‘tibbles’?

tibble vs. data frame
- Das ‘tibble’ Paket ermöglicht eine neue ‘tbl_df’ Klasse, welche striktere Qualitätschecks bereitstellt und sich besser formatieren lässt als die traditionellen ‘data frames’.
- Alle Funktionen für ‘data frames’ funktionieren auch bei ‘tibbles’.
- Alle ‘tidyverse’ Pakete generieren automatisch ‘tibbles’.
- Werden automatisch erstellt
- beim Import von Daten mittels
read_XXX()Funktionen des ‘readr’ Pakets. - bei starken Umstrukturierungen von ‘data frames’ mit Funktionen aus einem der ‘tidyverse’ Pakete.
- beim Import von Daten mittels
- Mehr zu ‘tibbles’ gibt es hier:
vignette("tibble").
‘tibbles’ erstellen
Umwandeln
Bestehende ‘data frames’ lassen sich mit tibble::as_tibble(your_dataframe) umwandeln.
[1] "data.frame"[1] "tbl_df" "tbl" "data.frame"Wie Sie sehen, iris_tbl erbt auch die ‘data.frame’ Klasse, hat jetzt aber eine zusätzliche ‘tbl’ bzw. ‘tbl_df’ Klasse.
Drucken von ‘tibbles’
Ein kurzer Überblick
- Es wird immer der Objekttyp und die Dimensionen angezeigt.
- Jede Spalte gibt ihren Datentyp mit an.
- Nur die ersten 10 Zeilen und alle Spalten, die in die Konsole passen, werden angezeigt → einfacheres Handling bei großen Datensätzen.
- Wenn die Anzeige der Zeilen (n) und Spalten (width) geändert werden sollen, nutzen Sie
print()und ändern Sie die Argumente:
Übungsaufgabe

Optionale swirl-Lektionen zur Vertiefung
![]()
Kurs DS1-03-Datenaufbereitung oder per Anleitung durchs Tidyversum
- L01-Import und Export von Daten
- L02-Daten sichten
- L03-Einführung ins Tidyversum
- L04-Datenbereinigung mit tidyr
Übungsskripte
![]()
- Vervollständigung eines Lückenskripts zur Exploration des ‘daphnia’ Datensatzes (bekannt aus Calc Übungswoche 1): DS1_W07_Uebungsskript_Import_daphnia_mLuecken.R
- Import mehrerer Datensätze unterschiedlichen Dateityps: DS1_W07_Uebungsskript_ImportDatenpruefung.R
R Skript vervollständigen
Beispieldatensatz daphnia.txt
![]()
Der folgende Datensatz sollte aus der Übungswoche 1 (Datenimport in Calc) bekannt sein. Ziel war hier, nach dem Import und der Bereinigung die Daten nach der Kontrollgruppe, der Müggelsee-Population und einer Überlebensrate an Tag 14 von null zu filtern.
(Beim folgenden Code-Fenster sind oben rechts 2 Buttons versteckt: Fullscreen und In die Zwischenablage kopieren
\(\rightarrow\) einfach mit der Maus rüber hovern und Button wählen.)
# AUFGABE: Fuellen Sie die Luecken '...' und fuehren Sie den Code in R aus!
# ------------------------------ Vorbereitung ----------------------------------
#### Laden von Paketen
library(tidyverse) # laedt 9 Pakete
#### Eigene Funktionen
# -------------------- Import und Datenaufbereitung ----------------------------
# (analog zu Uebungswoche 1)
#### Import von TXT-Datei
# (-> Lektion L01 in DSB-03-Datenaufbereitung oder per Anleitung durchs Tidyversum)
daphnia <- ...("daphnia.txt", sep = ",", header = TRUE)
#### Datensichtung
# (-> Lektion L02 in DSB-03)
# Betrachtung des Inhalts
daphnia
# View(daphnia)
...(daphnia) # zeigt erste 6 Zeilen (Kopfzeilen)
...(daphnia) # zeigt letzte 6 Zeilen (Endzeilen)
# Identifikation der Objektklasse (Vektor, Matrix, dataframe,..)
...(daphnia)
# Ausgabe der Anzahl an Zeilen und Spalten
...(daphnia) # Anzahl Zeilen
...(daphnia) # Anzahl Spalten
...(daphnia) # Anzahl aller Dimensionen
# Spaltennamen
...(daphnia)
# Datentyp pro Spalte
...(daphnia$round)
...(daphnia$pop.treatment)
# Die gesamte Struktur auf einmal anzeigen lassen
...(daphnia)
# Bei kategorialen Variablen ist es sinnvoll sich alle Gruppen anzeigen zu lassen
unique(daphnia...) # Spalte 'exp_id' auswaehlen
unique(daphnia...) # Spalte 'clone' auswaehlen
unique(daphnia...) # Spalte 'pop.treatment' auswaehlen
# -> man kann diese Funktion auch auf alle Spalten gleichzeitig anwenden:
# mit der map() Funktion aus dem tidyverse Paket 'purrr'
# (bald im Kurs DSB-06-Fortgeschrittene Programmierung)
map(daphnia, ~unique(.))
#### Datentransformation
# (-> siehe Lektion L04-L06 in DSB-03 und L01-L02 in DSB-05-Handling spezieller Datentypen)
# Trennen der Spalte pop.treatment und Umkonvertierung von character zu factor
daphnia <- daphnia |>
...(col = "pop.treatment", into = c("population", "treatment"), sep = ...) |>
mutate(population = as.factor(population), treatment = as.factor(treatment))
# Filtern und Anzahl Zeilen ausgeben
daphnia |>
filter(population == "popM", treatment == "control", survived_t14 == 0) |>
count() # -> bei 2 Zeilen trifft die Bedingung zuFallstudie

Sie sind jetzt so weit, …
..dass Sie externe Daten im CSV-, XLSX- oder ODS-Format in R einlesen und sichten können. Versuchen Sie nun auch Ihre einzelnen Fallstudiendateien zu importieren, zu sichten und numerisch zusammenzufassen!
Wie fühlen Sie sich jetzt…?

Total konfus?

Lesetipps
- Kapitel 11 Data import und
- Kapitel 12 Tidy data in ‘R for Data Science’.
- Posit und DSB Cheatsheets (s. nächste Folien)
Posit Cheatsheet | 1
Überblick an tidyverse Funktionen zum Datenimport

Cheatsheet zum tidyr Paket frei verfügbar unter diesem Link.
Posit Cheatsheet | 2
Überblick an Funktionen im tidyr Paket

Cheatsheet zum tidyr Paket frei verfügbar unter diesem Link.
DSB Cheatsheet: Basic R functions

Enthält wichtigste Funktionen der Datenaufbereitung
Download Link (alternativ auch im Moodle-Kurs)
Total gelangweilt?

Dann testen Sie doch Ihr Wissen in folgendem Abschlussquiz…
Abschlussquiz
![]()

Bei weiteren Fragen: saskia.otto(at)uni-hamburg.de

Diese Arbeit is lizenziert unter einer Creative Commons Attribution-ShareAlike 4.0 International License mit Ausnahme der entliehenen und mit Quellenangabe versehenen Abbildungen.
Kurswebseite: Data Science 1