[[1]]
[1] 1 2 3
[[2]]
[1] "a"
[[3]]
[1] TRUE FALSE TRUE
[[4]]
[1] 2.3 5.9 1.1 4.8 11.0
Grundlagen in R:
Komplexere Objekte
Data Science 1 - Programmieren & Visualisieren
Saskia Otto & Monika Eberhard
Universität Hamburg, IMF
Wintersemester 2025/2026
Lernziele
![]()
Am Ende dieser VL- und Übungseinheit werden Sie
- einen Überblick über alle Objekte in R haben.
- Daten bzw. Elemente in Listen, Matrizen und data frames filtern, auswählen und ändern können.
- deskriptive Statistiken wie den Mittelwert, Median oder die Varianz in R berechnen können.
- sauberen Programmcode schreiben können.
Objekttypen
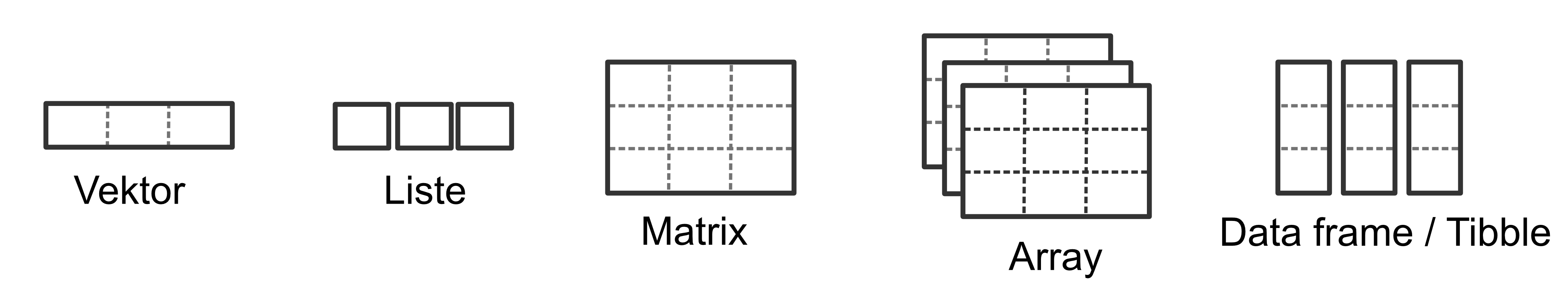
Datenstrukturen in R lassen sich bzgl. ihrer Dimensionalität und der Homogenität bzw. Heterogenität ihrer Datentypen in fünf verschiedene Objekttypen unterscheiden:
| Dimensionen | Homogen | Heterogen |
|---|---|---|
| 1D | (Atomarer) Vektor | Liste |
| 2D | Matrix | ‘Data frame’ (& ‘Tibbles’) |
| >2D | ‘Array’ |
Listen

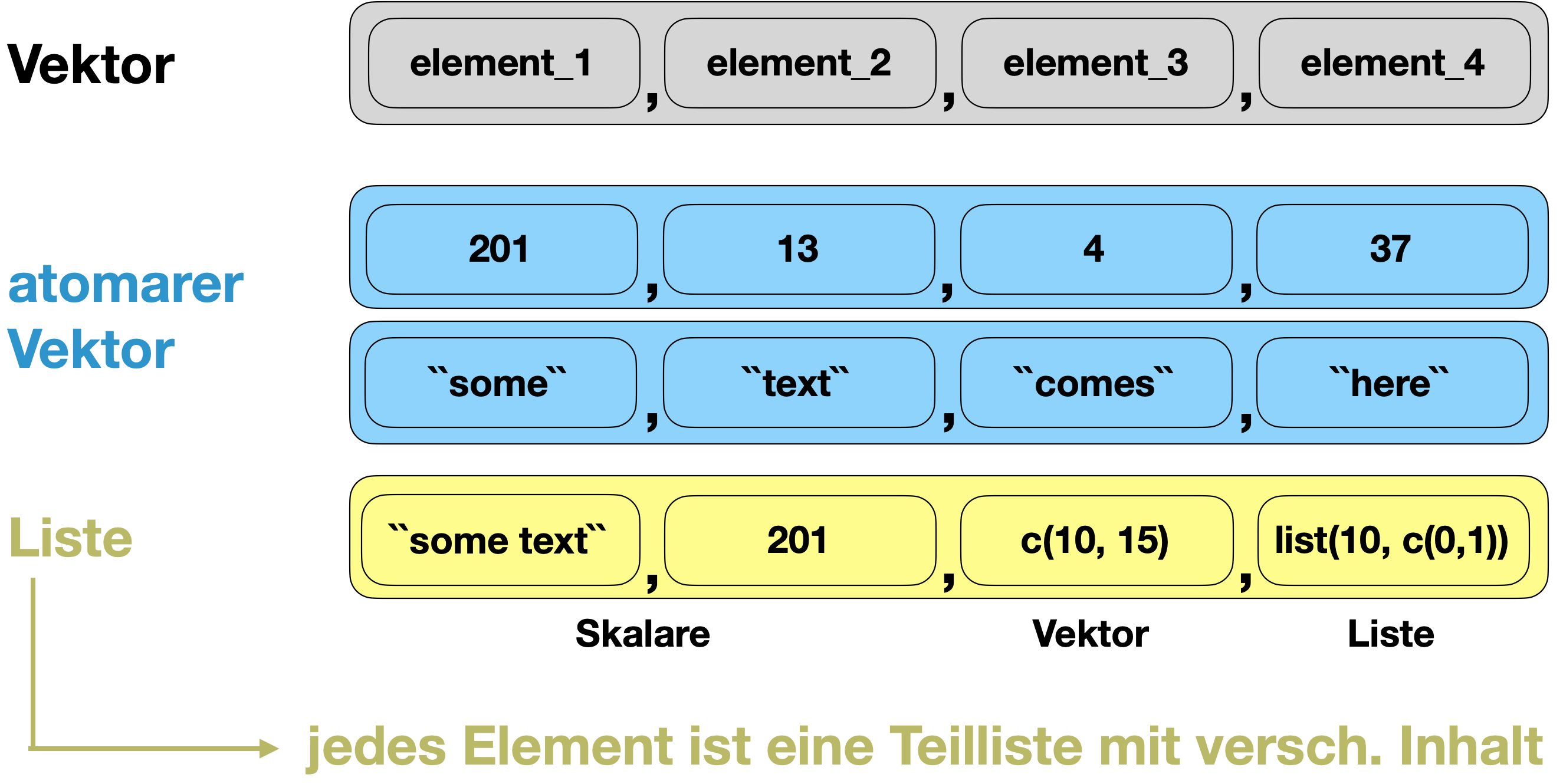
Listen | Erstellen
- Sind anders als atomare Vektoren weil ihre Elemente unterschiedliche Datentypen und auch Datenstrukturen haben dürfen.
- Sie sind besonders praktisch in Schleifen und werden oft als Output von Funktionen verwendet.
- Werden mit
list()erstellt:
Listen zählen auch zu den Vektoren
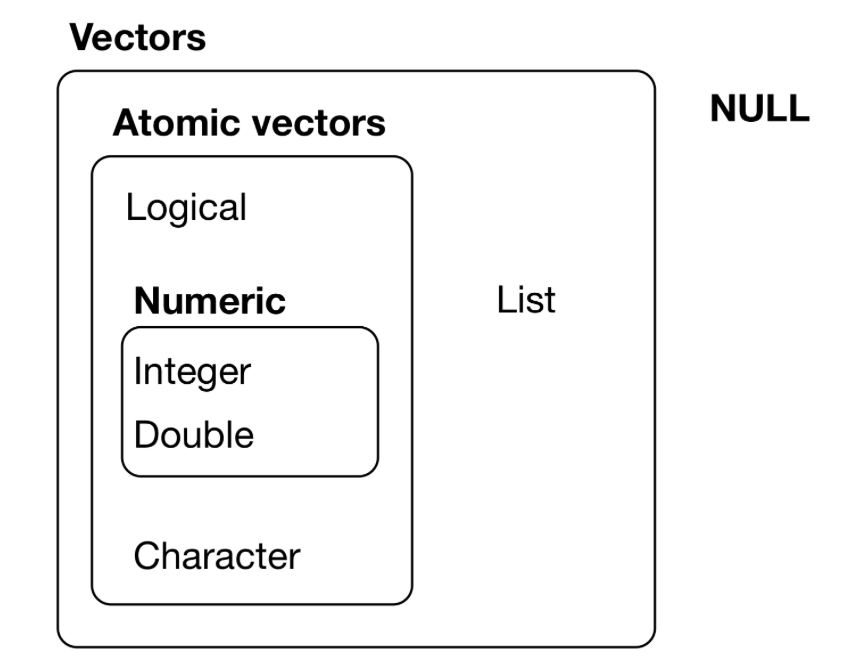
Quelle: R for Data Science von Wickam & Grolemund, 2017 (lizensiert unter CC-BY-NC-ND 3.0 US).
Warum wird eine Liste als Vektor betrachtet?
Listen | Struktur
Eine nützliche Funktion zum Anzeigen der Objektstruktur ist str():
Listen | Rekursion
- Werden manchmal als rekursive Vektoren bezeichnet, weil eine Liste weitere Listen enthalten kann.
Visualisierung folgender Listen
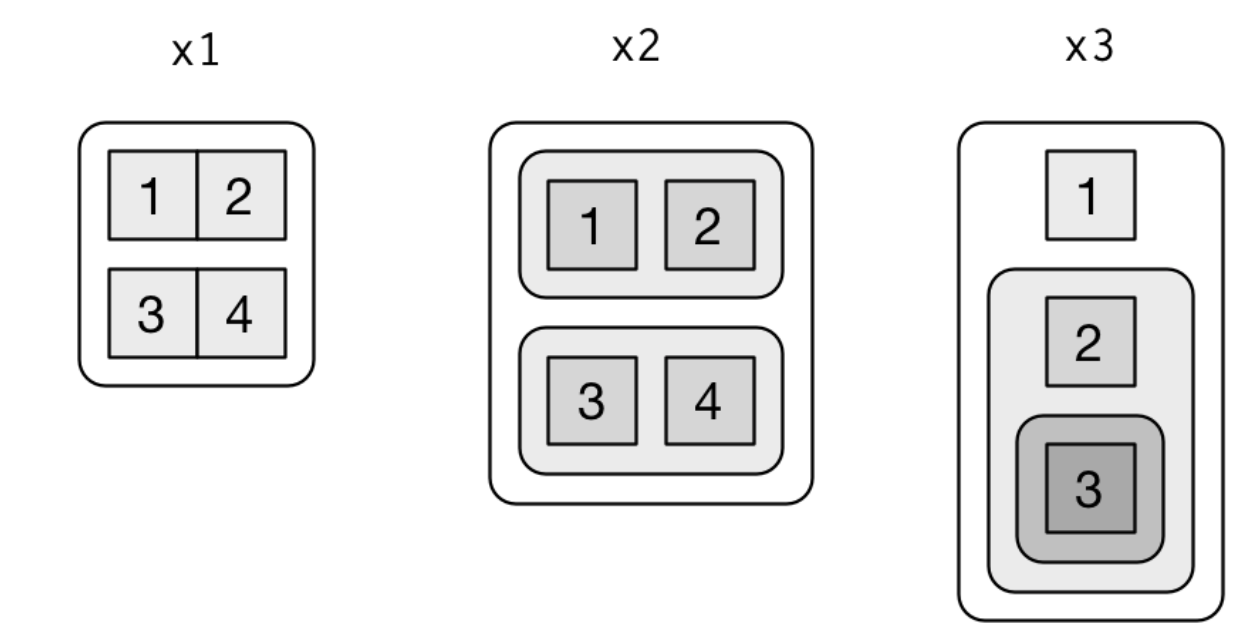
Quelle: R for Data Science von Wickam & Grolemund, 2017 (lizensiert unter CC-BY-NC-ND 3.0 US).
2- und mehrdimensionale Objekte

Matrizen
Matrizen sind 2-dimensionale Objekte dessen Elemente alle den gleichen Datentyp haben müssen. Sie werden vor allem in der numerischen Modellierung verwendet.
- werden mit
matrix()erstellt:
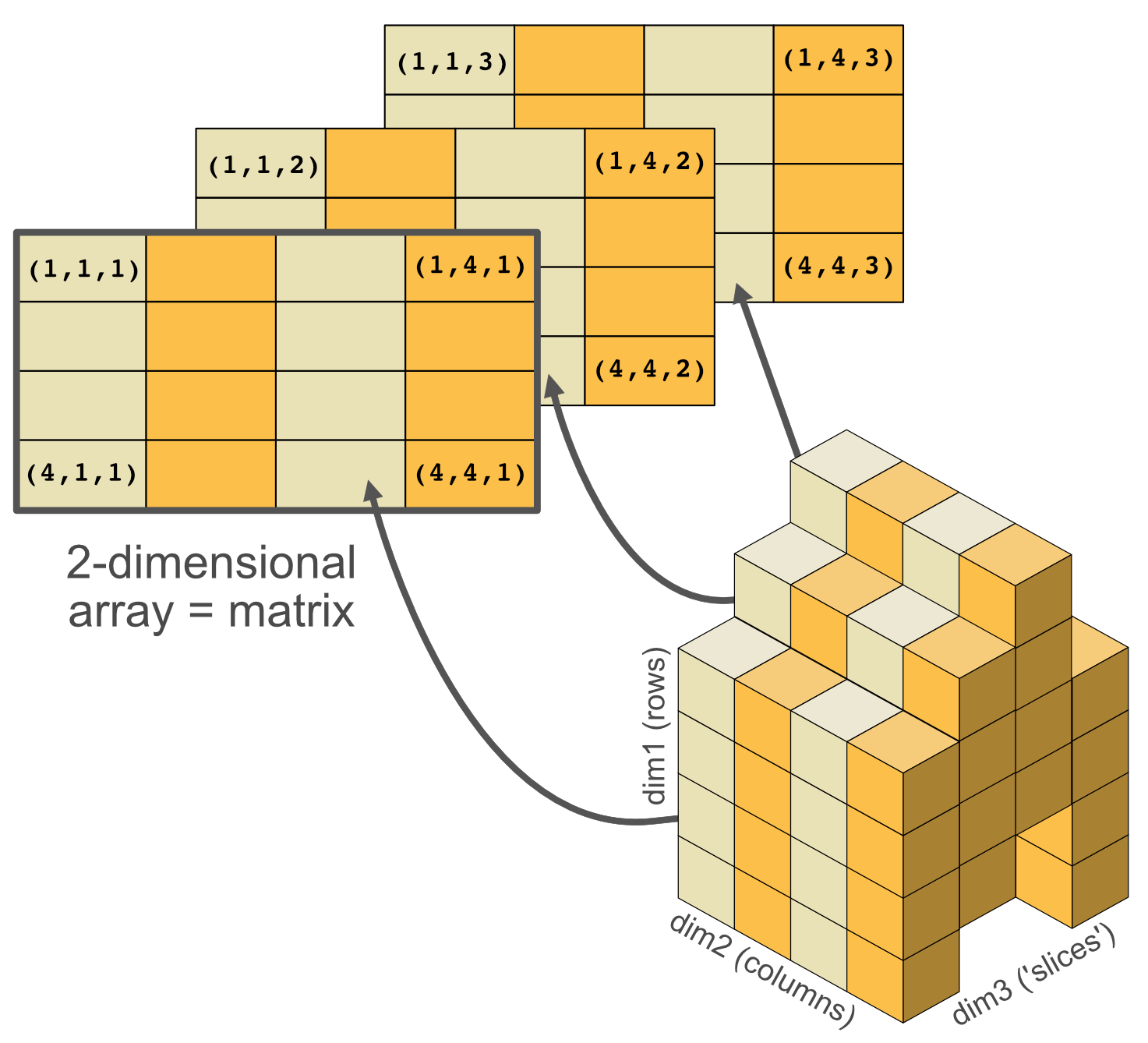
Arrays
Arrays sind multi-dimensionale Objekte des gleichen Datentyps. Sie werden in der Datenanalyse in R aber eher selten genutzt und daher hier nicht näher beschrieben.
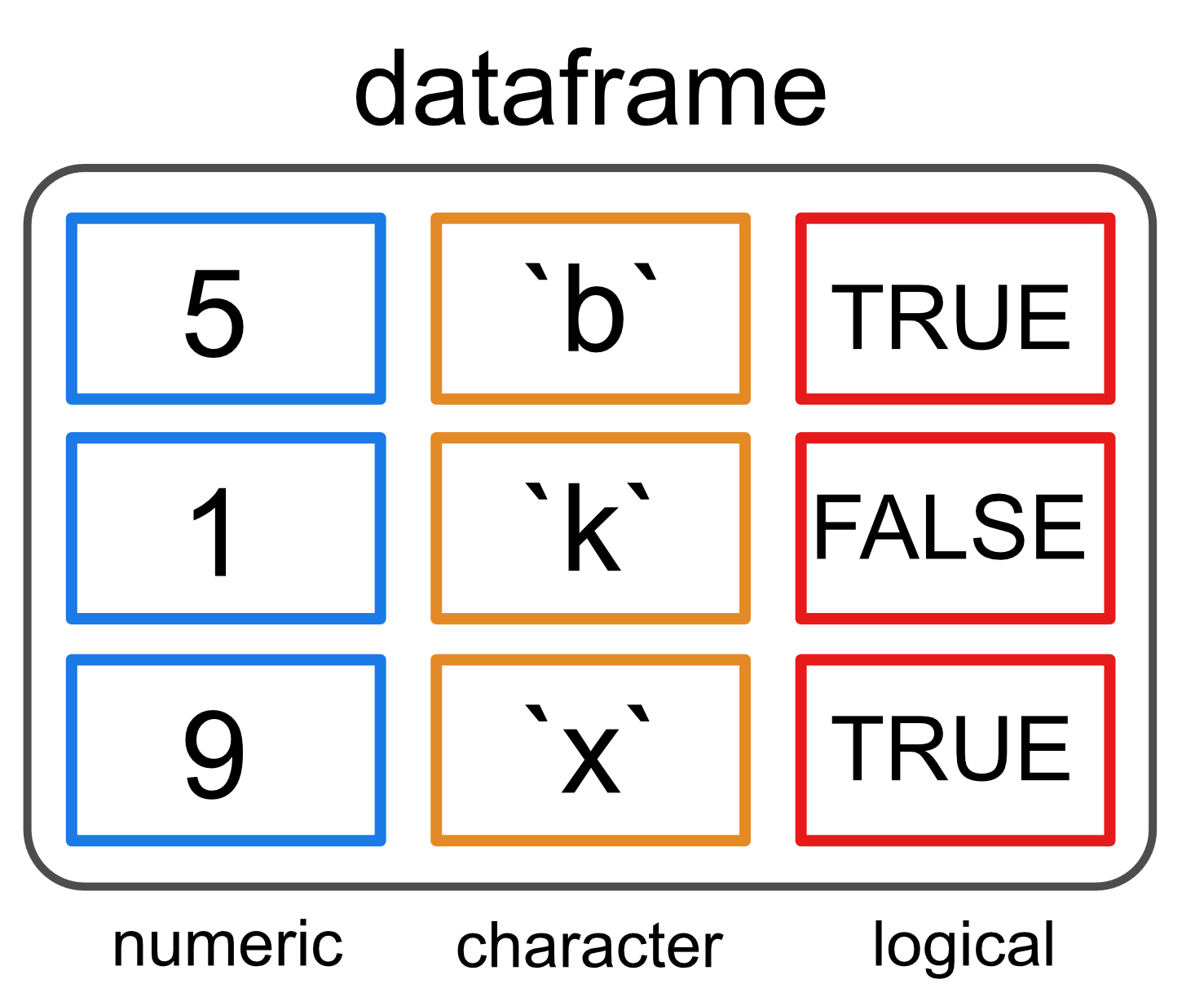
‘Data frames’ | Was ist das?
- Die gebräuchlichste Datenstruktur zum Speichern von Daten in R.
- Repräsentiert eine Liste von gleich langen Vektoren als Spalten
- → dadurch 2-dimensionale Struktur
- Sehr flexibel da sie die gleichen Eigenschaften wie eine Liste und Matrix besitzt.
- Datentypen dürfen pro Vektor, also Spalte, unterschiedlich sein!
‘Data frames’ | Erstellen

- ‘Data frames’ kann man mit der Funktion
data.frame()erstellen, welche Vektoren mit Namen als Input nimmt:
‘Data frames’ | Aus Matrix konvertieren
- Matrizen lassen sich in ‘data frames’ umwandeln mit
as.data.frame()und wieder zurück mitas.matrix()(solange der Datentyp gleich ist):
Längenattribute
length() in 2- und multi-dimensionalen Objekten lässt sich verallgemeinern zu
Namensattribute
names() zeigt in ‘data frames’ den Spaltennamen an:
Erste und letzte Zeilen anzeigen
Sepal.Length Sepal.Width Petal.Length Petal.Width Species
1 5.1 3.5 1.4 0.2 setosa
2 4.9 3.0 1.4 0.2 setosa
3 4.7 3.2 1.3 0.2 setosa
4 4.6 3.1 1.5 0.2 setosa
5 5.0 3.6 1.4 0.2 setosa
6 5.4 3.9 1.7 0.4 setosa2-dimensionale Indexierung

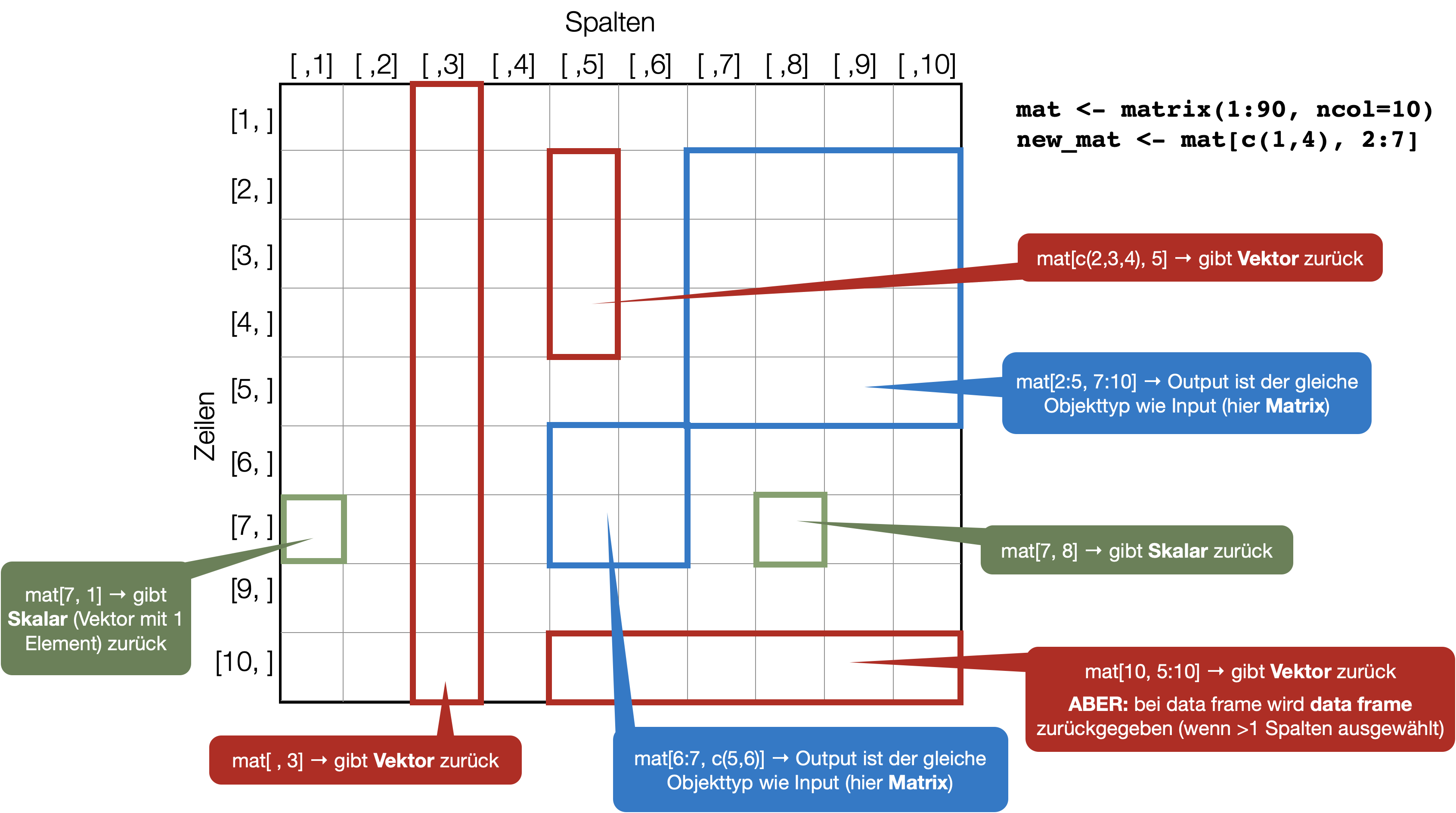
Indexierung | [Zeilen, Spalten]
- Die Indexierung von Matrizen und ‘data frames’ funktioniert genauso wie die Vektor-basierte Indexierung, nur dass die Auswahl für jede Dimension erfolgen muss.
- Es wird jede Dimension mit einem 1D Index getrennt durch ein Komma angegeben (‘integer’, ‘logical’, oder ‘character’ Indizes sind erlaubt).
- Leere Indizes sind erlaubt um alle Zeilen bzw. Spalten auszuwählen.
Neue Objekte über Indexierung erstellen
Teile einer Matrix mittels Positionsindexierung auswählen und neu abspeichern:
Positionsindexierung genauer erklärt
Spalten-Indexierung | $
$ist eine Kurzschreibweise (‘shorthand notation’) zum Extrahieren von benannten Elementen in einer Liste.- Da ‘data frames’ Listen von Vektoren darstellen (in Spalten angelegt), kann diese Kurzschreibweise hier auch zur Auswahl von Spalten verwendet werden:
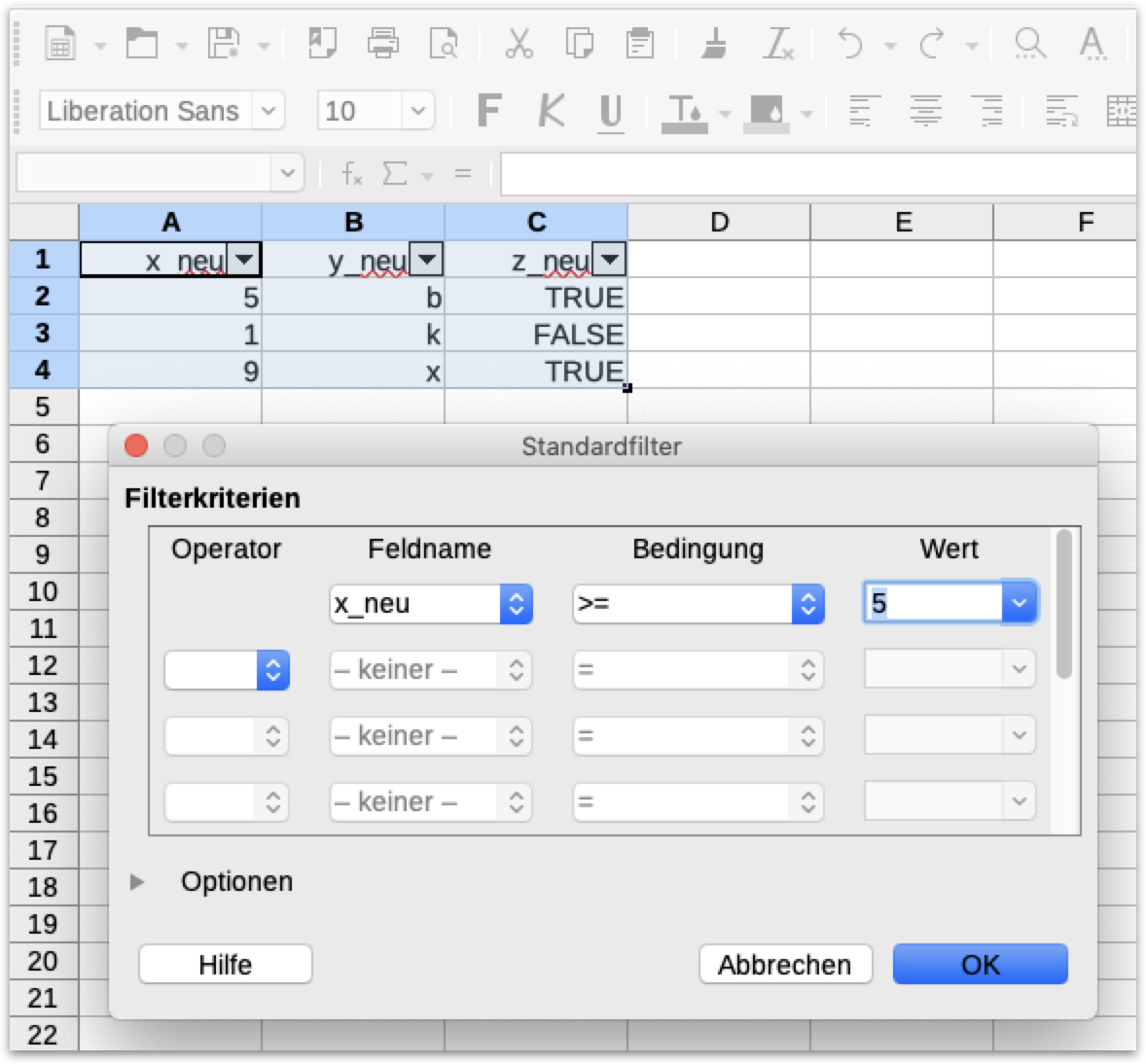
Daten filtern mit logischer Indexierung | 1
Calc vs. R
- Typischerweise filtert man die Zeilen, nicht die Spalten (wie auch in Calc):
df[Zeilenabfrage, Spalten] - D.h. der Zeilenindex stellt eine Abfrage dar, und diese bezieht sich aber immer auf Elemente der ausgewählten Spalte(n).
Daten filtern mit logischer Indexierung | 2
Calc vs. R - ein Vergleich
Auswahl aller Spalten und der Zeilen, wo die Werte in Spalte ‘x_neu’ >= 5 sind.
Daten filtern mit logischer Indexierung | 3
Weitere Beispiele
Werte mittels Indexierung ersetzen
Neue Spalten (Vektoren) mit dem $ Operator anhängen
Your turn …
![]()
00:40
Quiz 1 | PlantGrowth - Objekttyp
![]()
Schauen Sie sich folgenden Datensatz an:
Quiz 2 | PlantGrowth - Objektstruktur
![]()
Schauen Sie sich die Struktur vom PlantGrowth Datensatz an:
'data.frame': 30 obs. of 2 variables:
$ weight: num 4.17 5.58 5.18 6.11 4.5 4.61 5.17 4.53 5.33 5.14 ...
$ group : Factor w/ 3 levels "ctrl","trt1",..: 1 1 1 1 1 1 1 1 1 1 ...Data frames numerisch zusammenfassen
Übersicht der built-in Funktionen
| Funktion | Statistik |
|---|---|
sum() |
Summe |
mean() |
Arithmetischer Mittelwert |
median() |
Median |
min() |
Minimalwert |
max() |
Maximalwert |
range() |
Bereich zwischen Minimum und Maximum |
var() |
Varianz |
sd() |
Standardabweichung |
quantile() |
Quantilen |
cov() |
Kovarianz |
cor() |
(Pearsons) Korrelationskoeffizient |
summary() |
Berechnet unterschiedliche Statistiken |
Es gibt keine built-in Funktion für den Standardfehler und den Variationskoeffizienten sowie die Schiefe und Kurtosis!
Beispiel: Mittelwert
Deskriptive Zusammenfassung
Shortcut mit summary()
Sepal.Length Sepal.Width Petal.Length Petal.Width Species
1 5.1 3.5 1.4 0.2 setosa
2 4.9 3.0 1.4 0.2 setosa
3 4.7 3.2 1.3 0.2 setosa
4 4.6 3.1 1.5 0.2 setosa
5 5.0 3.6 1.4 0.2 setosa
6 5.4 3.9 1.7 0.4 setosa Sepal.Length Sepal.Width Petal.Length Petal.Width
Min. :4.300 Min. :2.000 Min. :1.000 Min. :0.100
1st Qu.:5.100 1st Qu.:2.800 1st Qu.:1.600 1st Qu.:0.300
Median :5.800 Median :3.000 Median :4.350 Median :1.300
Mean :5.843 Mean :3.057 Mean :3.758 Mean :1.199
3rd Qu.:6.400 3rd Qu.:3.300 3rd Qu.:5.100 3rd Qu.:1.800
Max. :7.900 Max. :4.400 Max. :6.900 Max. :2.500
Species
setosa :50
versicolor:50
virginica :50
Zurück zu unseren Demos..

Von Calc zu R wechseln | Demo A
Lineare Regression zum
Fütterungsversuch beim Kabeljau
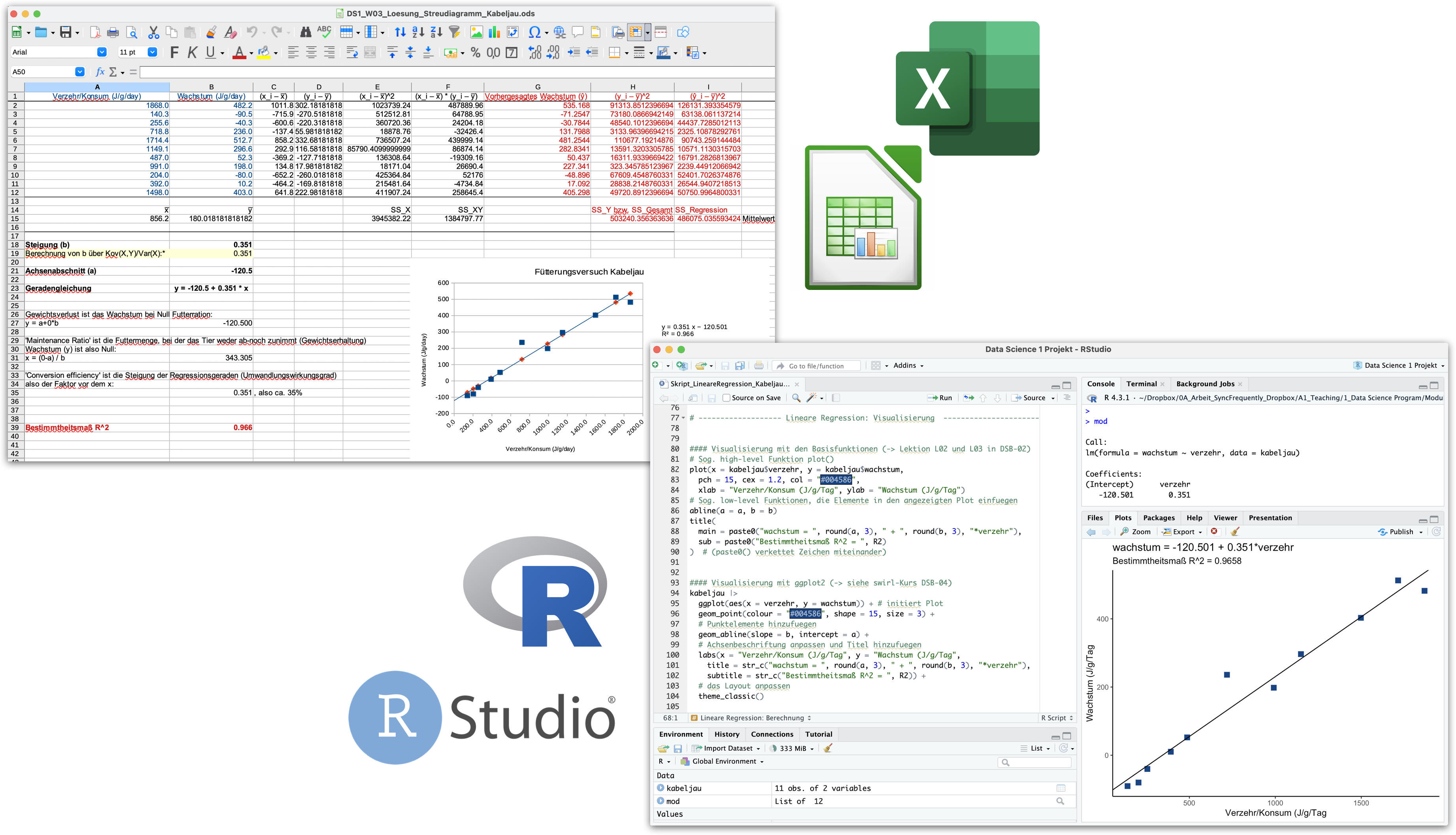
Diese Funktionen kennen wir jetzt alle schon:
# ------------------------------ Vorbereitung ----------------------------------
#### Laden von Paketen
library(tidyverse)
# library(readODS)
# -------------------- Import und Datenaufbereitung ----------------------------
#### Import einer Textdatei im CSV-Format (das gaengigste Format)
kabeljau_csv <- read.csv(file = "Wachstum-Futter.csv")
str(kabeljau_csv) # --> output ist ein data frame
#### Import von Calc- und Excel-Dateien (ODS- und XLSX-Format)
# Import einer ODS-Datei mit dem 'readODS' Paket
kabeljau <- readODS::read_ods("DS1_W03_Streudiagramm_Kabeljau.ods",
sheet = "Daten_Visualisierung")
str(kabeljau) # --> output ist ein tibble
# Import einer XLSX-Datei mit z.B. dem 'readxl' Paket
kabeljau_xlsx <- readxl::read_excel("DS1_W03_Streudiagramm_Kabeljau.xlsx",
sheet = "Daten_Visualisierung")
str(kabeljau_xlsx) # --> output ist ein tibble
#### Datensichtung und -transformation
# Anpassen der Spaltennamen
names(kabeljau) <- c("verzehr", "wachstum")
str(kabeljau)
# Wertebereich pruefen
summary(kabeljau)
# ---------------------- Lineare Regression: Berechnung ------------------------
# (-> Swirl-Lektion L03 in DSB-02-Datenexploration mit R)
#### Manuelle Berechnung
# Um Tipparbeit zu sparen, speichern wir die Spalten als einzelne Vektoren
x <- kabeljau$verzehr
y <- kabeljau$wachstum
# Steigungsparameter b berechnen
b <- cov(x = x, y = y)/ var(x) # der shortcut mit der Kovarianz und Varianz
b
# Achsenabschnitt a berechnen
a <- mean(y) - b*mean(x)
a
# Das Bestimmtheitsmass R^2 berechnen
y_obs <- a + b*x # die vorhergesagten Werte
ss_gesamt <- sum( (y - mean(y))^2 ) # Summenquadrate Gesamt
ss_regression <- sum( (y_obs - mean(y))^2 ) # Summenquadrate der Regression
R2 <- round(ss_regression/ss_gesamt, 4)
R2
##### Zum Vergleich die Regression automatisch berechnen mit lm()
# Erstellung des Modells
mod <- lm(formula = wachstum ~ verzehr, data = kabeljau)
mod
# Ausgabe nur der beiden Koeffizienten
coef(mod)
# Ausgabe aller wichtigen Statistiken des Modells, inklusive von R^2
# (mehr dazu in Data Science 2)
summary(mod)
# ------------------- Lineare Regression: Visualisierung ----------------------
#### Visualisierung mit den Basisfunktionen (-> Lektion L02 und L03 in DSB-02)
# Sog. high-level Funktion plot()
plot(x = kabeljau$verzehr, y = kabeljau$wachstum,
pch = 15, cex = 1.2, col = "#004586",
xlab = "Verzehr/Konsum (J/g/Tag", ylab = "Wachstum (J/g/Tag")
# Sog. low-level Funktionen, die Elemente in den angezeigten Plot einfuegen
abline(a = a, b = b)
title(
main = paste0("wachstum = ", round(a, 3), " + ", round(b, 3), "*verzehr"),
sub = paste0("Bestimmtheitsmaß R^2 = ", R2)
) # (paste0() verkettet Zeichen miteinander)
#### Visualisierung mit ggplot2 (-> siehe swirl-Kurs DSB-04)
kabeljau |>
ggplot(aes(x = verzehr, y = wachstum)) + # initiert Plot
geom_point(colour = "#004586", shape = 15, size = 3) +
# Punktelemente hinzufuegen
geom_abline(slope = b, intercept = a) +
# Achsenbeschriftung anpassen und Titel hinzufuegen
labs(x = "Verzehr/Konsum (J/g/Tag", y = "Wachstum (J/g/Tag",
title = str_c("wachstum = ", round(a, 3), " + ", round(b, 3), "*verzehr"),
subtitle = str_c("Bestimmtheitsmaß R^2 = ", R2)) +
# das Layout anpassen
theme_bw()DS1_VL04a_Demo_LineareRegression_Kabeljau.R
Von Calc zu R wechseln | Demo B
Deskriptive Statistik mit iris
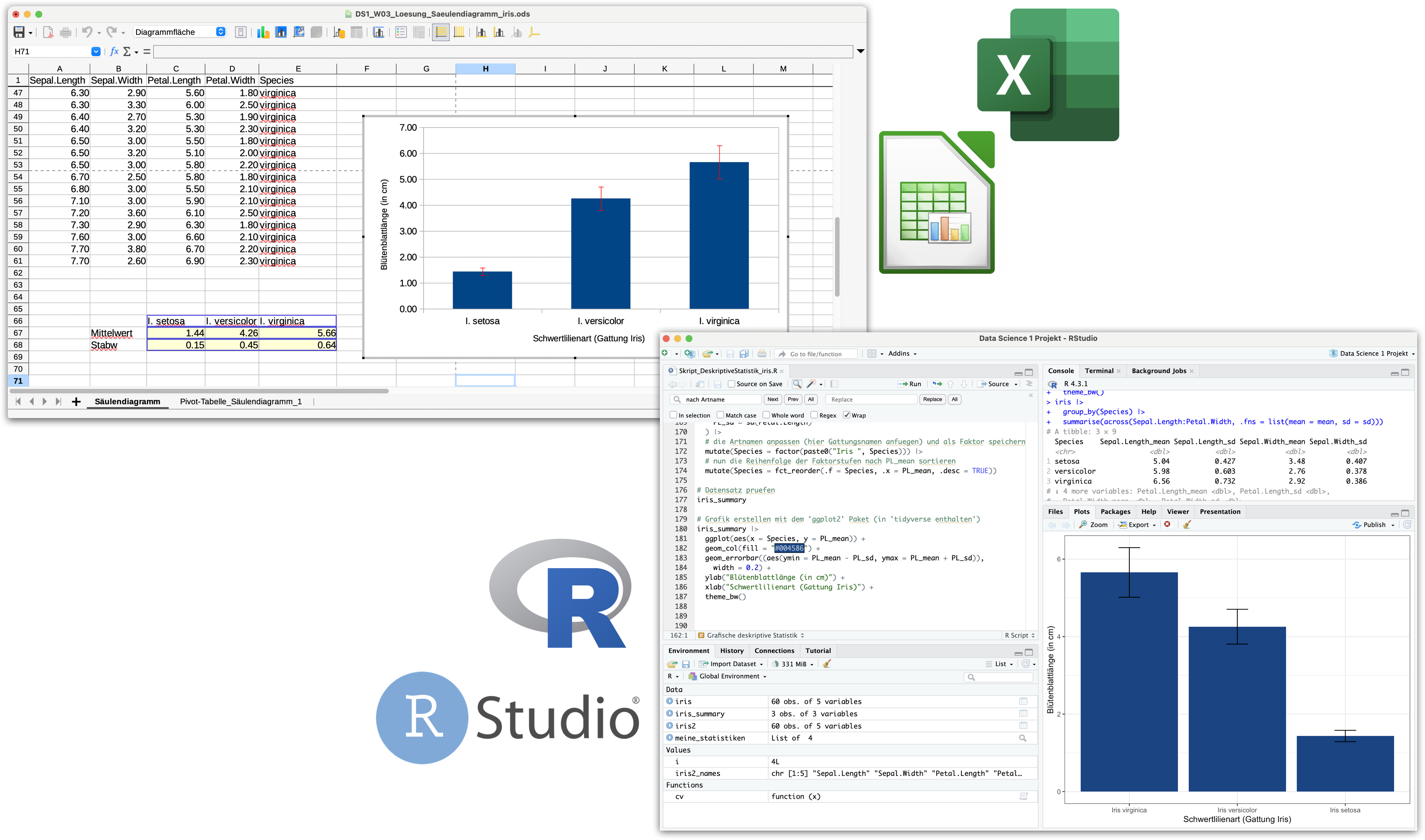
Diese Funktionen kennen wir jetzt alle schon:
# ------------------------------ Vorbereitung ----------------------------------
#### Laden von Paketen
library(tidyverse) # laedt 9 Pakete
# # (das gleiche wie alle Pakete einzeln zu laden)
# library(dplyr)
# library(forcats)
# library(ggplot2)
# library(lubridate)
# library(purrr)
# library(readr)
# library(stringr)
# library(tibble)
# library(tidyr)
#### Eigene Funktionen
# Variantionskoeffizient
cv <- function(x) {
sd(x)/mean(x)
}
# -------------------- Import und Datenaufbereitung ----------------------------
#### Import von CSV-Dateien
# (-> Swirl-Lektion L01 in DSB-03-Datenaufbereitung oder per Anleitung durchs Tidyversum)
# Import der ODS-Datei, welche in den Zeilen 66-68 noch Text enthaelt
iris <- readODS::read_ods("DS1_W03_Saeulendiagramm_mit_iris.ods")
#### Pruefung des Imports und Datensichtung
# (-> Swirl-Lektion L02 in DSB-03)
# Pruefung des Datentyps --> IMMER DIREKT NACH DEM IMPORT VERWENDEN!
str(iris) # str = structure
# Betrachtung des Inhalts
iris
# View(iris)
# Korrektur des Datentyps und der Zeilen
iris <- iris[1:60, ] |>
mutate(across(Sepal.Length:Petal.Width, as.numeric)) |>
mutate(Species = as.factor(Species))
str(iris)
# Welche Werte kommen in jeder Spalte vor?
lapply(iris, unique)
### Weitere Funktionen zur Sichtung einzelner Aspekte
head(iris) # zeigt erste 6 Zeilen (Kopfzeilen)
tail(iris) # zeigt letzte 6 Zeilen (Endzeilen)
class(iris) # Identifikation der Objektklasse (Vektor, Matrix, dataframe,..)
nrow(iris) # Anzahl Zeilen
ncol(iris) # Anzahl Spalten
dim(iris) # Anzahl aller Dimensionen
names(iris) # Spaltennamen
typeof(iris$Sepal.Length) # Datentyp von Spalte 'Sepal.Length'
typeof(iris$Species) # Datentyp von Spalte 'Species'
# ----------------------- Deskriptive Statistik --------------------------------
#### Berechnung mehrerer Statistiken für jede Spalte im data frame
summary(iris)
#### Berechnung versch. Statistiken der Kronblattlaenge, gruppiert nach Art
# (-> Lektion L01 in DSB-02-Datenexploration mit R)
# (-> Lektion L06-Gruppierte Aggregation in DSB-03)
iris_summary <- iris |>
group_by(Species) |>
summarise(
PL_mean = mean(Petal.Length), # Mittelwert
PL_median = median(Petal.Length), # Median
PL_var = var(Petal.Length), # Varianz
PL_sd = sd(Petal.Length), # Standardabweichung
PL_se = sd(Petal.Length)/sqrt(length(Petal.Length)), # Standardfehler
PL_cv = cv(Petal.Length) # Variationskoeffizient
) |>
# die Artnamen anpassen (hier Gattungsnamen anfuegen) und als Faktor speichern
mutate(Species = factor(paste0("Iris ", Species))) |>
# nun die Reihenfolge der Faktorstufen nach PL_mean sortieren
mutate(Species = fct_reorder(.f = Species, .x = PL_mean, .desc = TRUE))
# Zusammenfassung ansehen
iris_summary
#### Saeulendiagramm erstellen mit dem 'ggplot2' Paket
# (siehe auch swirl-Kurs DSB-04-Datenvisualisierung mit ggplot2)
iris_summary |>
ggplot(aes(x = Species, y = PL_mean)) + # initiert Plot
# die Saeulen hinzufuegen
geom_col(fill = "#004586") +
# die Fehlerbalken hinzufuegen
geom_errorbar((aes(ymin = PL_mean - PL_sd, ymax = PL_mean + PL_sd)),
width = 0.2) +
# Achsenbeschriftung anpassen
ylab("Kronblattlänge (in cm)") +
xlab("Schwertlilienart (Gattung Iris)") +
# das Layout anpassen
theme_bw()DS1_VL04b_Demo_DeskriptiveStatistik_iris.R
Übungsaufgabe

Zu bearbeitende swirl-Lektionen
![]()
Kurs DS1-01-R Grundlagen
- L13-Listen
- (L14-Matrizen und Arrays)
- L15-Data frames
- (L16-Praktische Tipps rund ums Programmieren)
Kurs DS1-02-Datenexploration mit R (optional)
- L01-Erste numerische Analyse
- (L02-Erste grafische Analyse)
- (L03-Regressionsgerade berechnen)
Übungsskript
![]()
- 3 interne Datensätze sichten: DS1_W06_Uebungsskript_Datensichtung.R
Wie fühlen Sie sich jetzt…?

Total konfus?

Keine Sorge…
… im swirl-Kurs werden Sie direkt an die Hand genommen und Stück für Stück angeleitet.
Überblick aller Objekttypen
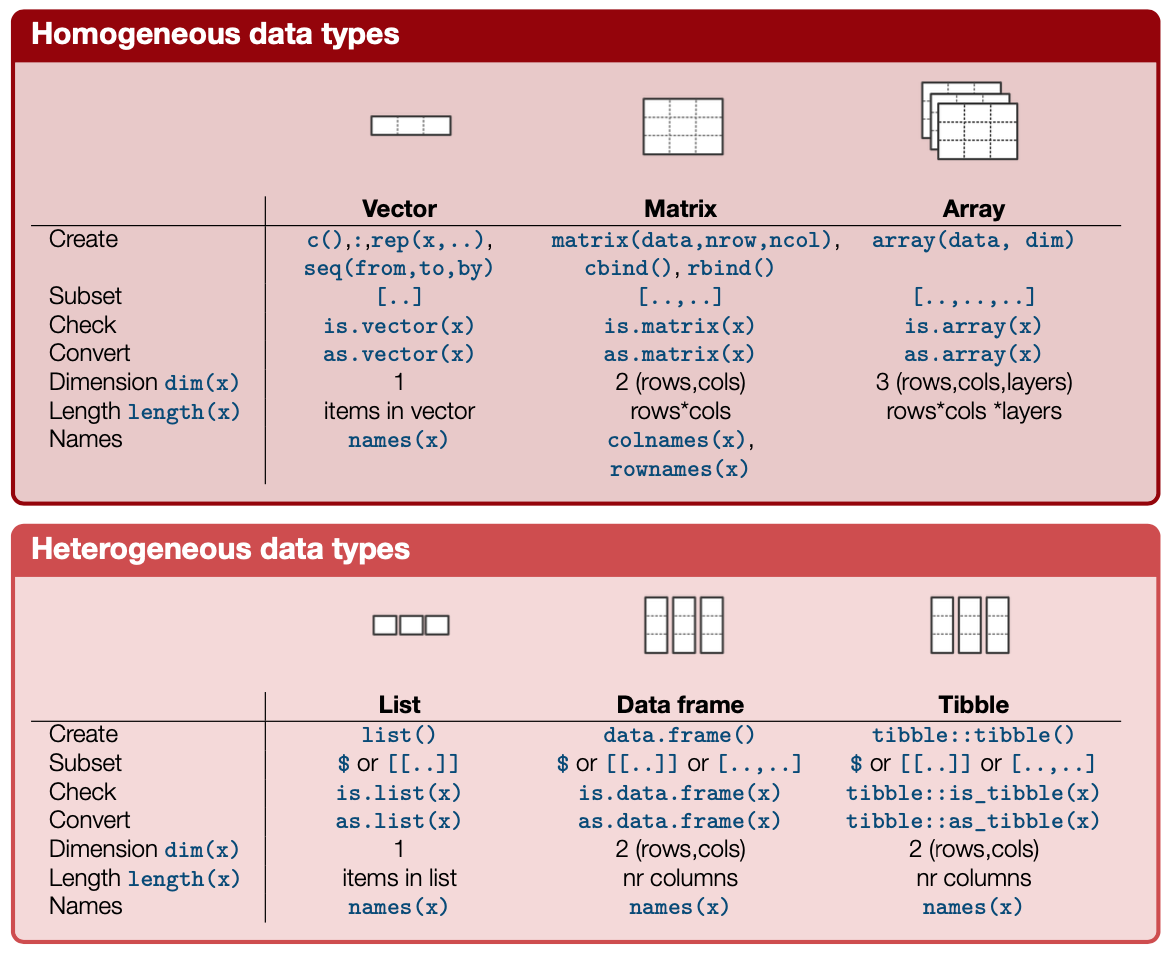
Siehe auch DSB cheatsheet
Basic R functions (auf Moodle).
Überblick aller Objektindexierungen
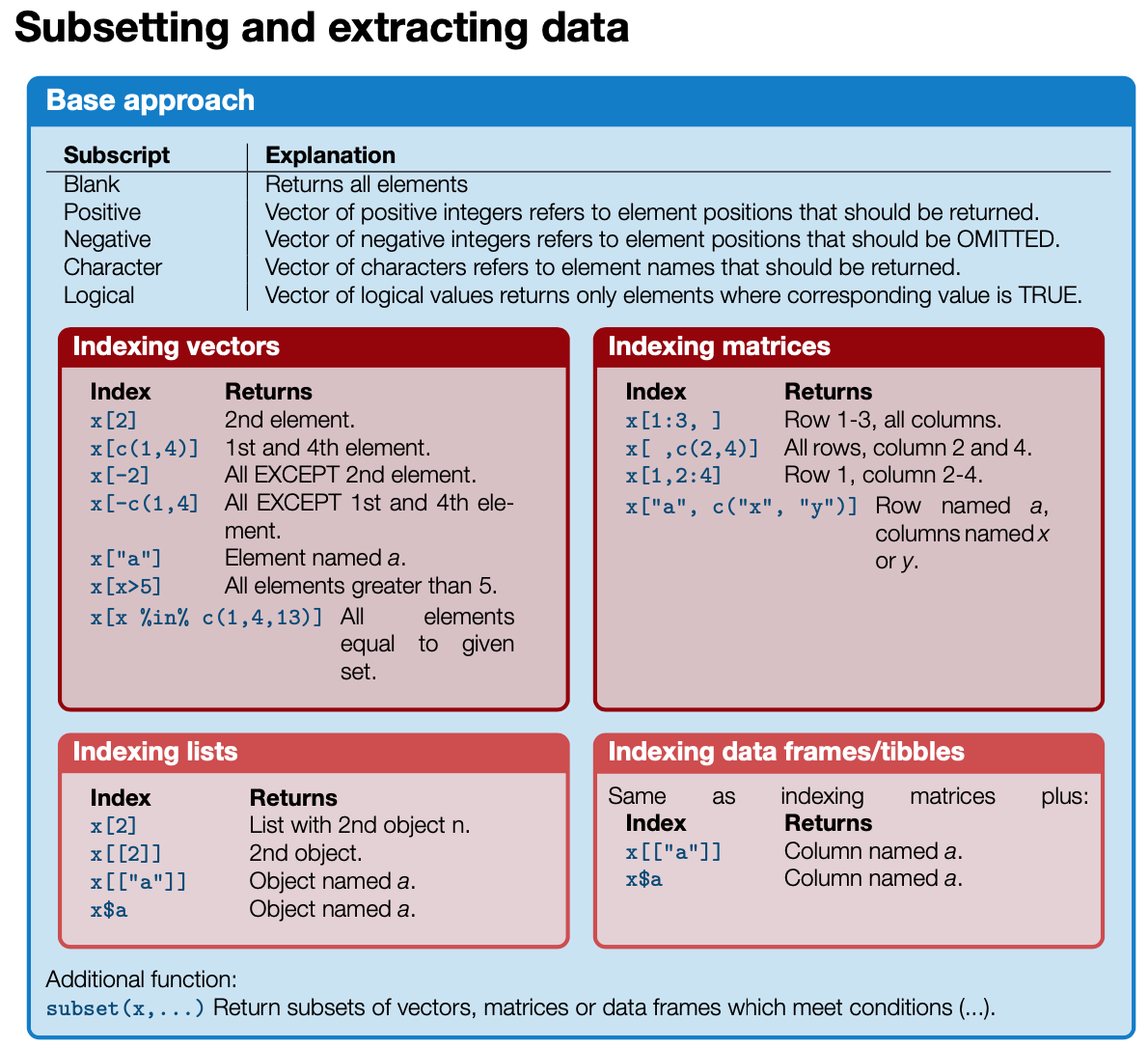
Siehe auch DSB cheatsheet
Basic R functions (auf Moodle).
Total gelangweilt?

Dann machen Sie doch einfach die nächsten swirl Lektionen auch schon…

Bei weiteren Fragen: saskia.otto(at)uni-hamburg.de

Diese Arbeit is lizenziert unter einer Creative Commons Attribution-ShareAlike 4.0 International License mit Ausnahme der entliehenen und mit Quellenangabe versehenen Abbildungen.
Kurswebseite: Data Science 1