
Grundlagen in R:
Variablen und Vektoren
Data Science 1 - Programmieren & Visualisieren
Saskia Otto & Monika Eberhard
Universität Hamburg, IMF
Wintersemester 2025/2026
Lernziele
![]()
Am Ende dieser VL- und Übungseinheit werden Sie
- einen ersten Eindruck für eine deskriptive statistische Analyse in R bekommen haben.
- das erste der 4 Grundelemente (Variablen) in R kennen und beherrschen.
- Datentypen abfragen und umwandeln können.
- Vektoren und Zahlenfolgen in R erstellen können.
- Vektoren indexieren und mittels Operatoren filtern können.
- den Unterschied zwischen Zuweisungs- Vergleichs- und Verknüpfungsoperatoren kennen.
Grundelement - Variablen
Variablen
Zur Erinnerung
Eine Variable ist eine Möglichkeit, um im Programm eine Zahl, ein Zeichen oder Zeichenkette oder einen boolschen Wert (wahr oder falsch) zu speichern.
Variablen | Zuweisung
- In R betrachtet man einzelne Werte oft als Skalare oder Vektoren mit einem Element.
- Werte werden gespeichert, indem man sie einem Variablen- bzw. Skalarnamen zuweist - und zwar mit dem Zuweisungsoperator
<-:
Variablen | Zugriff
Über den Namen kann auf den Inhalt zugegriffen werden:
- es kann der Inhalt in der Konsole nur angezeigt werden
- oder es können weitere Operationen erfolgen, die sich auch wieder abspeichern lassen.
Variablen | Die globale Umgebung
- Variablen, Objekte und Funktionen, die Sie in R generieren, erscheinen im Environment Reiter unter Global Environment.
- Die globale Umgebung ist der erste Ort des Suchverzeichnisses von R und stellt Ihren aktuellen Arbeitsbereich dar.
- Dieser Arbeitsbereich wird mit jedem Schließen von R/RStudio gelöscht (es sei denn Sie verwenden ein RStudio Projekt und speichern es explizit).
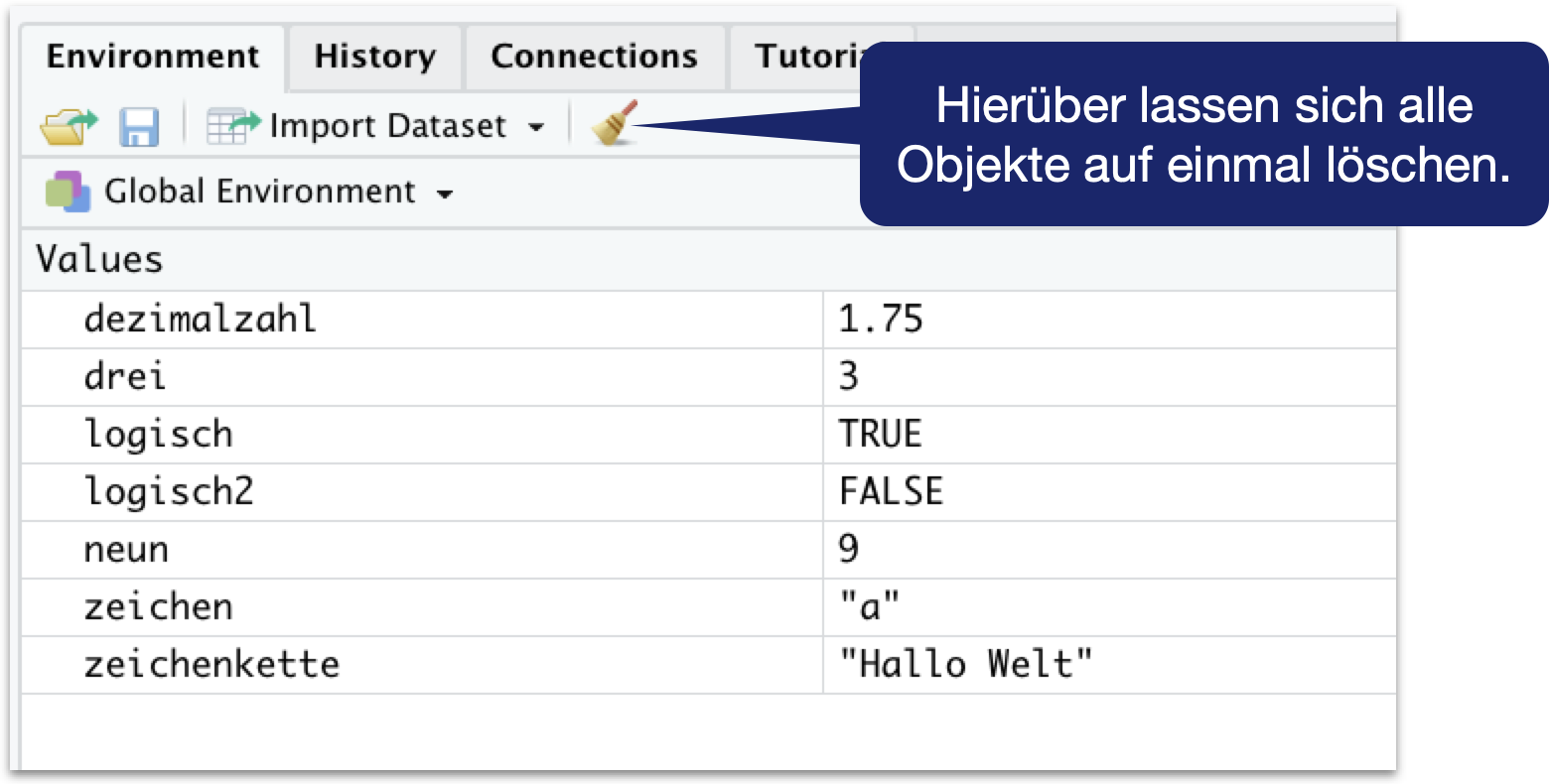
Variablen | Entfernen
Einzelne Variablen, Objekte und Funktionen lassen sich aus dem Arbeitsbereich mit rm() und dem entsprechenden Namen löschen:
Sie können die Liste aller Objekte des Arbeitsbereichs auch in der Konsole anzeigen lassen mit ls():
→ Wie Sie sehen, ist logisch2 nicht mehr gelistet, nachdem es gelöscht wurde.
Variablen | Elementare Datentypen in R
Das Datum (date) und Faktoren (factor) stellen zusammengesetzte Datentypen dar.
| Datentyp | Definition | Variablentyp | Skalenniveau |
|---|---|---|---|
| logical | Logische (Boolean) Werte (TRUE oder FALSE) |
kategorial | nominalskaliert |
| integer | Natürliche ganze Zahlen | quantitativ (diskret) | ordinal-/intervall-/verhältnisskaliert |
| double | Gleitkommazahl | quantitativ (kontinuierlich) | intervall-/verhältnisskaliert |
| numeric | Alle Zahlen (integer und double) | quantitativ | |
| complex | Komplexe Zahlen (z.B. 5i) | quantitativ | |
| character | Zeichen (oder Zeichenkette) | qualitativ/kategorial | nominalskaliert |
| NULL | leerer Inhalt |
Den Datentyp ermitteln | typeof()
Datentypen gezielt abfragen | is.xxx()
Diese Funktionen geben ein TRUE oder FALSE zurück:
Datentypen umwandeln | as.xxx()
- Der Datentyp lässt sich ineinander umwandeln, allerdings nicht in jede Richtung.
- Vom spezifischeren in den allgemeineren Datentyp funktioniert immer und ohne Datenverlust: logical > integer > double > character
Your turn …
![]()
01:30
Quiz 1 | Zuweisungen
![]()
Quiz 2 | Zuweisungen
![]()
Quiz 3 | Datentypen
![]()
Bevor auf my_value in Zeile 2 zugegriffen werden kann, muss das Skalar erstmal definiert werden. Dies geschieht mit dem Zuweisungsoperator (<-).
Anschl. muss eine Funktion aufgerufen werden, die einen logischen Wert zurückgibt. Dies kann nur eine der is.xxx Funktionen sein. Welchen Datentyp könnte ‘A1’ haben?
Die einfachste Struktur: Vektoren

Vektoren erstellen | 1
Vektoren erstellen | 2
Struktur von Vektoren | 1
- sind immer flach, auch wenn man
c()verschachtelt:
- ist das gleiche wie:
Struktur von Vektoren | 2
- haben eine bestimmte Länge und Struktur die mit
length()undstr()ermittelt werden kann:
Your turn …
![]()
01:00
Quiz 4 | Vektoren erstellen
![]()
Quiz 5 | Vektoren erstellen
![]()
Vektorregeln

Arbeiten mit Vektoren
4 wichtige Regeln
Implizierte Datentyp-Umwandlung (‘coercion’)
Handling von Vektoren unterschiedlicher Länge (‘recycling’)
Namen für Elemente in Vektoren
Extrahieren von Elementen bzw. Teilmengen bilden (Indexierung oder ‘indexing’)
1. ‘Coercion’ | 1
- Alle Elemente eines Vektors müssen den gleichen Datentypen haben.
- Unterschiedliche Typen werden zwangsumgewandelt (‘coerced’) und zwar zum flexibleren Typen (‘logical’ → ‘integer’ → ‘double’ → ‘character’)
1. ‘Coercion’ | 2
Nutzen beim Rechnen
- Wenn logische Elemente zu ‘integer’ oder ‘double’ umgewandelt werden, wird jedes
TRUEzu einer 1 undFALSEwird zu 0. - Dies kann bei Berechnungen mit
sum()undmean()sehr nützlich sein:
2. Recycling-Regel
- Implizierte Verlängerung von Vektoren bei Operationen mit 2 Vektoren durch:
- der kürzere Vektor wird auf die gleiche Länge des längeren Vektors verlängert, indem die Lücke mit den bereits vorhandenen Elementen gefüllt wird.
3. Vektorelemente benennen
Alle Elemente in Objekten können Namen erhalten - auch in Vektoren:
- Entweder beim Erstellen mit der Funktion
c()
- oder anschließend mit der Funktion
names().
4. Indexierung (‘indexing’) | 1
[...]ist die Funktion mit der Vektoren indexiert werden.- Die entstandene Teilmenge lässt sich dann als neuer Vektor abspeichern:
neuer_vektor <- alter_vektor[auswahl]. - Wenn >1 Element ausgewählt/ausgeschlossen werden, müssen diese als Vektor übergeben werden!
3 Arten einen Vektor zu indexieren:
- Über die Angabe der Position der auszuwählenden (oder auszuschließenden!) Elemente.
- Über die Namen der auszuwählenden (oder auszuschließenden!) Elemente
- Mittels logischem Vektor.
4. Indexierung (‘indexing’) | 2
Ausschluss von Elementen
- Ein negatives Vorzeichen definiert alle auszuschließenden Elemente (bei Positionsindexierung).
- Der Nicht-Operator ! wählt genau das Gegenteil von dem was angegeben ist (bei logischer Indexierung).
4. Indexierung | Über die Position 1
![]()
Die Positionsangabe erfolgt über eine Ganzzahl oder einen numerischen Vektor mit Ganzzahlen. Der Index startet immer mit 1.
[1] "zwei"[1] "eins" "drei" "vier" "fünf"[1] "fünf" "eins" "drei"[1] "zwei" "vier"[1] "eins" "zwei" "drei"[1] "drei" "zwei" "eins"[1] "eins" "eins" "zwei" "zwei" "drei" "drei" "drei" "drei"4. Indexierung | Über die Position 2
Calc vs. R
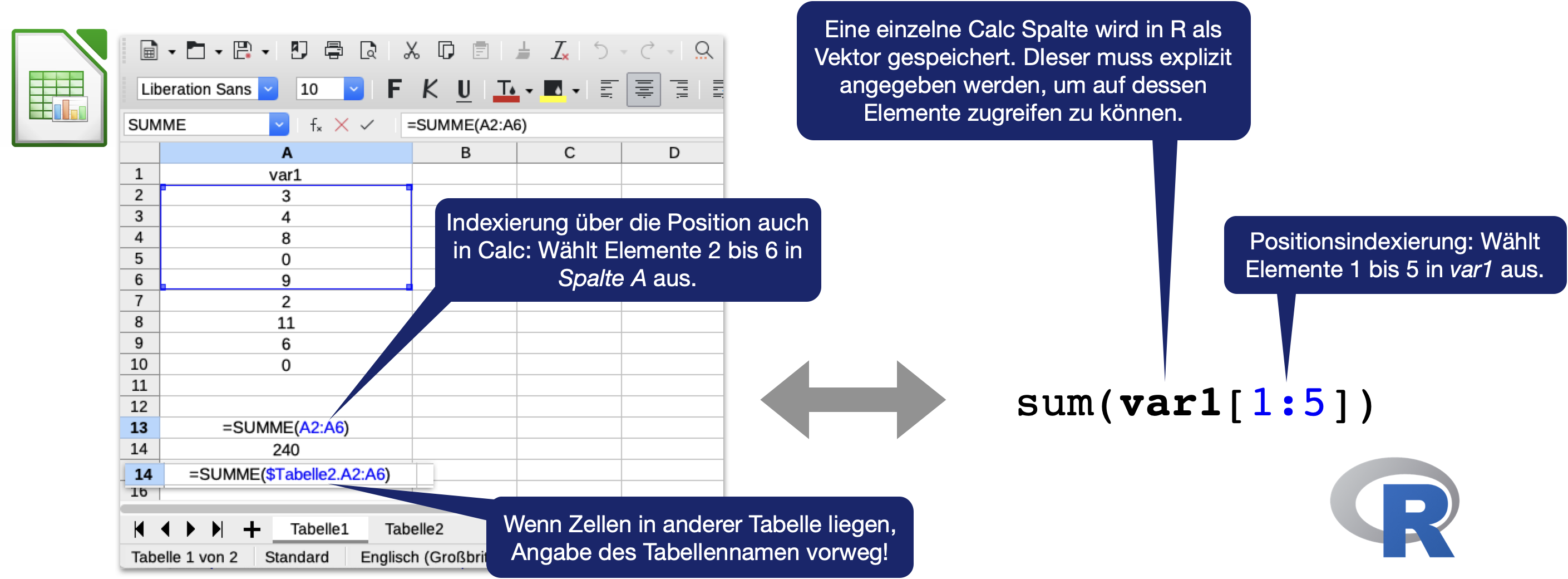
4. Indexierung | Mittels logischem Vektor
- Hier erfolgt die Auswahl über eine logische Abfrage (
Vektor[Abfrage]) mittels Vergleichs- und Verknüpfungsoperatoren oder Funktionen, die auch TRUE/FALSE zurückgeben:
Logische Indexierung für NA Handling
- Fehlende Werte werden durch
NAangezeigt (= ‘not available’). NAnimmt immer den Datentyp der anderen Elemente an, auch wenn das meist nicht sichtbar ist.- Mit
is.na()kann geprüft werden, ob ein Element ein NA, um sich anschl. die Summe aller NAs ausgeben zu lassen.
Your turn …
![]()
01:30
Quiz 6 | ‘Coercion’-Regel
![]()
Quiz 7 | Recycling-Regel
![]()
Quiz 8 | Indexierung
![]()
Ein Vektor x enthält 20 verschiedene Elemente.
Quiz
Operatoren

Nützliche Operatoren für Abfragen
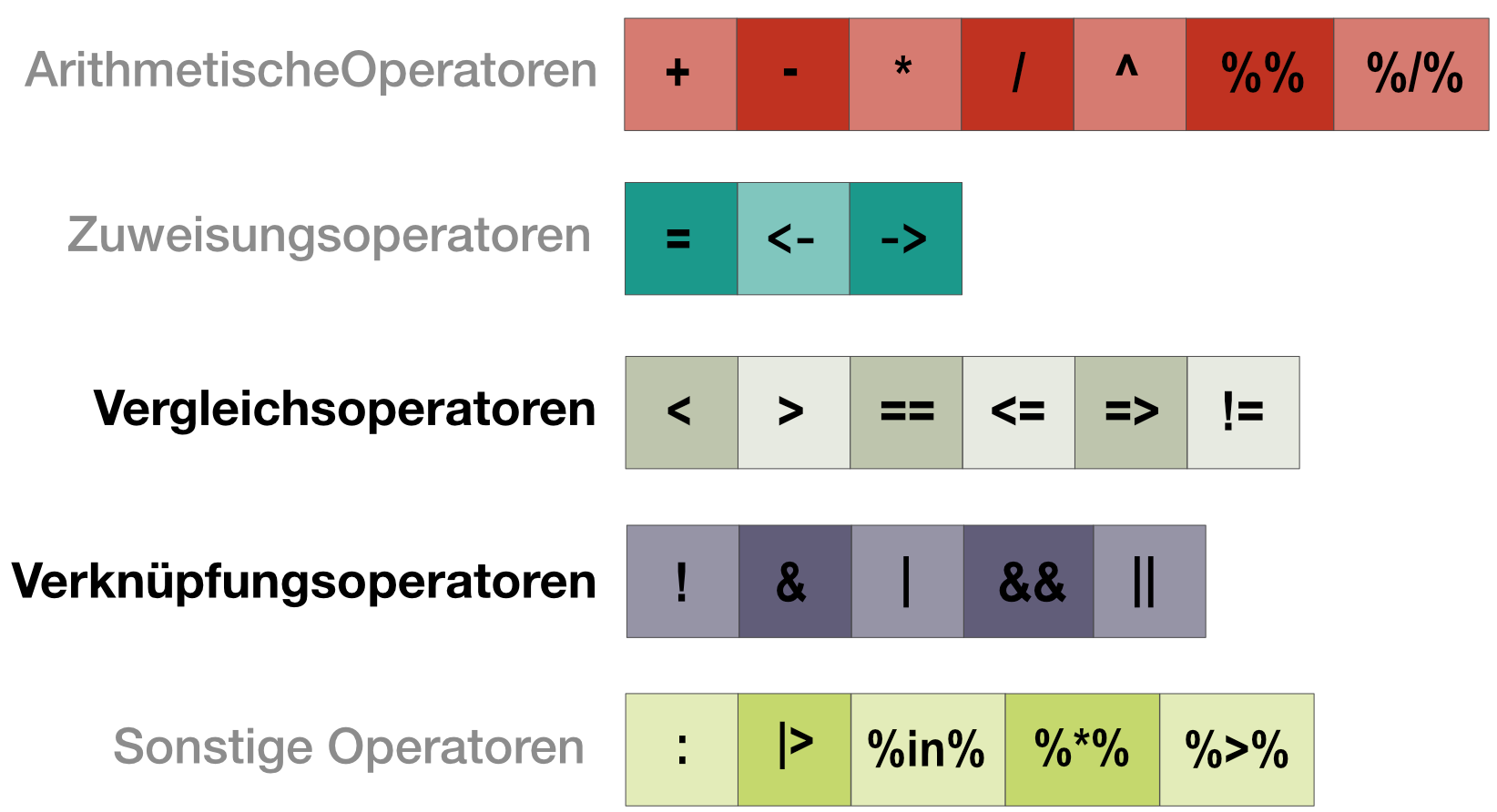
Vergleichs- und Verknüpfungsoperatoren geben immer einen logischen Wert (TRUE oder FALSE) zurück.
Vergleichsoperatoren
Im Englischen ‘relational operators’
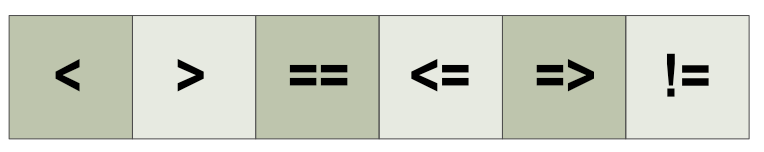
| Operator | Anwendung | Beschreibung |
|---|---|---|
| < | a < b | a ist KLEINER als b |
| > | a > b | a ist GRÖSSER als b |
| == | a == b | a ist GLEICH wie b |
| <= | a <= b | a ist KLEINER als oder GLEICH wie b |
| >= | a >= b | a ist GRÖSSER als oder GLEICH wie b |
| != | a!=b | a ist NICHT GLEICH wie b |
Vergleichsoperatoren | Beispiele
Verknüpfungsoperatoren
Im Englischen ‘logical operators’

| Operator | Anwendung | Beschreibung |
|---|---|---|
| & | a & b | Elementweise UND Operation (arbeiten mit Vektoren) |
| | | a | b | Elementweise ODER Operation (arbeiten mit Vektoren) |
| ! | !a | Elementweise NICHT Operation (arbeiten mit Vektoren) |
Verknüpfungsoperatoren | 2
‘Regenschirm-Logik’
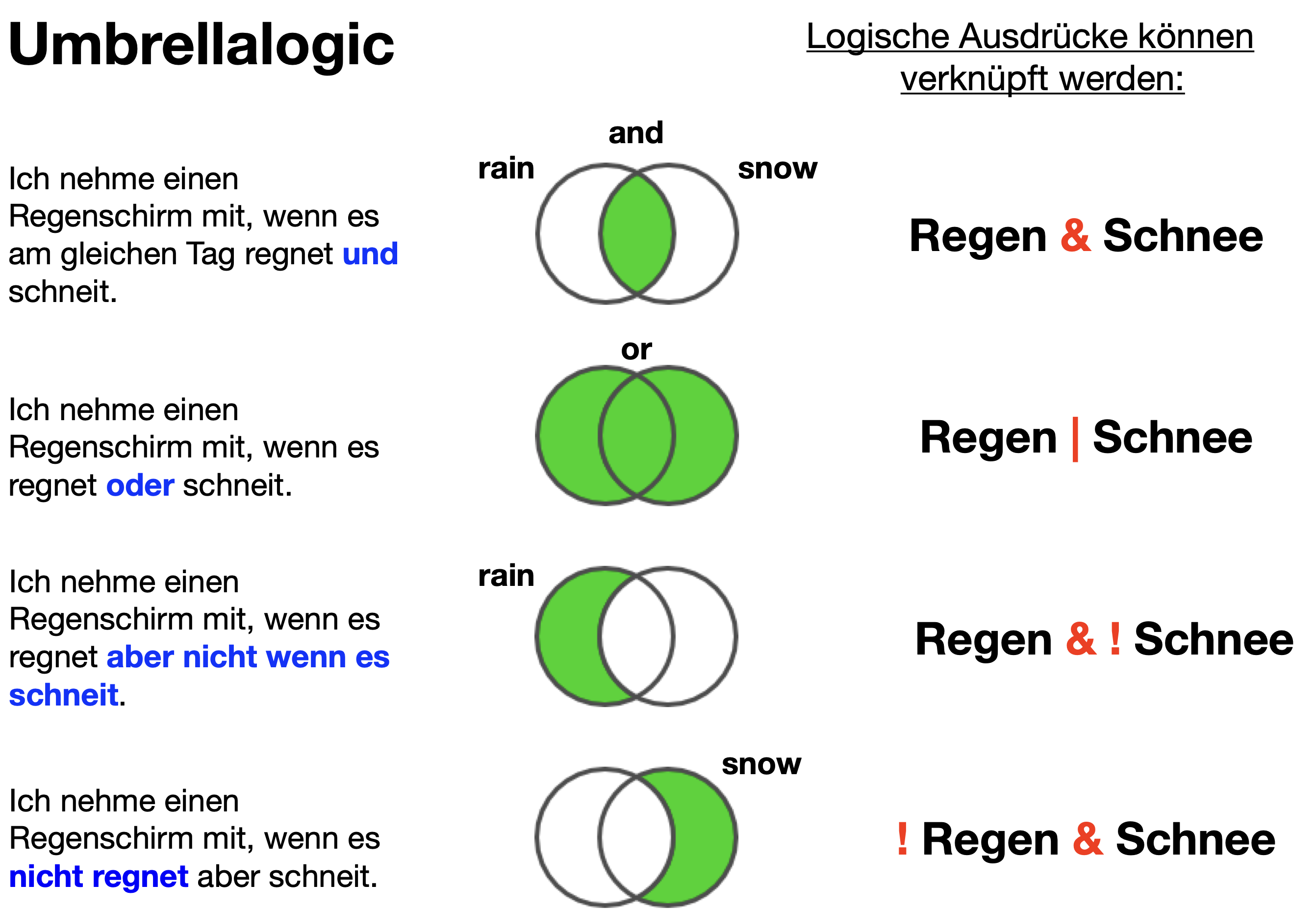
Verknüpfungsoperatoren | 3
Beispiel 1 zu kombinierten logischen Abfragen
Verknüpfungsoperatoren | 4
Beispiel 2 zu kombinierten logischen Abfragen
Vektorisierte Operationen in R

Einfache Berechnungen
Warum 4 Werte???
Vektorisierte Berechnungen
Berechnungen in R sind vektorisiert, d.h. die Berechnung wird auf jedes einzelne Element des Vektors angewendet.
Calc vs. R | Bsp. proportionaler Anteil 1
Spinnendichte im Golf von Kalifornien
Gegeben ist eine Messreihe der Spinnendichte (\(no./m^2\)) auf verschiedenen Inseln im Golf von Kalifornien aus dem Jahre 1992 (aus Studie von Polis et al., 1998).

Calc vs. R | Bsp. proportionaler Anteil 2
Nun soll der proportionale Anteil der Dichte pro Insel berechnet werden: \(p_i=\frac{d_i}{\sum(d_i)}\)
In Calc gäbe es 2 Ansätze
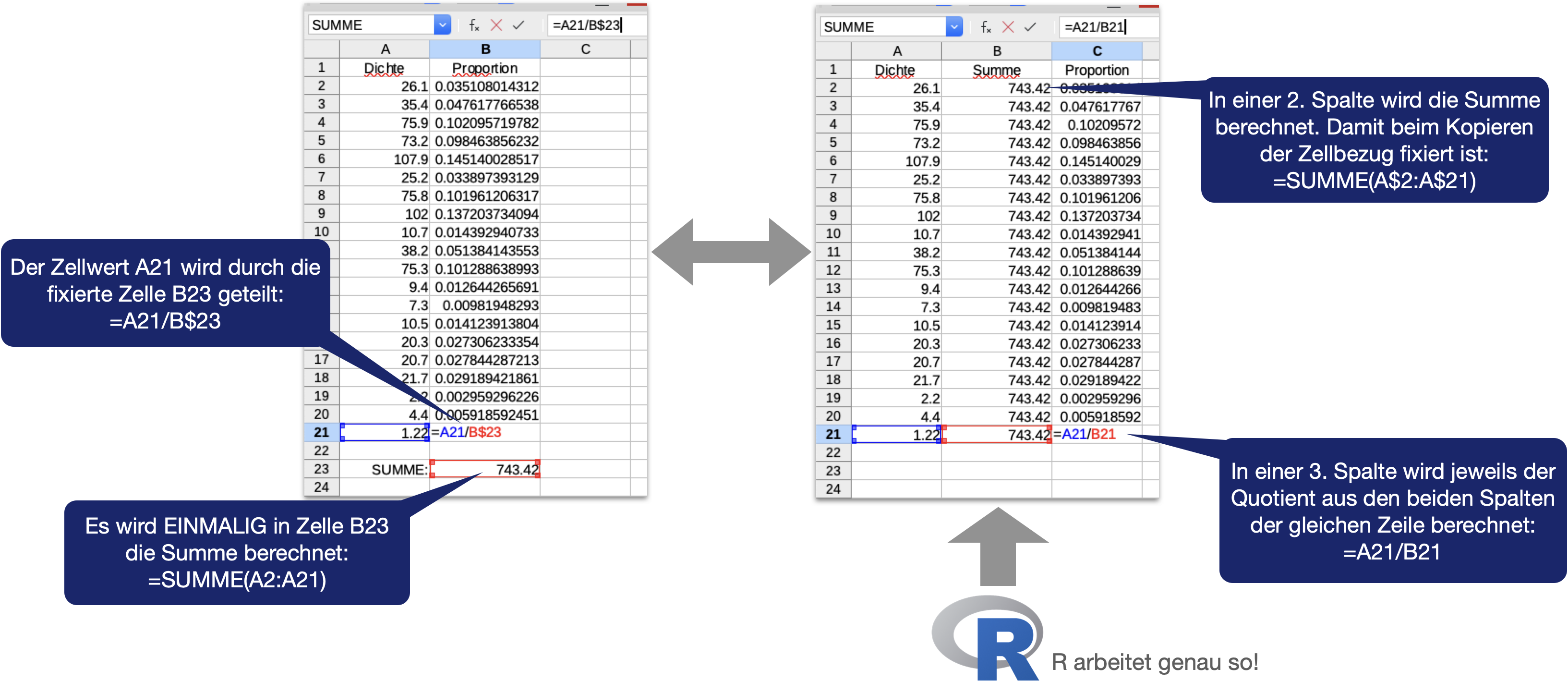
Calc vs. R | Bsp. proportionaler Anteil 3
Calc vs. R | Bsp. proportionaler Anteil 4
Berechnung in R
Übungsaufgabe

Zu bearbeitende swirl-Lektionen
![]()
Kurs DS1-01-R Grundlagen
- L08-Atomare Vektoren als einfachster Objekttyp
- L09-Zahlenfolgen
- L10-Verhalten von Vektoren
- L11-Vektorindexierung
- L12-Filtern von Vektoren
Wie fühlen Sie sich jetzt…?

Total konfus?

Keine Sorge…
… im swirl-Kurs werden Sie direkt an die Hand genommen und Stück für Stück angeleitet.
Total gelangweilt?

Dann testen Sie doch Ihr Wissen in folgendem Abschlussquiz…
Abschlussquiz
![]()
Abschlussquiz
![]()
1 | Zuweisungen
Der Skalar n in der letzten Operation wurde vorher nicht definiert. Daher muss sich die vorherige Zuweisung auf dieses Skalar beziehen. Auch y wurde vorher nicht definiert, daher kann es sich beim ‘Grösser’-Zeichen in in der letzten Zeile nicht um einen Vergleichsoperator handeln, sondern muss der erste Teil des Zuweisungsoperators sein.
2 | ‘Coercion’-Regel
Eine Zeichenkette (in diesem Fall ‘a’) hat keine korrespondierende Zahl, in die es umgewandelt werden kann. Dagegen kann eine Zahl einfach in eine Zeichenkette umgewandelt werden.
3 | ‘Coercion’-Regel
In diesem Fall ist 1 klar als ‘integer’ definiert, daher wird das TRUE zum ‘integer’ umgewandelt.
4 | ‘Coercion’-Regel
Alle TRUEs werden zu 1 umgewandelt und alle FALSEs zu 0. Summiere einfach den Vektor mit der sum() Funktion: sum(x)
5 - Challenge
Wie könnten alle NAs in einem Vektor x mit sehr vielen Elementen schnell ermittelt werden?
Zuerst wird mit der is.na() Funktion bei jedem Element abgefragt, ob es sich um ein NA handelt oder nicht.
Anschließend kann mit dem zurückgegebenen logischen Vektor die Summe aller TRUEs berechnet werden - denn R wandelt bei mathematischen Operationen von logischen Werten alle FALSEs in eine 0 um und alle TRUEs in eine 1!
6 | Indexierung
Ein Vektor x enthält 20 verschiedene Elemente.
7 | Indexierung
Ein Vektor x enthält 20 verschiedene Elemente.
8 | Indexierung
Ein Vektor vec enthält mehrere Zahlen.
Wir nutzen dazu den Modulo-Operator, welcher eine Restwert-Division zweier Elemente ausführt. Bei der Restwert-Division von 43 (Dividend) durch 5 (Divisor) wird z.B. gefragt, wie man die Zahl 43 als Vielfaches von 5 und einem kleinen Rest darstellen kann: \(43=5*c+r\) In diesem Beispiel wäre die 4 der sog. Ganzzahlquotient c und die 3 der Restwert r.
Ein Rest ungleich 0 ergibt sich folglich genau dann, wenn der Dividend kein Vielfaches des Divisors ist. Man sagt auch: Der Dividend ist nicht durch den Divisor teilbar, weshalb ein Rest übrigbleibt.
Dies können wir uns zunutze machen, indem wir als Divisor die 2 nehmen. Wenn kein Restbetrag übrig bleibt, muss der Dividend gerade sein!
9 | Indexierung
Ein Vektor vec enthält mehrere Zahlen.
10 | Indexierung
Ein numerischer Vektor nvec enthält eine unbekannte Anzahl an Elementen.
Abschlussquiz 11 | Indexierung
12 | Vektorisierte Berechnung
Der kürzere Vektor 1:3 wird durch das recyceln der eigenen Werte auf die gleiche Länge wie der erste Vektor gebracht (=6): c(1,2,3,1,2,3).
Nun wird das 5. Element (=2) mit dem 5.Element des 1. Vektors (=10) multipliziert.
13 | Vektorisierte Berechnung
Zur Erinnerung die Formel der Varianz: \(s^{2} = \frac{\sum\limits_{i=1}^{n} \left(x_{i} - \bar{x}\right)^{2}} {n-1}\)
df im Quiz steht für ‘degrees of freedom’, was im Deutschen die Freiheitsgrade sind. Und die betragen bei der Varianz n-1 (die Stichprobengröße oder Länge des Vektors minus 1).
Bei der Teilanweisung x-mean_x wird der eine Wert in mean_x auf die Länge von x recycelt. Anschl. wird der 1. Wert in mean_x vom 1. Wert in x, abgezogen, dann der zweite Wert, etc.
Das Ergebnis ist ein Vektor der gleichen Länge wie x mit den Differenzen, die anschl. quadriert und aufsummiert werden.

Bei weiteren Fragen: saskia.otto(at)uni-hamburg.de

Diese Arbeit is lizenziert unter einer Creative Commons Attribution-ShareAlike 4.0 International License mit Ausnahme der entliehenen und mit Quellenangabe versehenen Abbildungen.
Kurswebseite: Data Science 1