
Einführung in die (R) Programmierung
Data Science 1 - Programmieren & Visualisieren
Saskia Otto & Monika Eberhard
Universität Hamburg, IMF
Wintersemester 2025/2026
Lernziele
![]()
Am Ende dieser VL- und Übungseinheit werden Sie
- Begriffe wie Programm, Algorithmus, Quellcode, Syntax besser einordnen können.
- die 4 Grundelemente von (Objektorientierten) Programmiersprachen kennen.
- vertraut sein mit der GUI von RStudio/Posit,
- einfache Berechnungen mit arithmetischen Operatoren und built-in Funktionen durchführen können,
- wissen, wie man einzelne Werte als Variablen speichert und mit ihnen rechnet,
- einfache Abfragen mit Vergleichs- und Verknüpfungsoperatoren machen können.
- einen ersten Eindruck haben, wie die Calc-Analyse aus Woche 2 und 3 in R leicht umgesetzt werden kann!
Programmiersprachen

Was ist erstmal ein Programm?
- Ein Programm ist die Umsetzung eines Algorithmus und der zugehörigen Datenbereiche in eine Programmiersprache.
- Während Algorithmen relativ allgemein beschrieben werden können, sind Programme wesentlich konkreter.
Und was ist ein Algorithmus?
Ganz einfach …
…ein Algorithmus ist eine Reihe von Anweisungen, die Schritt für Schritt ausgeführt werden, um eine Aufgabe oder ein Problem zu lösen
Algorithmen im Alltag
Rezepte

Bauanleitungen
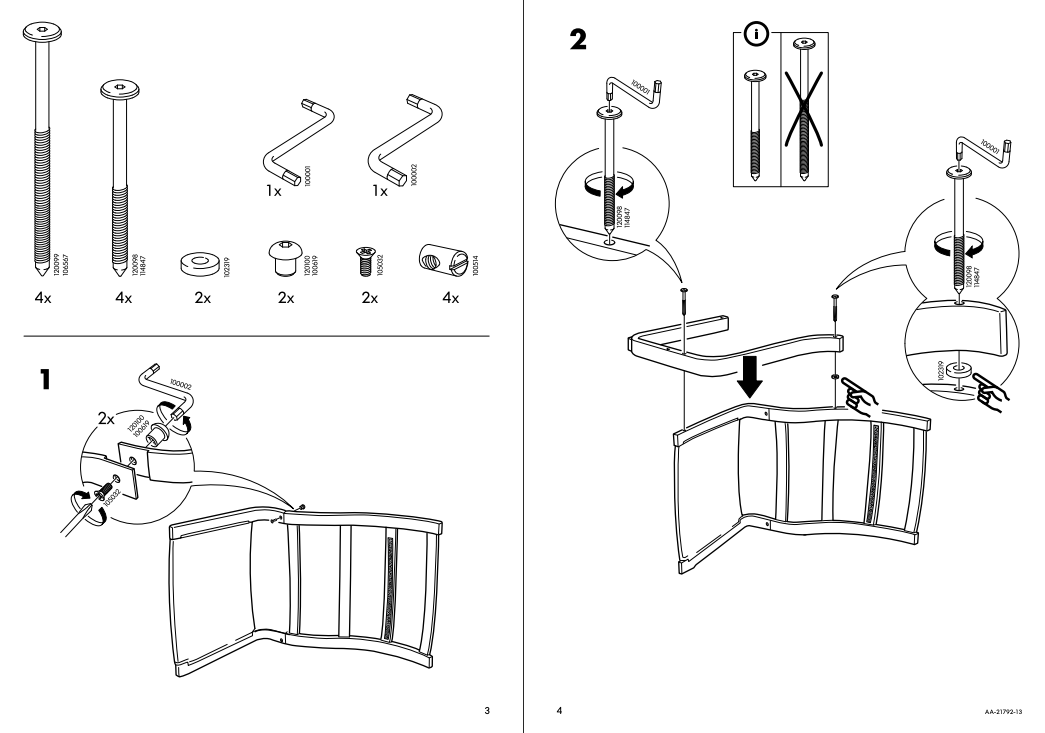
Bildquelle links: Sommer Vektor erstellt von freepik; Bildquelle rechts: Stuhlanleitung von IKEA
Swift Playgrounds App
Tipp zum Algorithmen lernen
Was sind Programmiersprachen?
Programmiersprachen sind formale Sprachen, mit denen sich Rechenvorschriften formulieren lassen. Diese Vorschriften können dann von einem Computer ausgeführt werden.
- Die Gesamtheit aller Anweisungen, die in einer bestimmten Programmiersprache geschrieben sind, bezeichnet man als Quellcode.
- Ein Quellcode lässt sich mit Hilfe eines einfachen Texteditors erstellen.
- Bei größeren Projekten setzt man meist eine Entwicklungsumgebung (IDE) ein.
- Die Syntax einer Programmiersprache ist deren “Grammatik”. Sie beschreibt sowohl die zulässigen Sprachelemente als auch, wie diese Sprachelemente zu verwenden sind.
Beispiel von Quellcode für Websites
HTML
CSS
Zusammen sieht das dann so aus…
Quellcode für Datenanalysen
R

Python

Welche Programmiersprachen werden in Data Science genutzt?
Viele

Ranking
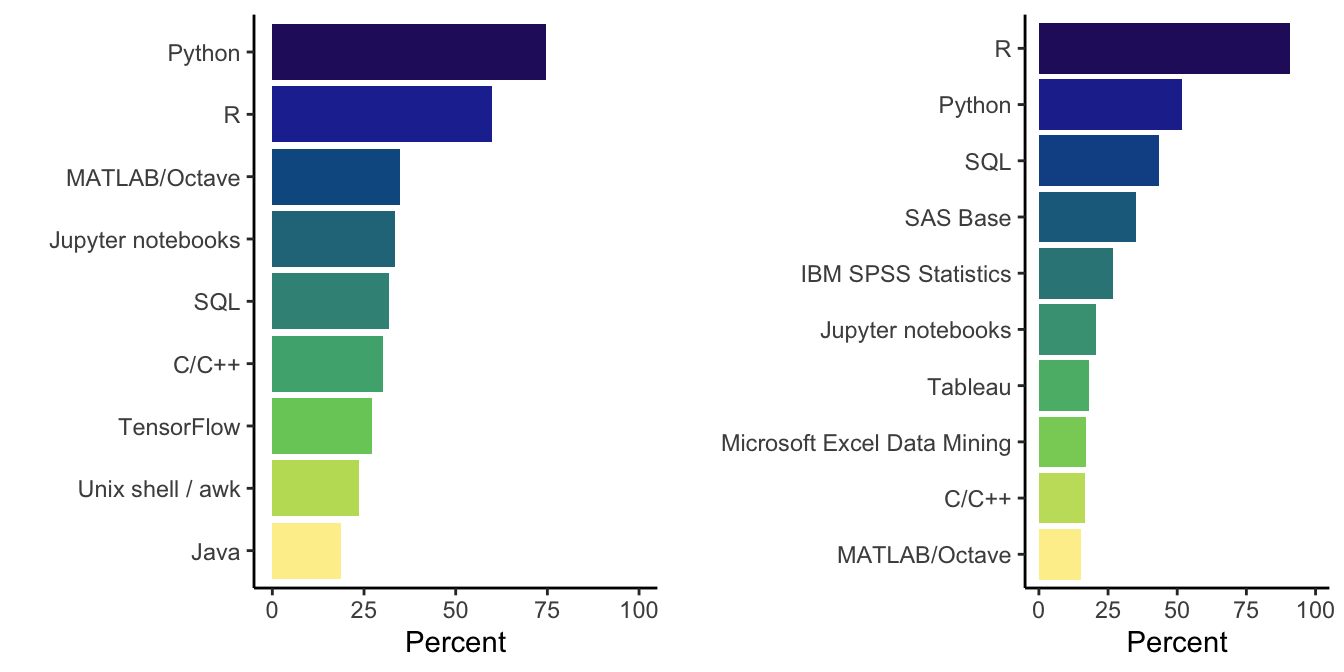
Säulendiagramm der häufigsten Programme und Sprachen bei akademischen ‘data scientists’ (links) and Statistikern (rechts).
Datenquelle: Kaggle Survey, 2017
Die Grundelemente
- von Objekt-orientierten Sprachen wie R, Python und Javascript

Variablen
- Eine Variable ist eine Möglichkeit, um im Programm eine Zahl, ein Zeichen oder Zeichenkette oder einen boolschen Wert (wahr oder falsch) zu speichern.
Beispiel
R
x <- 10JavaScript
var x = 10;Bedingungen
- Eine Bedingung ist eine Abfrage; trifft diese zu, so wird das, was innerhalb der Bedingung ist, ausgeführt.
- Um eine Bedingung zu erstellen, gibt es Operatoren (gleich, ungleich, größer als, kleiner als, wahr, falsch).
Beispiel
R
if (Bedingung) {
ausführen
}if (x < 20) {
print("Stimmt, x ist kleiner als 20!")
}JavaScript
if (Bedingung) {
ausführen;
}if (x < 20) {
alert("Stimmt, x ist kleiner als 20!");
}Schleifen
- Eine Schleife enthält Code der so lange ausgeführt wird, wie die Bedingung, die zur Schleife gehört, wahr ist.
- Der Code kann also z.B. zehn Mal ausgeführt werden, das hat den Vorteil, dass der Code nicht zehn Mal geschrieben werden muss.
Beispiel
R
for (i in 1:10) {
print(i)
}JavaScript
for (i=0; i < 10; i++) {
alert(i);
}Funktionen
- Eine Funktion ist eine Sammlung von Code, die jederzeit über den jeweiligen Funktionsnamen aufgerufen werden kann.
- Sie können auch Parameter enthalten.
- Dies sind Werte, wie z.B. eine Zahl oder ein String. Diese Zahl kann von der Funktion wie eine Variable verwendet werden.
Beispiel
R
sag_hallo <- function(name) {
print(paste("Hallo", name))
}
sag_hallo("Jan")
sag_hallo("Lena")JavaScript
function sagHallo(name) {
alert("Hallo " + name);
}
sagHallo("Jan");
sagHallo("Lena");Einführung in R und Entwicklungsumgebungen
Was ist R?

“R is a system for statistical computation and graphics. It is a GNU project which is similar to the S language and environment which was developed at Bell Laboratories by John Chambers and colleagues. R can be considered as a different implementation of S…..R is available as Free Software under the terms of the Free Software Foundation’s GNU General Public License in source code form. It compiles and runs on a wide variety of UNIX platforms, Windows and MacOS.” | aus: http://r-project.org/
Link Youtube video: R The most powerful and most widely used statistical software
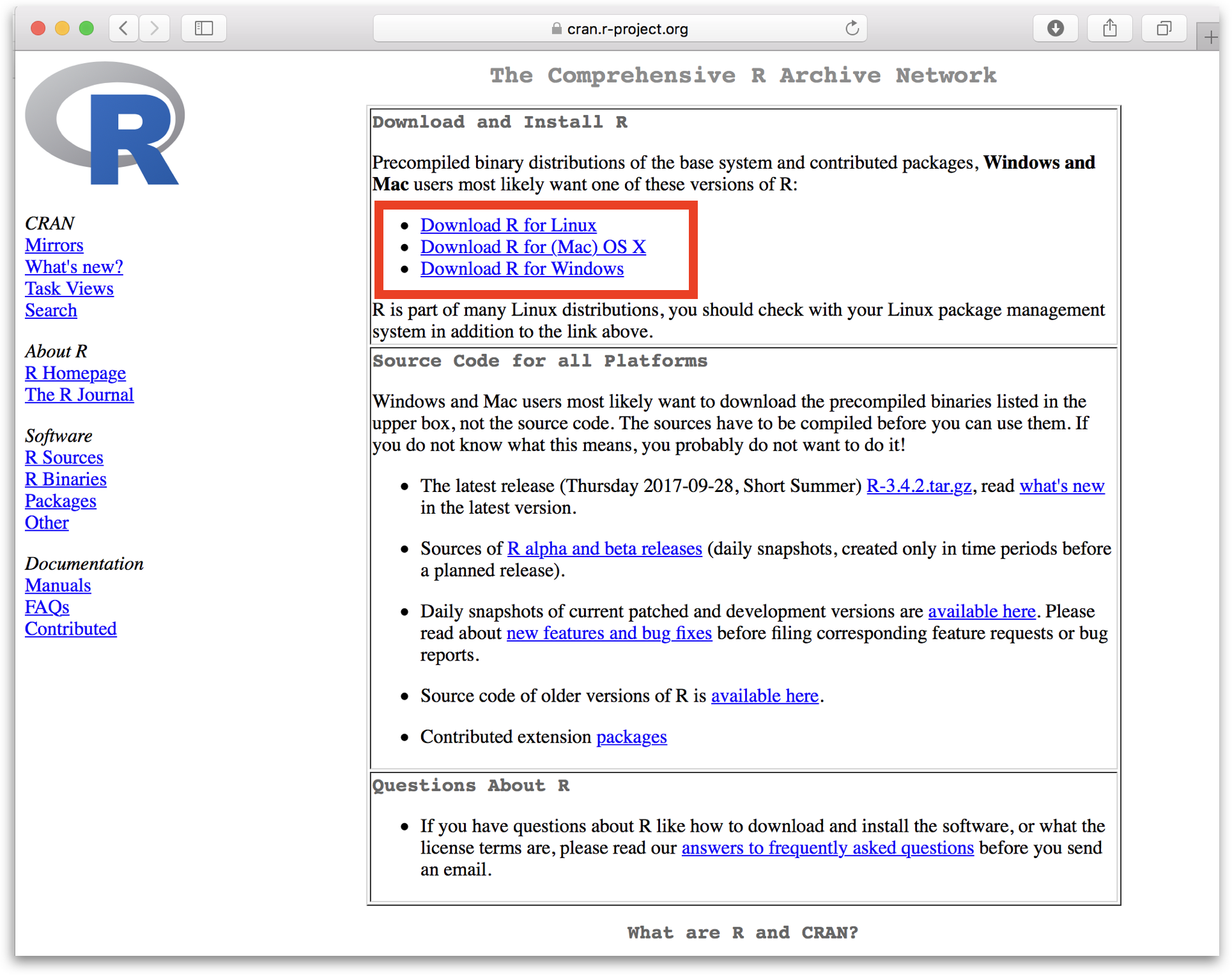
Wo gibt es R?
Direkt auf der Website https://cran.r-project.org
Editoren und Entwicklungsumgebungen
- R kommt nur mit einer sehr einfachen Nutzeroberfläche daher, die einen Einstieg in die Sprache nicht leicht macht.
- Es wird daher empfohlen, zur Ausführung von R geeignete Editoren oder integrierte Entwicklungsumgebung (IDE) zu verwenden:
- RCommander → eine der ersten IDEs
- RStudio → jetzt Posit → die IDE für R schlechthin
- Jupyter Notebook
- Visual Studio Code → Quelltext-Editor für eine Vielzahl von Programmier- und Auszeichnungssprachen; beliebt bei Web-Entwicklern, jetzt auch mit R-Plugin
- PyCharm → ursprünglich für Python, jetzt auch mit R-Plugin
Umfrage bei R Nutzern in 2020/2021
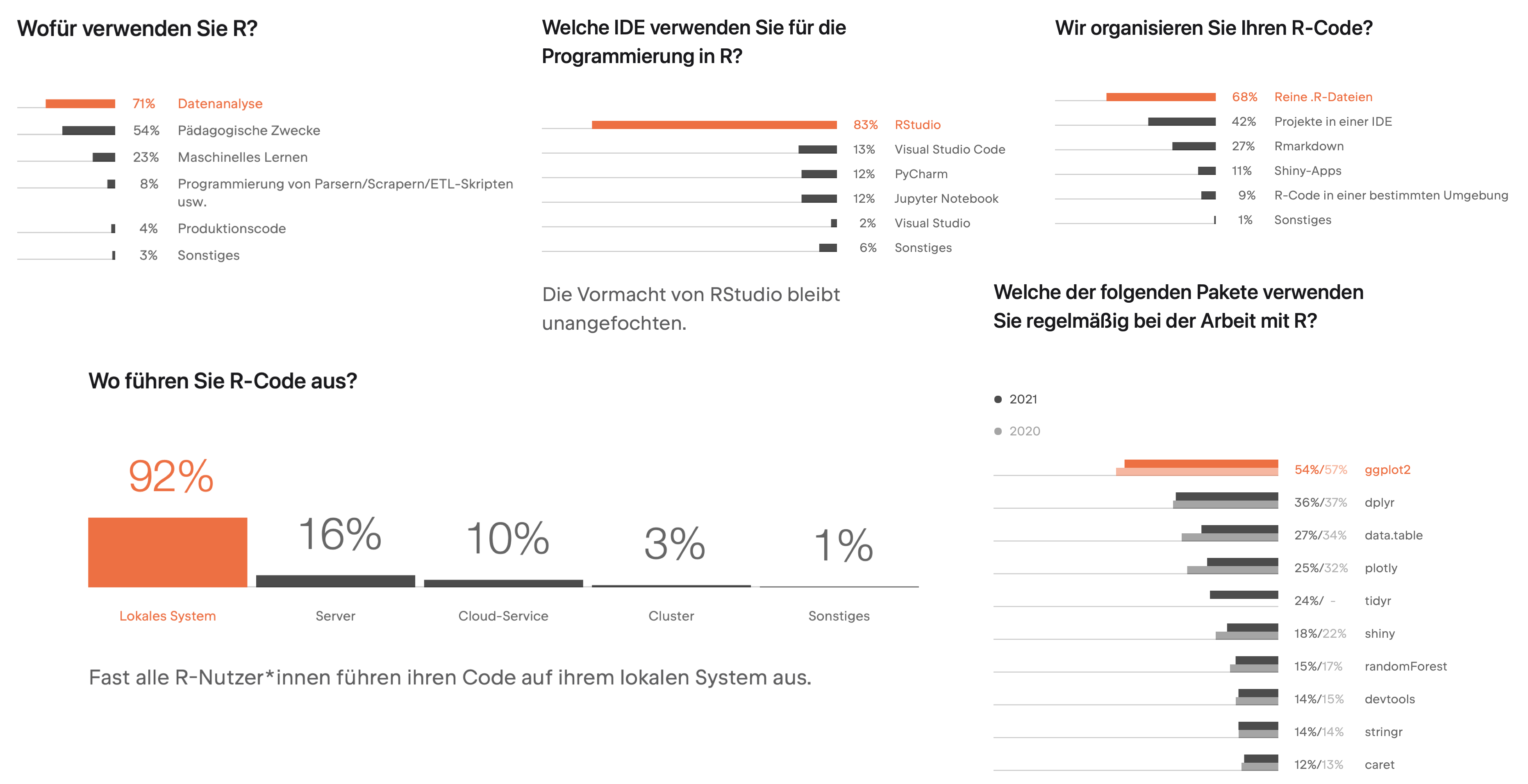
Link zur Website: https://www.jetbrains.com/de-de/lp/devecosystem-2021/r/
Standard bei R Usern

- Eine Software mit einer Entwicklungsumgebung für R.
- Hilft R Code zu schreiben und auszuführen und Daten zu analysieren.
- Integriert einen Texteditor mit einem Daten- und Paketmanager
- Bietet Versionskontrollen, LaTeX Integration, Tastenkombinationen, und sog. ‘debugging’ tools zur Fehlerdiagnose.
- Code in C, C++ oder Fortran kann kompiliert und direkt eingebunden werden.
- Markdown-basierte Dokumente werden mit Hilfe von knitr oder Sweave in Berichte, Webseiten oder Präsentationen umgewandelt → hier können R, Python, SQL, Stan, Julia, C und Fortran kombiniert werden.
- Es gibt die Software als frei verfügbare und als kommerzielle Version auf posit.co für Windows, Mac und Linux.
RStudio/Posit Workbench
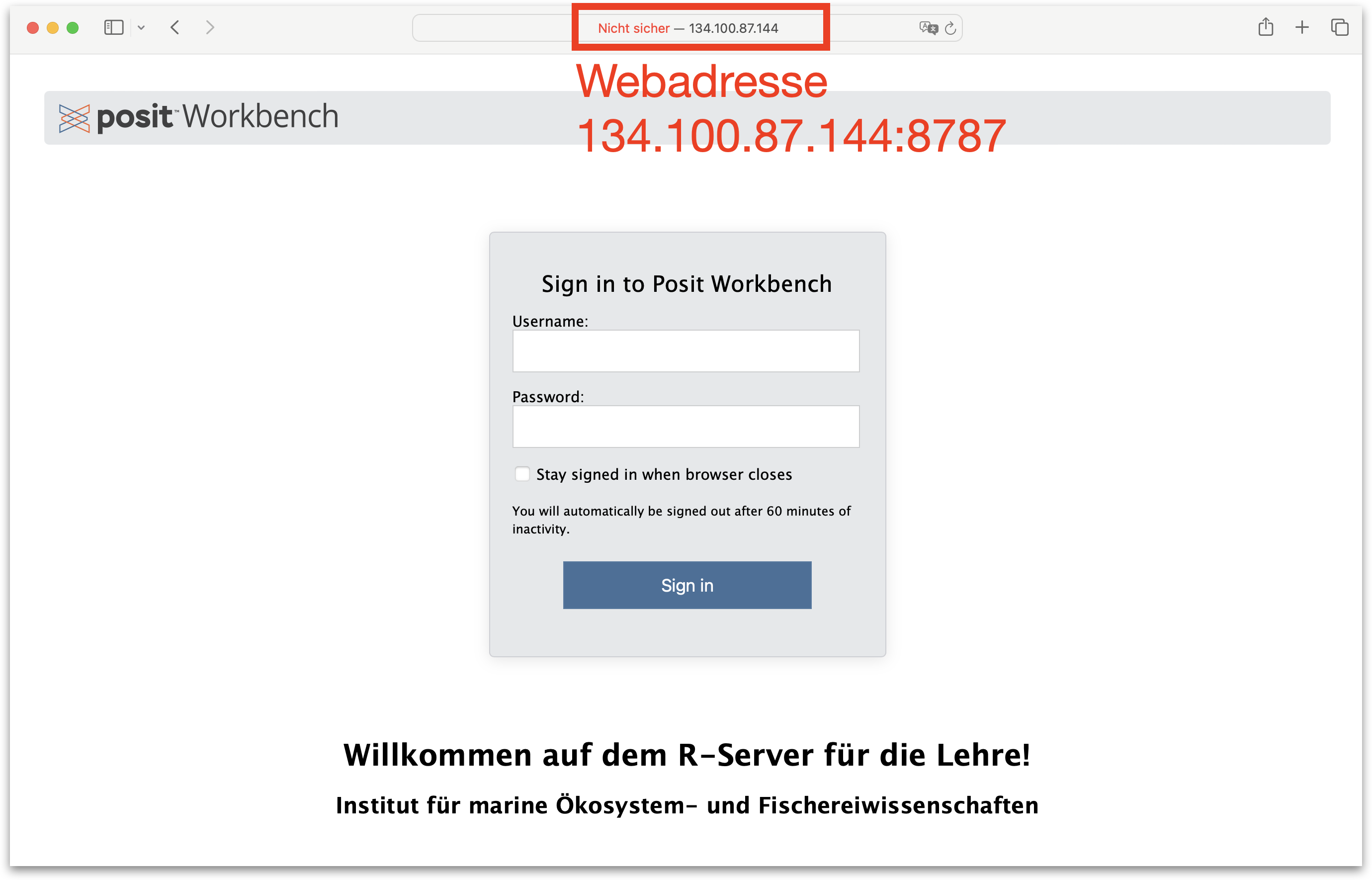

Link zur Webadresse der Workbench
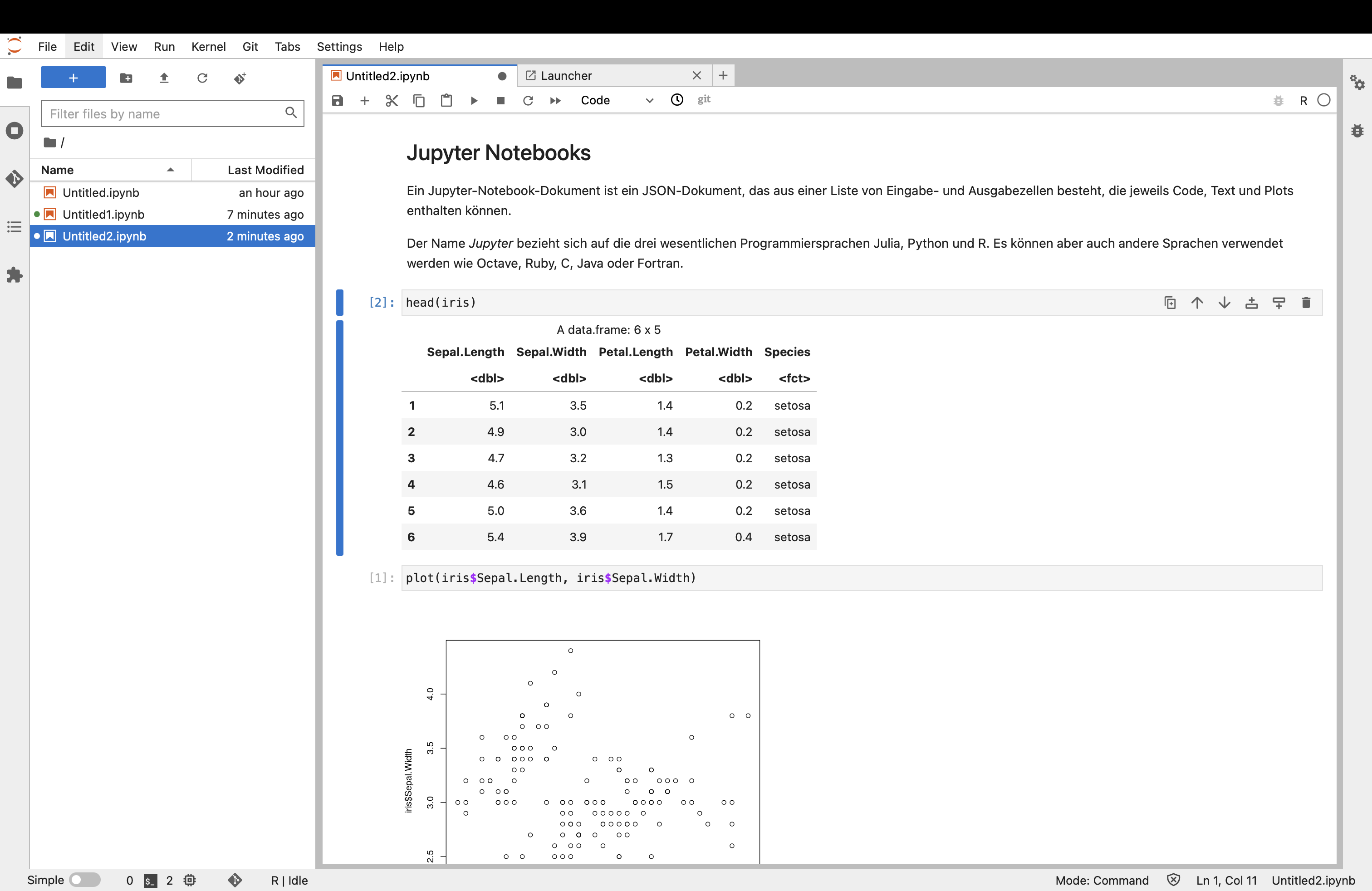
Alternativ: Jupyter Notebooks
![]()

- Zum Arbeiten mit Notebooks bietet sich auch Jupyter Notebooks und die neue Benutzeroberfläche JupyterLab an: https://jupyter.org/
- Die MIN Fakultät der Uni Hamburg bietet die Servervariante JupyterHub an:
- https://code.min.uni-hamburg.de/hub
- Hier kann man sich ganz einfach mit der BXX1234-Kennung einloggen.

NEU: RStudio über JupyterHub Server
- RStudio kann jetzt auch über den Server der MIN Fakultät ‘gelaunched’ werden:

Die RStudio/Posit Umgebung

Wie ist RStudio aufgebaut?
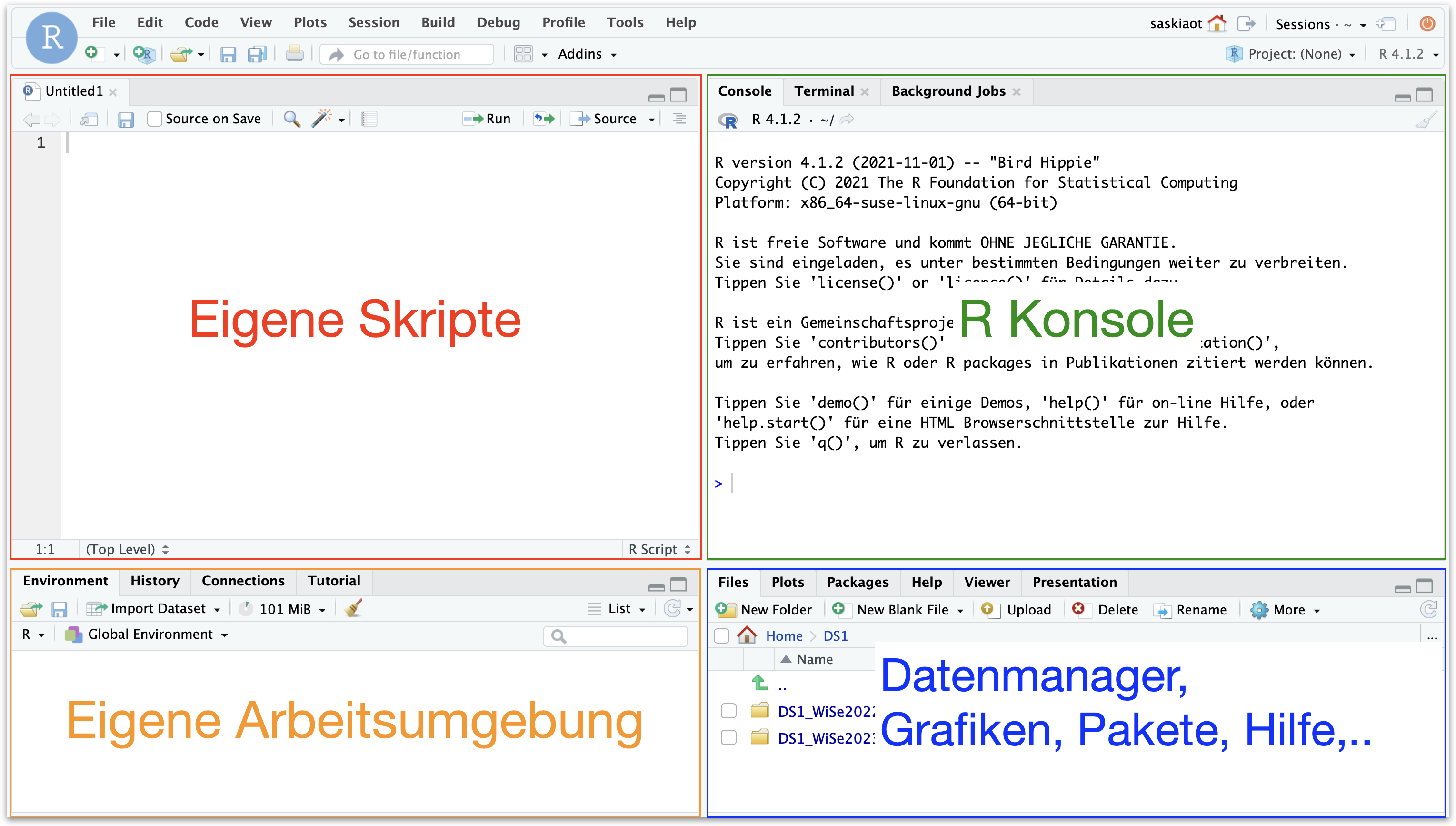
Die 4 Fenster
Skripte
- Für ganze Datenanalysen sollten IMMER Skripte verwendet werden.
- Skripte sind simple Textdateien, die Deinen R Quellcode enthalten.
- Neben ausführbarem Code kann man auch Kommentare als nicht ausführbaren Code schreiben (um den Code zu erklären). Diese werden in R mit einem ‘#’ vorweg angegeben.
R Konsole
- Das Kernstück von R
- Hier werden Befehle eingegeben und Ergebnisse ausgegeben.
- Für schnelle Berechnungen kann gerne nur die Konsole genutzt werden (ohne Skripte).
Arbeitsumgebung
- Die Arbeitsumgebung zeigt die eigenen gespeicherten Funktionen und Objekte an.
- Sie ist bei jeder neuen Session anfangs leer.
Datenmanager
- Anstelle Dateien im Windows Explorer zu suchen und öffnen, kann direkt der Datenmanager genutzt werden.
- Am besten ist, wenn das Verzeichnis, welches im Datenmanager zu sehen ist, auch das Arbeitsverzeichnis von R.
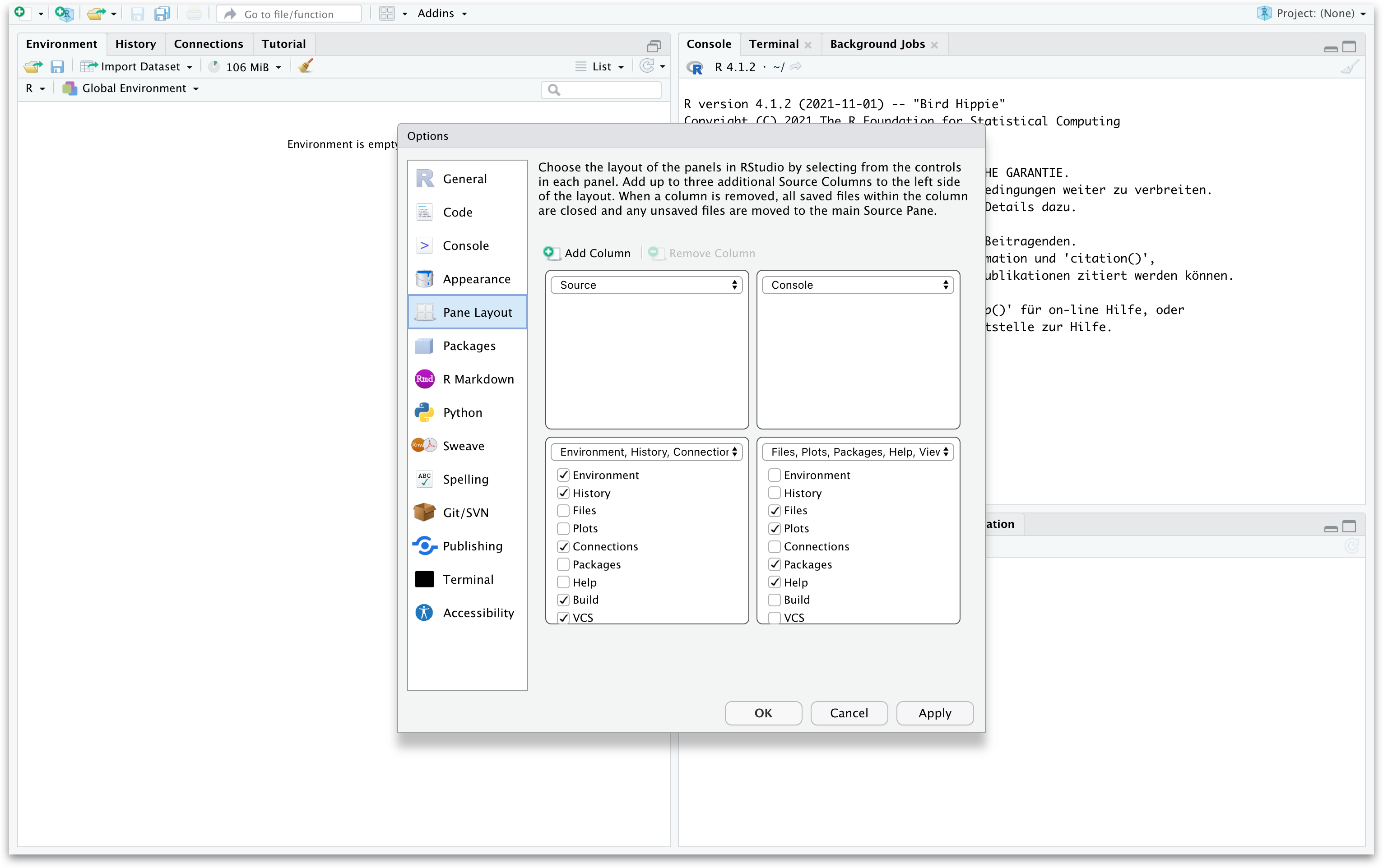
Anpassen der Fenster
Über Tools > Global Options > Pane Layout:
Skripte | Struktur und Vorteile
Struktur
Vorteile
- Transparenz und Reproduzierbarkeit - Nicht nur die Resultate sind einsehbar, sondern jeder einzelne Schritt der Analyse.
- Flexibilität - Einige Analysen brauchen nur ein paar kleine Veränderungen bestehender R Skripte.
- Austausch - Theoretisch sollte jeder R User ein Skript verstehen, so dass Code leichter austauschbar ist.
Mit Skripten in RStudio/Posit arbeiten
- Öffnen eines neues Skripts: File ➔ New File ➔ R Script
- SOFORTIGES Speichern des Skripts: File ➔ Save As
- In das leere Skript Code schreiben, z.B.
2+2 - Senden des Codes an die R Konsole:
- Codeschnipsel kopieren und in die Konsole einfügen (NICHT zu empfehlen!)
- ctrl+enter drücken➔ Ausführung des Codes der entsprechenden Zeile (wo der Cursor gerade ist); anschl. springt der Cursor eine Zeile weiter.
- Markieren des Codeabschnitts (können mehrere Zeilen sein), der ausgeführt werden soll, und ctrl+enter drücken bzw. Auswahl des Run buttons:
![]()
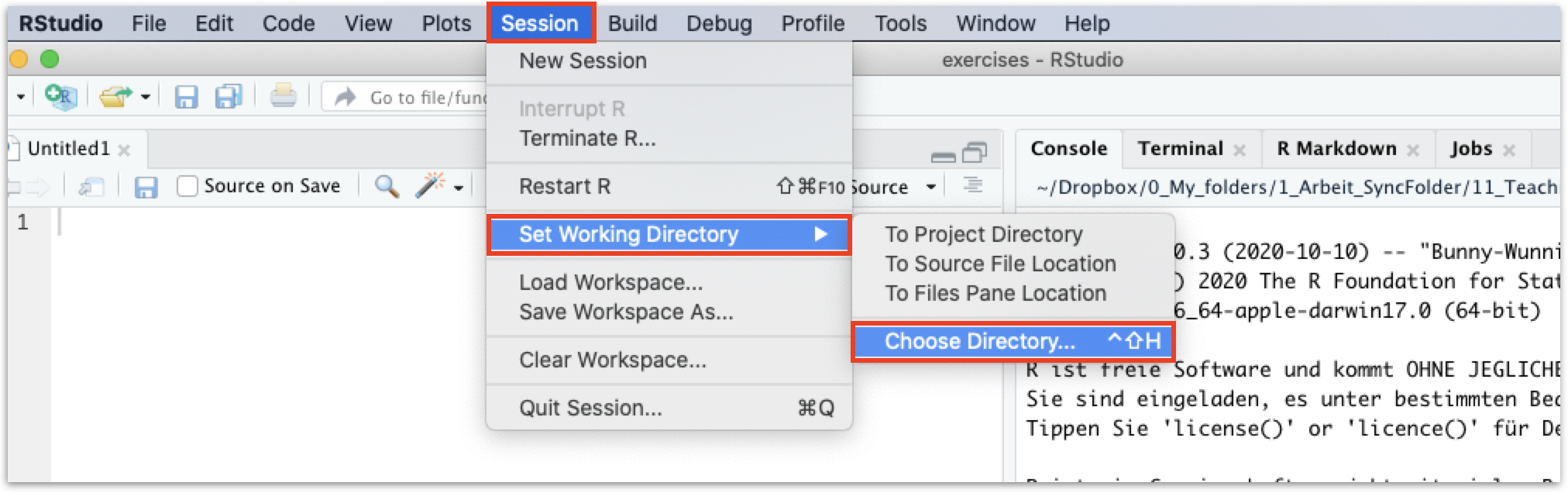
Das Arbeitsverzeichnis
- Das Arbeitsverzeichnis ist ein Dateipfad auf Ihrem Computer.
- Dieser Pfad legt den standardmäßigen Speicherort aller Dateien fest, die Sie in R einlesen oder aus R heraus speichern möchten.
- Gewöhnen Sie sich an, jedes Mal bevor Sie loslegen, das Arbeitsverzeichnis festzulegen!
- Dafür gibt es mehrere Wege, merken Sie sich aber erstmal nur diesen hier:
Empfehlung: R Projekte
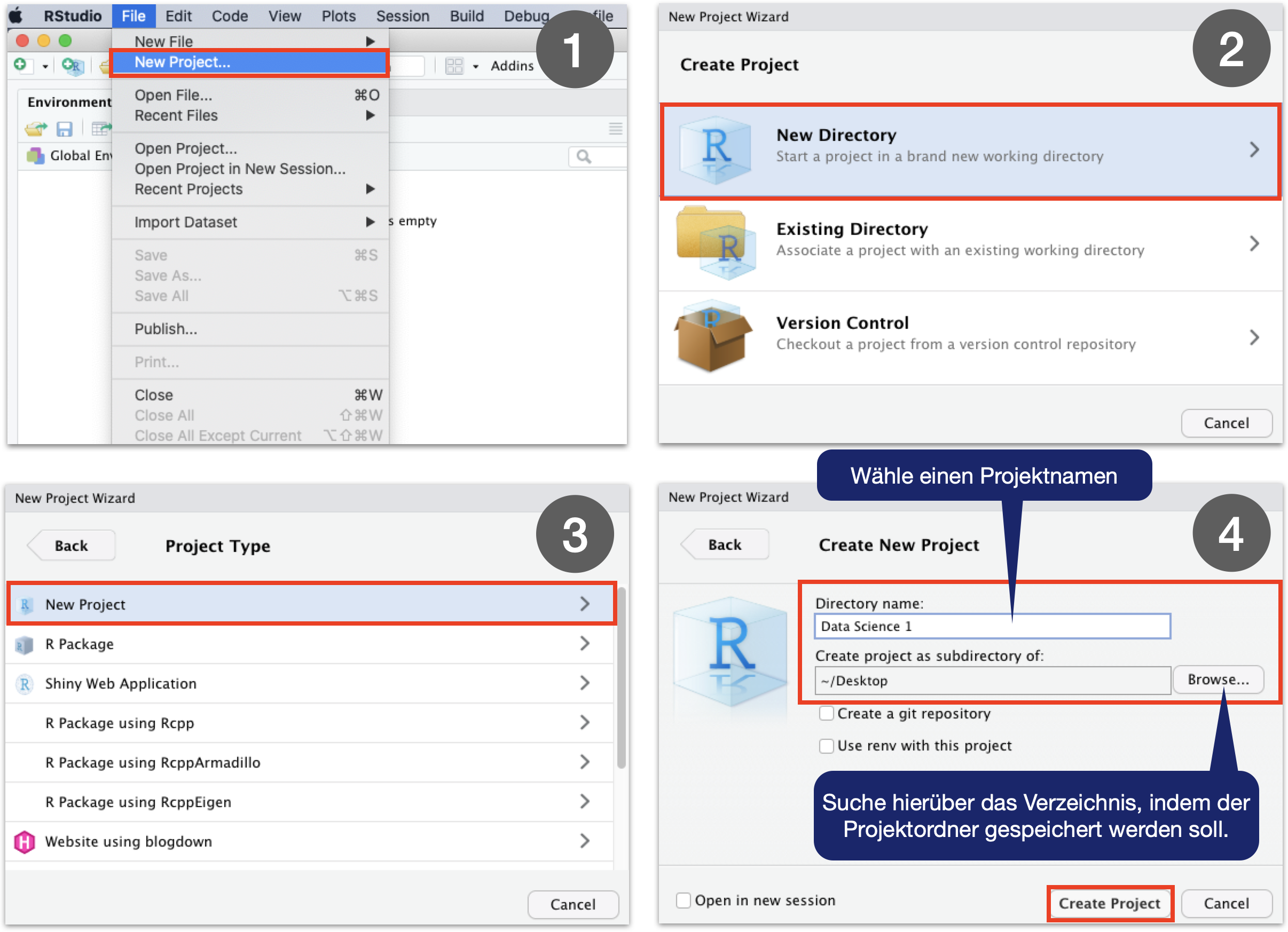
R Projekte
Neue Ordnerstruktur
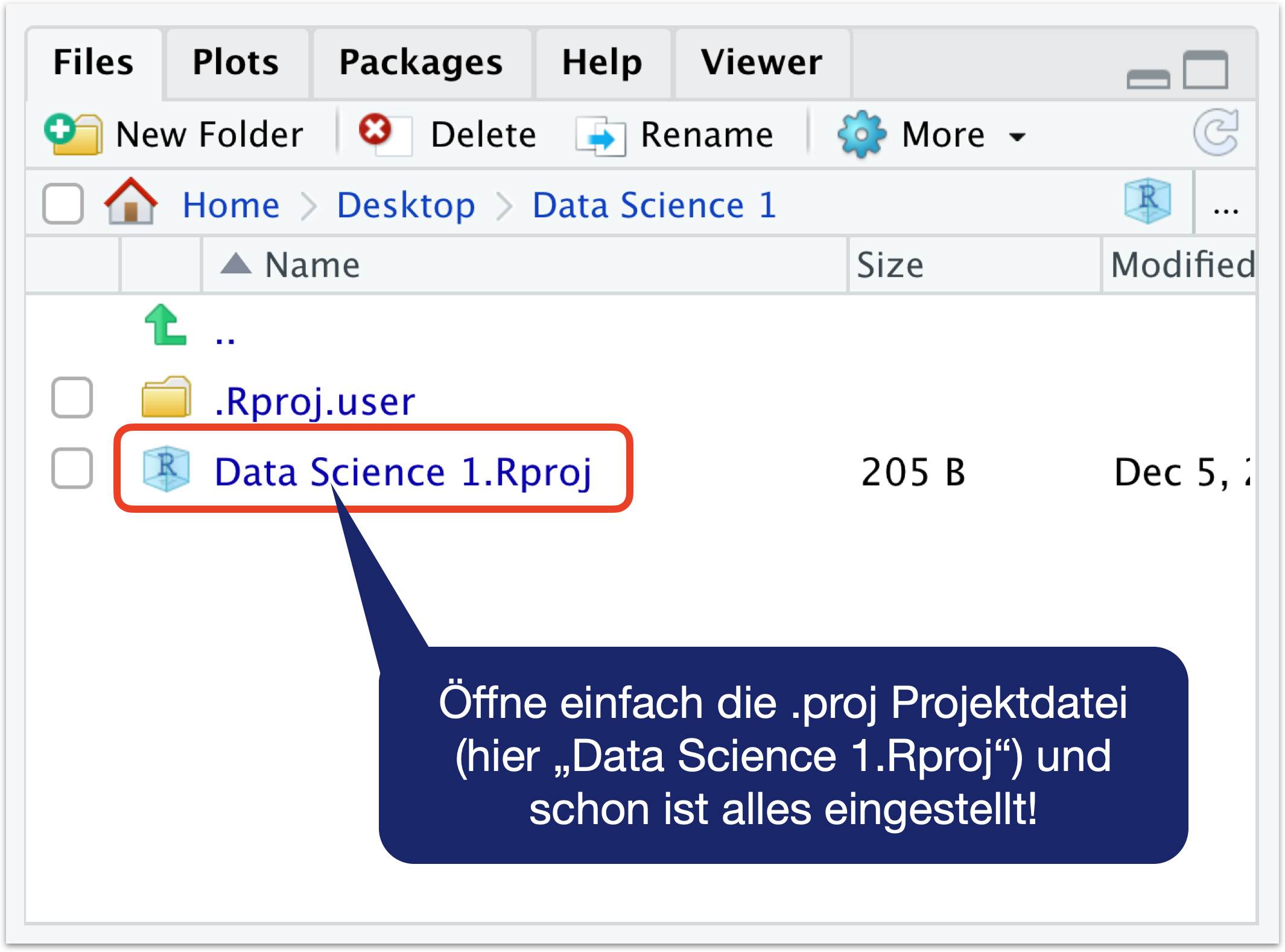
Vorteile von Projekten
- Vordefinierte Ordnerstruktur..
- Das Arbeitsverzeichnis wird automatisch gesetzt
- Alle Skripte in diesem Projekt sind sofort verfügbar.
- Öffnet eine neue R Instanz so dass man zwischen verschiedenen Instanzen wechseln kann.
Rechnen in R

R als simpler Taschenrechner
In seiner einfachsten Form ist R vergleichbar mit einem einfachen Taschenrechner.
Einfache Berechnungen
Folgende arithmetische Operatoren gibt es:
Rechnen mit Funktionen

R hat aber auch Funktionen mit denen man anspruchsvollere Manipulationen durchführen kann, die durch Klammern kombinierbar sind:
Tip
Reihenfolge der Berechnung: von der innersten zur äußersten Klammer:
- Funktionen
sqrt(9)undexp(2)(sqrt = square root, exp = exponent) ausführen, - dann
12zum Ergebnis vonsqrt(9)und1zum Ergebnis vonexp(2)addieren, - dann teilen.
+ prompt
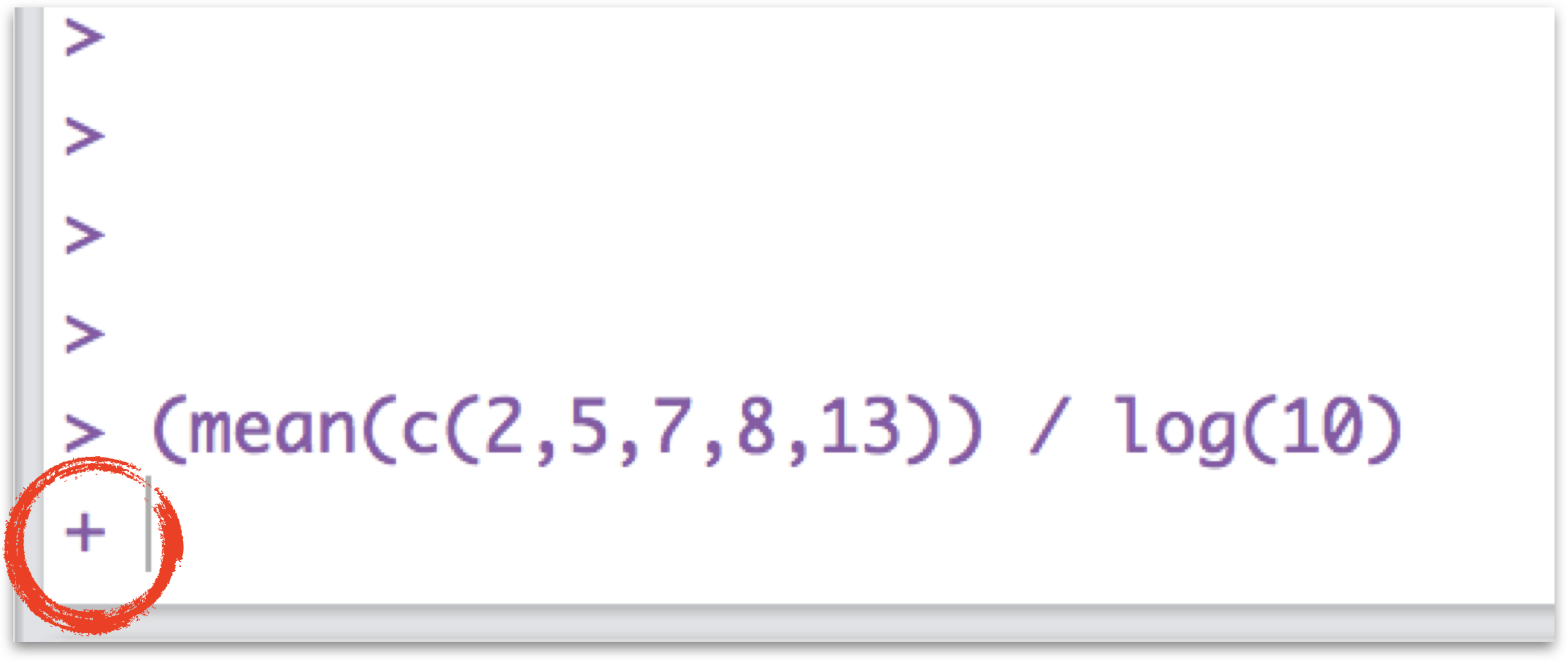
Wenn das sog. prompt Zeichen von “>” zu “+” wechselt, ist das ein Zeichen, dass der Befehl noch nicht zu Ende ist. Meist fehlen eine oder mehrere schließende Klammern. Führe entweder den Befehl zu Ende oder drücke (2mal) auf esc.
Built-in Funktionen und Pakete

Eine kurze Einführung
- Einige Funktionen sind in den Basispaketen von R enthalten, andere sind erst durch die Installation und das Laden bestimmter Zusatzpakete verfügbar.
- R Funktionen arbeiten ähnlich wie Funktionen in anderen Programmen:

Dokumentation (Hilfe) aufrufen
Jede Funktion hat seine eigene Hilfe die mit help() oder ? aufgerufen werden kann:
R Dokumentation für mean() | 1
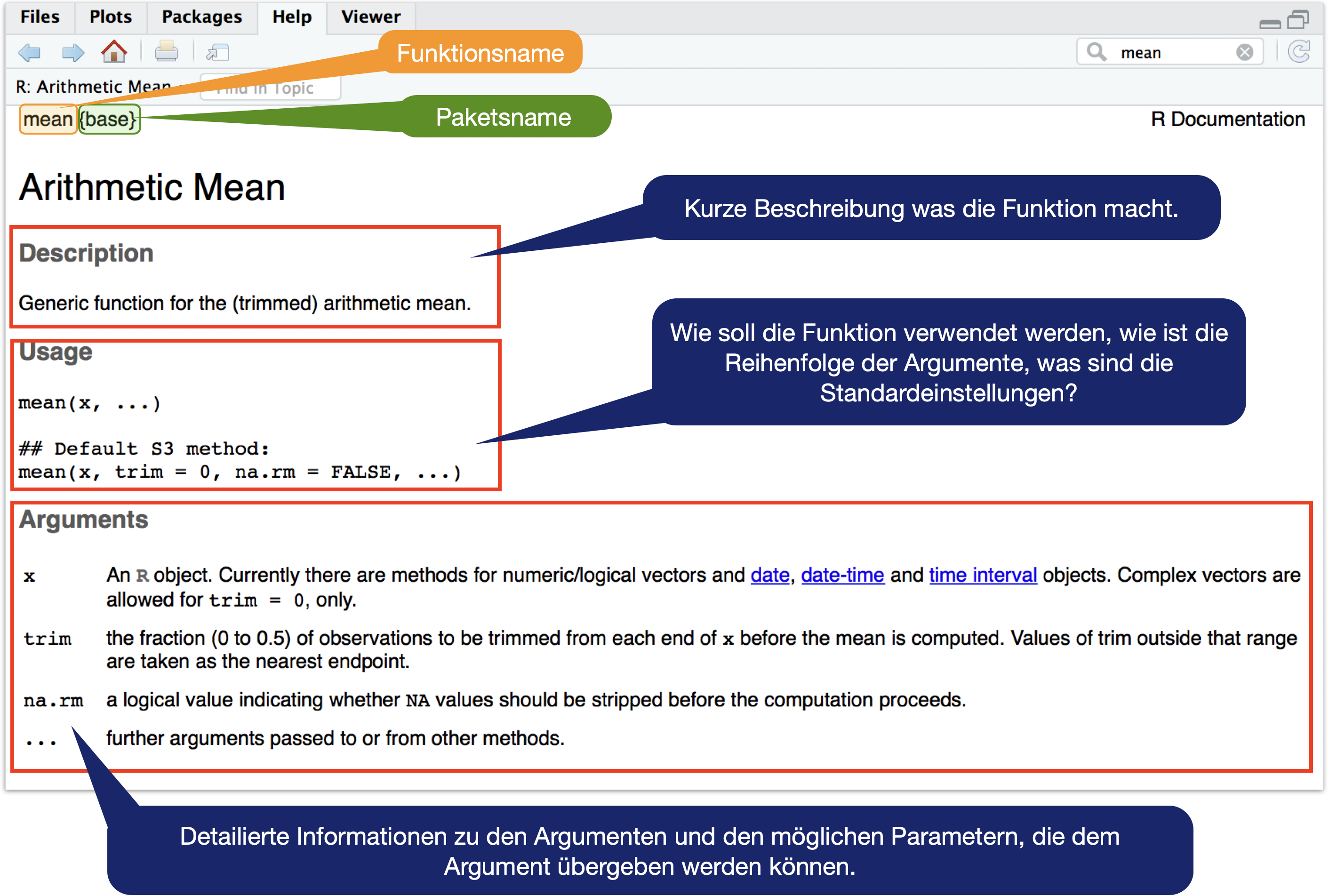
R Dokumentation für mean() | 2

Pakete für mehr Funktionalität
- Pakete werden von der R Gemeinschaft geschrieben.
- Sind sind eine Sammlung von
- R Funktionen,
- der Dokumentation, die beschreibt wie die Funktionen anzuwenden sind,
- und oft auch Beispieldaten.
- Die offizielle CRAN package Webseite enthält aktuell 18786 (!) Pakete (Stand 2025-09-26).
Entwicklung veröffentlichter Pakete
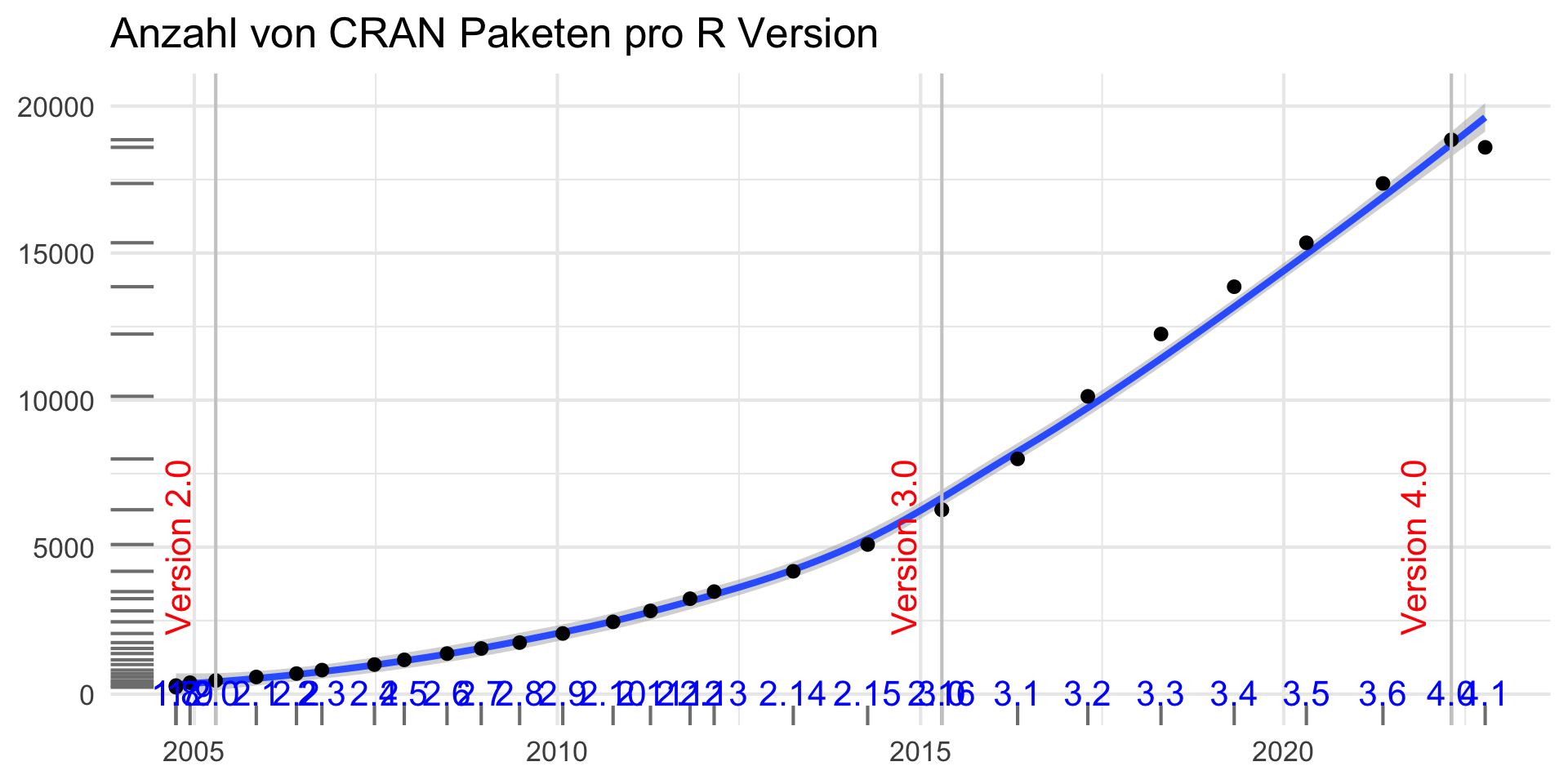
Dieses Diagramm basiert auf einer Modifikation des Codes von Andrie de Vries Blog Post.
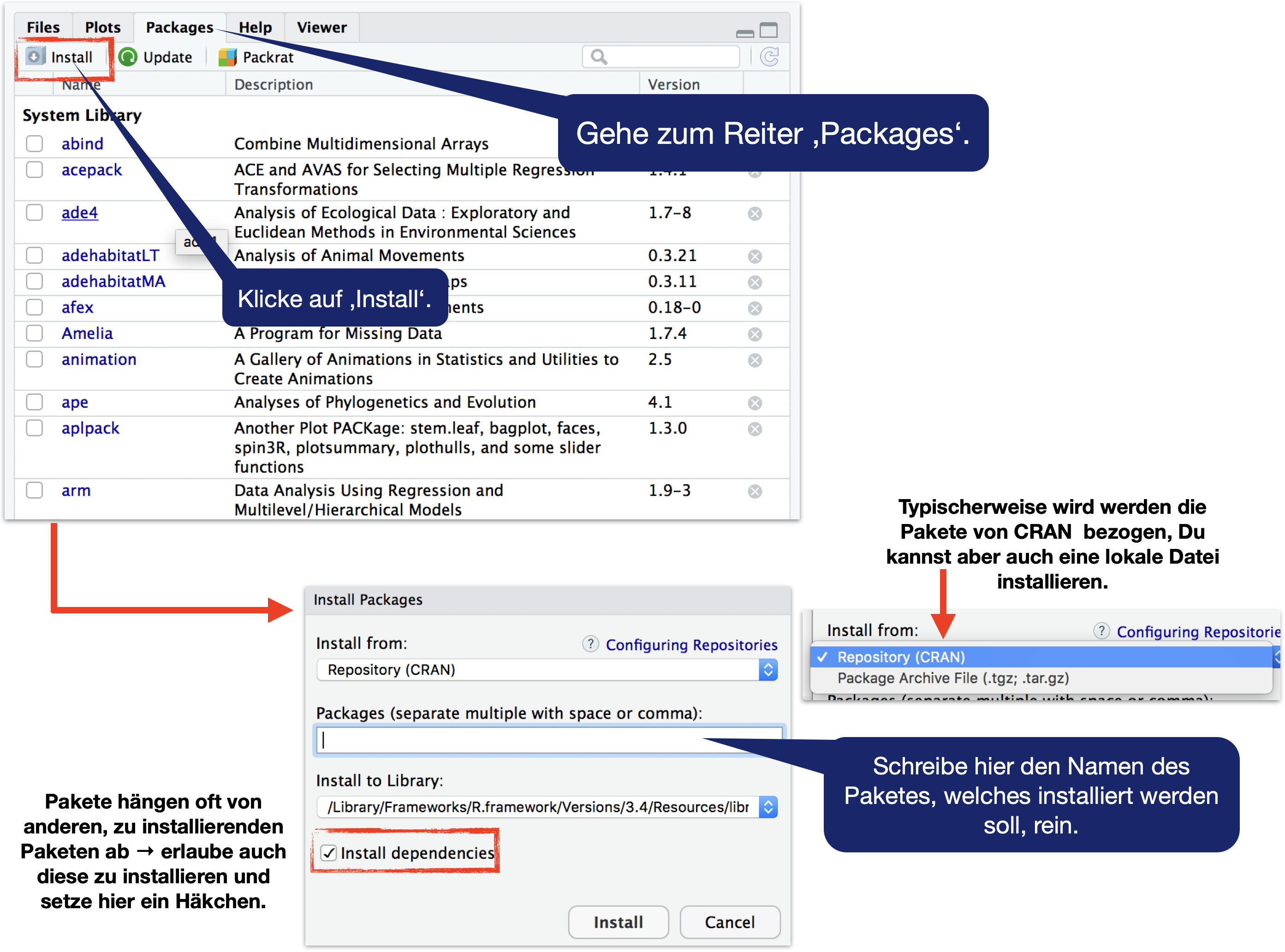
Installation eines Pakets (EINMALIG) | 1
Mittels Funktion
Die von CRAN offiziell anerkannten Versionen können direkt über die Funktion install.packages() installiert werden:
Installation eines Pakets (EINMALIG) | 2
Aus RStudio/Posit heraus
Laden eines Pakets (JEDE Sitzung)
Ein Paket muss für jede Sitzung geladen werden, um auf die Funktionen zugreifen zu können. Dazu gibt es die Funktion library(). R überprüft dann erstmal, ob dieses Paket auch installiert ist.
Wie kann man Pakete aus der Sitzung deaktivieren?
Pakete werden einfach mit detach() deaktiviert …
.. oder indem das Häkchen neben dem Paketnamen im ‘Packages’ Reiter entfernt wird:

Your turn …
![]()
Quiz
Einfache Berechnungen
- Loggen Sie sich bei der RStudio Workbench ein und machen Sie sich mit der GUI vertraut.
- Führen Sie anschl. folgende Berechnungen durch und vergleichen Sie Ihre Ergebnisse mit den Lösungen:
- Subtrahieren Sie 10 von 23 und multiplizieren Sie das Ergebnis mit 2.
- Subtrahieren Sie 10 von 23, multiplizieren Sie das Ergebnis mit 2, addieren Sie dann 100 dazu und Sie das Ergebnis durch 5.
- Subtrahiere Sie 23 von 10, multiplizieren Sie das Ergebnis mit -10 und nehmen Sie dann davon die Quadratwurzel (verwenden Sie die Funktion
sqrt()).
Link zur Webadresse der Workbench
Live-Demo in R

Von Calc zu R wechseln | Demo A
Lineare Regression zum Fütterungsversuch beim Kabeljau | 1
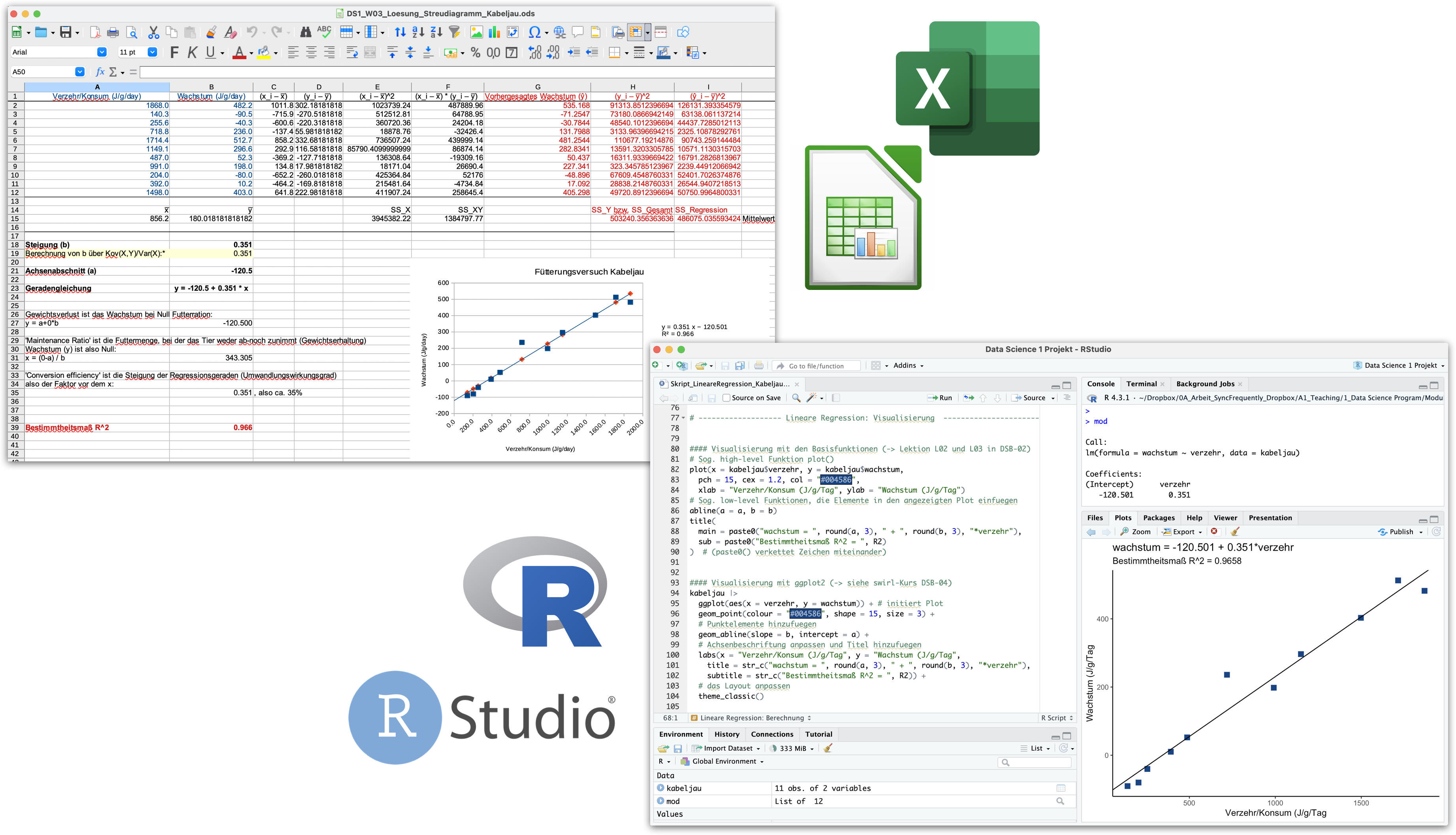
Beispiel der Übungsaufgabe aus Woche 3
Von Calc zu R wechseln | Demo A
Lineare Regression zum Fütterungsversuch beim Kabeljau | 2
![]()
Das vollständige R Skript (DS1_VL04a_Demo_LineareRegression_Kabeljau.R)
# ------------------------------ Vorbereitung ----------------------------------
#### Laden von Paketen
library(tidyverse)
# library(readODS)
# -------------------- Import und Datenaufbereitung ----------------------------
#### Import einer Textdatei im CSV-Format (das gaengigste Format)
kabeljau_csv <- read.csv(file = "Wachstum-Futter.csv")
str(kabeljau_csv) # --> output ist ein data frame
#### Import von Calc- und Excel-Dateien (ODS- und XLSX-Format)
# Import einer ODS-Datei mit dem 'readODS' Paket
kabeljau <- readODS::read_ods("DS1_W03_Streudiagramm_Kabeljau.ods",
sheet = "Daten_Visualisierung")
str(kabeljau) # --> output ist ein tibble
# Import einer XLSX-Datei mit z.B. dem 'readxl' Paket
# kabeljau_xlsx <- readxl::read_excel("DS1_W03_Streudiagramm_Kabeljau.xlsx",
# sheet = "Daten_Visualisierung")
# str(kabeljau_xlsx) # --> output ist ein tibble
#### Datensichtung und -transformation
# Anpassen der Spaltennamen
names(kabeljau) <- c("verzehr", "wachstum")
str(kabeljau)
# Wertebereich pruefen
summary(kabeljau)
# ---------------------- Lineare Regression: Berechnung ------------------------
# (-> Swirl-Lektion L03 in DSB-02-Datenexploration mit R)
#### Manuelle Berechnung
# Um Tipparbeit zu sparen, speichern wir die Spalten als einzelne Vektoren
x <- kabeljau$verzehr
y <- kabeljau$wachstum
# Steigungsparameter b berechnen
b <- cov(x = x, y = y)/ var(x) # der shortcut mit der Kovarianz und Varianz
b
# Achsenabschnitt a berechnen
a <- mean(y) - b*mean(x)
a
# Das Bestimmtheitsmass R^2 berechnen
y_obs <- a + b*x # die vorhergesagten Werte
ss_gesamt <- sum( (y - mean(y))^2 ) # Summenquadrate Gesamt
ss_regression <- sum( (y_obs - mean(y))^2 ) # Summenquadrate der Regression
R2 <- round(ss_regression/ss_gesamt, 4)
R2
##### Zum Vergleich die Regression automatisch berechnen mit lm()
# Erstellung des Modells
mod <- lm(formula = wachstum ~ verzehr, data = kabeljau)
mod
# Ausgabe nur der beiden Koeffizienten
coef(mod)
# Ausgabe aller wichtigen Statistiken des Modells, inklusive von R^2
# (mehr dazu in Data Science 2)
summary(mod)
# ------------------- Lineare Regression: Visualisierung ----------------------
#### Visualisierung mit den Basisfunktionen (-> Lektion L02 und L03 in DSB-02)
# Sog. high-level Funktion plot()
plot(x = kabeljau$verzehr, y = kabeljau$wachstum,
pch = 15, cex = 1.2, col = "#004586",
xlab = "Verzehr/Konsum (J/g/Tag", ylab = "Wachstum (J/g/Tag")
# Sog. low-level Funktionen, die Elemente in den angezeigten Plot einfuegen
abline(a = a, b = b)
title(
main = paste0("wachstum = ", round(a, 3), " + ", round(b, 3), "*verzehr"),
sub = paste0("Bestimmtheitsmaß R^2 = ", R2)
) # (paste0() verkettet Zeichen miteinander)
#### Visualisierung mit ggplot2 (-> siehe swirl-Kurs DSB-04)
kabeljau |>
ggplot(aes(x = verzehr, y = wachstum)) + # initiert Plot
geom_point(colour = "#004586", shape = 15, size = 3) +
# Punktelemente hinzufuegen
geom_abline(slope = b, intercept = a) +
# Achsenbeschriftung anpassen und Titel hinzufuegen
labs(x = "Verzehr/Konsum (J/g/Tag", y = "Wachstum (J/g/Tag",
title = str_c("wachstum = ", round(a, 3), " + ", round(b, 3), "*verzehr"),
subtitle = str_c("Bestimmtheitsmaß R^2 = ", R2)) +
# das Layout anpassen
theme_bw()Von Calc zu R wechseln | Demo B
Deskriptive Statistik mit iris | 1
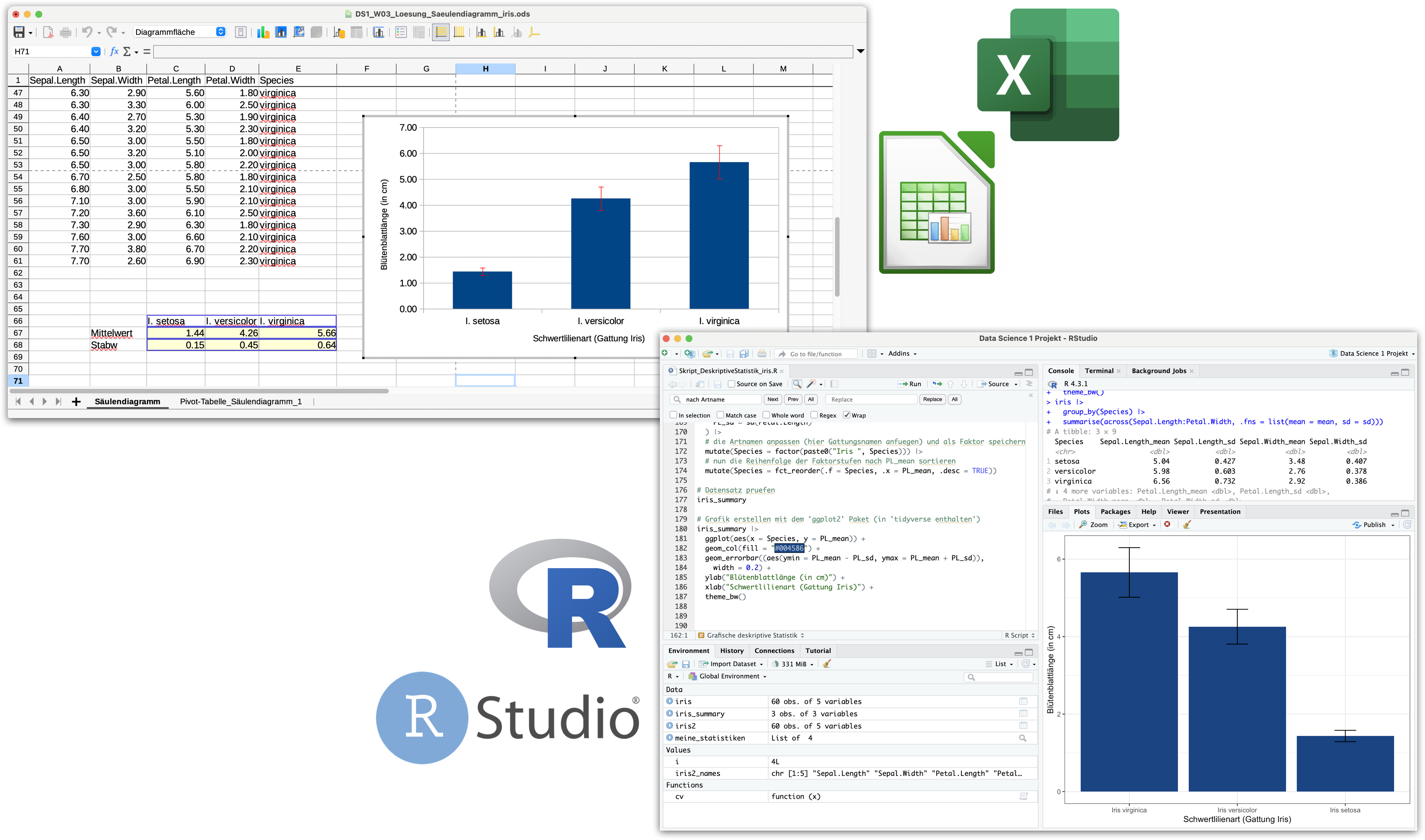
Beispiel der Übungsaufgabe in Woche 3
Von Calc zu R wechseln | Demo B
Deskriptive Statistik mit iris | 2
![]()
Das vollständige R Skript (DS1_VL04b_Demo_DeskriptiveStatistik_iris.R)
# ------------------------------ Vorbereitung ----------------------------------
#### Laden von Paketen
library(tidyverse) # laedt 9 Pakete
# # (das gleiche wie alle Pakete einzeln zu laden)
# library(dplyr)
# library(forcats)
# library(ggplot2)
# library(lubridate)
# library(purrr)
# library(readr)
# library(stringr)
# library(tibble)
# library(tidyr)
#### Eigene Funktionen
# Variantionskoeffizient
cv <- function(x) {
sd(x)/mean(x)
}
# -------------------- Import und Datenaufbereitung ----------------------------
#### Import von CSV-Dateien
# (-> Swirl-Lektion L01 in DSB-03-Datenaufbereitung oder per Anleitung durchs Tidyversum)
# Import der ODS-Datei, welche in den Zeilen 66-68 noch Text enthaelt
iris <- readODS::read_ods("DS1_W03_Saeulendiagramm_mit_iris.ods")
#### Pruefung des Imports und Datensichtung
# (-> Swirl-Lektion L02 in DSB-03)
# Pruefung des Datentyps --> IMMER DIREKT NACH DEM IMPORT VERWENDEN!
str(iris) # str = structure
# Betrachtung des Inhalts
iris
# View(iris)
# Korrektur des Datentyps und der Zeilen
iris <- iris[1:60, ] |>
mutate(across(Sepal.Length:Petal.Width, as.numeric)) |>
mutate(Species = as.factor(Species))
str(iris)
# Welche Werte kommen in jeder Spalte vor?
lapply(iris, unique)
### Weitere Funktionen zur Sichtung einzelner Aspekte
head(iris) # zeigt erste 6 Zeilen (Kopfzeilen)
tail(iris) # zeigt letzte 6 Zeilen (Endzeilen)
class(iris) # Identifikation der Objektklasse (Vektor, Matrix, dataframe,..)
nrow(iris) # Anzahl Zeilen
ncol(iris) # Anzahl Spalten
dim(iris) # Anzahl aller Dimensionen
names(iris) # Spaltennamen
typeof(iris$Sepal.Length) # Datentyp von Spalte 'Sepal.Length'
typeof(iris$Species) # Datentyp von Spalte 'Species'
# ----------------------- Deskriptive Statistik --------------------------------
#### Berechnung mehrerer Statistiken für jede Spalte im data frame
summary(iris)
#### Berechnung versch. Statistiken der Kronblattlaenge, gruppiert nach Art
# (-> Lektion L01 in DSB-02-Datenexploration mit R)
# (-> Lektion L06-Gruppierte Aggregation in DSB-03)
iris_summary <- iris |>
group_by(Species) |>
summarise(
PL_mean = mean(Petal.Length), # Mittelwert
PL_median = median(Petal.Length), # Median
PL_var = var(Petal.Length), # Varianz
PL_sd = sd(Petal.Length), # Standardabweichung
PL_se = sd(Petal.Length)/sqrt(length(Petal.Length)), # Standardfehler
PL_cv = cv(Petal.Length) # Variationskoeffizient
) |>
# die Artnamen anpassen (hier Gattungsnamen anfuegen) und als Faktor speichern
mutate(Species = factor(paste0("Iris ", Species))) |>
# nun die Reihenfolge der Faktorstufen nach PL_mean sortieren
mutate(Species = fct_reorder(.f = Species, .x = PL_mean, .desc = TRUE))
# Zusammenfassung ansehen
iris_summary
#### Saeulendiagramm erstellen mit dem 'ggplot2' Paket
# (siehe auch swirl-Kurs DSB-04-Datenvisualisierung mit ggplot2)
iris_summary |>
ggplot(aes(x = Species, y = PL_mean)) + # initiert Plot
# die Saeulen hinzufuegen
geom_col(fill = "#004586") +
# die Fehlerbalken hinzufuegen
geom_errorbar((aes(ymin = PL_mean - PL_sd, ymax = PL_mean + PL_sd)),
width = 0.2) +
# Achsenbeschriftung anpassen
ylab("Kronblattlänge (in cm)") +
xlab("Schwertlilienart (Gattung Iris)") +
# das Layout anpassen
theme_bw()Von Calc zu R wechseln | Demo B
Deskriptive Statistik mit iris | 3
Das Skript enthält
- viele Kommentare zur Nachvollziehbarkeit und Skriptstrukturierung
- viele Variablen bzw. Objekte
- viele Operatoren
- viele built-in Funktionen aus base R und tidyverse Paketen
- mit entsprechenden Datenverweisen (analog zu Zellverweisen in Calc und Excel)
- die Definition einer eigenen Funktion
- eine Schleife
Wir werden Abschnitte beider Skripte in den kommenden Vorlesungen wiederholt behandeln.
Übungsaufgabe

Vorbereitungen | 1
Posit/RStudio Workbench
![]()
- Loggen Sie sich in die Workbench ein.
- Wählen Sie im Fenster Files die Option Upload. Klicken Sie im Dialogfenster auf Datei auswählen und wählen Sie die ZIP-Datei “DS1-R-Uebungen.zip”, die Sie von Moodle heruntergeladen haben.
- Wichtig: Die Datei darf nicht entpackt sein!
- Im Fenster Files sollte nun ein Ordner mit dem Namen “DS1-R-Uebungen” erscheinen.
- Doppelklicken Sie auf die Datei “DS1-R-Uebungen.Rproj”. Bestätigen Sie, dass Sie das Projekt öffnen möchten. Die Oberfläche lädt neu, und oben rechts sehen Sie nun den Projektnamen ‘DS1-R-Uebungen’ anstelle von ‘Project: (None)’.
- Machen Sie sich kurz mit der Benutzeroberfläche (GUI) vertraut.
- Öffnen Sie das Skript “DS1_W04_Swirlkurs.R” und führen Sie die ersten Zeilen aus. Nutzen Sie dieses Skript für alle folgenden R-Code-Befehle im ersten Swirl-Kurs!
Link zur Workbench-Webadresse
Vorbereitungen | 2
Alternativ: RStudio Desktop lokal nutzen
![]()
![]()
- Installieren Sie R: https://cran.r-project.org/
- Installieren Sie RStudio Desktop: https://posit.co/products/open-source/rstudio/
- Entpacken Sie die ZIP-Datei “DS1-R-Uebungen.zip”, die Sie von Moodle heruntergeladen haben, und speichern Sie den Ordner an einem geeigneten Ort auf Ihrem Rechner.
- Doppelklicken Sie im Ordner “DS1-R-Uebungen” auf die Datei “DS1-R-Uebungen.Rproj”, um RStudio zu starten. Im Fenster Files sollte nun der Projektordner ‘DS1-R-Uebungen’ mit seinem Inhalt angezeigt werden.
- Machen Sie sich kurz mit der Benutzeroberfläche (GUI) vertraut.
- Öffnen Sie das Skript “Local_Setup.R” und führen Sie den gesamten Code einmalig aus, um die benötigten R-Pakete lokal zu installieren.
- Öffnen Sie anschließend das Skript “DS1_W04_Swirlkurs.R” und führen Sie die ersten Zeilen aus. Nutzen Sie dieses Skript für alle folgenden R-Code-Befehle im ersten Swirl-Kurs!
Wichtig vorab: Ausführen von 'Local_Setup.R'
### Fuehren Sie diesen Code aus bevor Sie mit der ersten Übungsstunde in R
### anfangen (Woche 4)
# Zu installierende Pakete von CRAN fuer dieses Modul
pcks_cran <- c(
"tidyverse", # fuer Datenaufbereitung und Visualisierung
"readODS", # fuer Import von Calc-Dateien (ODS-Format)
"readxl", # fuer Import von Excel-Dateien (XLSX-Format)
"swirl", # fuer die swirl-Kurse
"remotes" # fuer die Installation von Paketen, die z.B. auf GitHub sind
)
# Pakete installieren
install.packages(pcks_cran)
# Das DSBswirl Paket von GitHub installieren
remotes::install_github("uham-bio/DSBswirl")
# Installation der 6 Swirl-Kurse, welche in DSBswirl enthalten sind
DSBswirl::install_dsb_courses()swirl-Kurse in Data Science 1
![]()
- Begleitend zu diesem Modul gibt es 6 Kurse mit bis zu 16 Lektionen (im Englischen ‘lessons’), die alle jeweils 5-20min in Anspruch nehmen.
- Mit swirl können Sie interaktiv direkt in R bzw. RStudio lernen.
- Eine Unterbrechung ist jederzeit möglich, so dass alle Kurslektionen nach und nach bearbeitet werden können.
Kursübersicht
- DS1-01-R Grundlagen
- DS1-02-Datenexploration mit R (mit Basisfunktionen)
- DS1-03-Datenaufbereitung oder per Anleitung durchs Tidyversum
- DS1-04-Datenvisualisierung mit ggplot2
- DS1-05-Handling spezieller Datentypen
- DS1-06-Fortgeschrittene R Programmierung
Starten von swirl-Kursen
![]()
- Laden Sie das ‘swirl’ Paket und starten Sie das swirl-Kursprogramm mit der
swirl()Funktion (bei jeder session):
- Sie werden bei jeder neuen Session nach einem Namen gefragt:
- Um auf den Zwischenstand der letzten Lektionen zugreifen zu können, sollten Sie immer den gleichen Namen verwenden!
- Verwenden Sie bei RStudio Workbench Ihren Usernamen als swirl Nutzernamen!!!
- Wählen Sie anschl. den ersten Kurs aus (geben Sie die entsprechende Indexnummer in die Konsole).
- Wählen Sie nun eine Lektion in der Lektionsübersicht des Kurses.
swirl - Wichtige Befehle
![]()
- … → Enter drücken
- Esc → swirl verlassen, zurück zum R-Prompt
bye()→ swirl beenden, Fortschritt wird gespeichertskip()→ aktuelle Frage überspringenplay()→ selbst mit R arbeitennxt()→ zurück zu swirlmain()→ Hauptmenü öffneninfo()→ Befehlsübersicht anzeigen
Zu bearbeitende swirl-Lektionen
diese Woche
![]()
Kurs DS1-01-R Grundlagen
- L01-Kurseinfuehrung
- L02-R als Taschenrechner
- L03-Arbeitsbereich aufraeumen
- L04-Arbeitsverzeichnis einstellen
- L05-Verwendung der R Hilfe
- L06-Variablenzuweisungen und Grundfunktionen
- L07-Datentypen und Operatoren
Wie fühlen Sie sich jetzt…?

Total konfus?

Keine Sorge…
… im swirl-Kurs werden Sie direkt an die Hand genommen und Stück für Stück angeleitet.
Total gelangweilt?

Dann testen Sie doch Ihr Wissen in folgendem Abschlussquiz…
Abschlussquiz
![]()

Bei weiteren Fragen: saskia.otto(at)uni-hamburg.de

Diese Arbeit is lizenziert unter einer Creative Commons Attribution-ShareAlike 4.0 International License mit Ausnahme der entliehenen und mit Quellenangabe versehenen Abbildungen.
Kurswebseite: Data Science 1
