
Kurseinführung
Data Science 1 - Programmieren & Visualisieren
Saskia Otto & Monika Eberhard
Universität Hamburg, IMF
Wintersemester 2025/2026
Was ist ‘Data Science’?

Aktuell sehr populär!
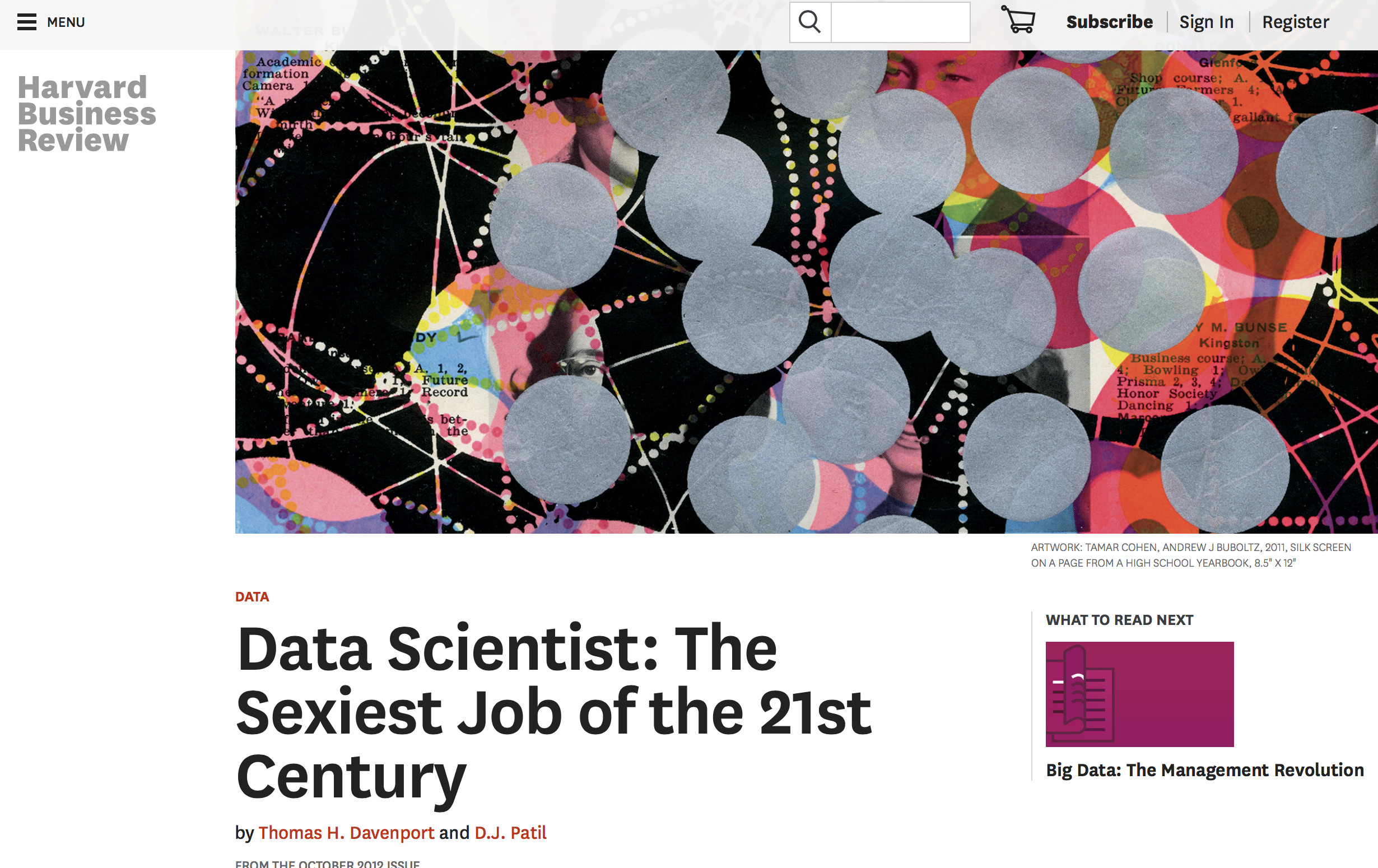
Quelle: Screenshot der Harvard Business Review Website vom 11. November 2017.
Die perfekten Wissenschaftler

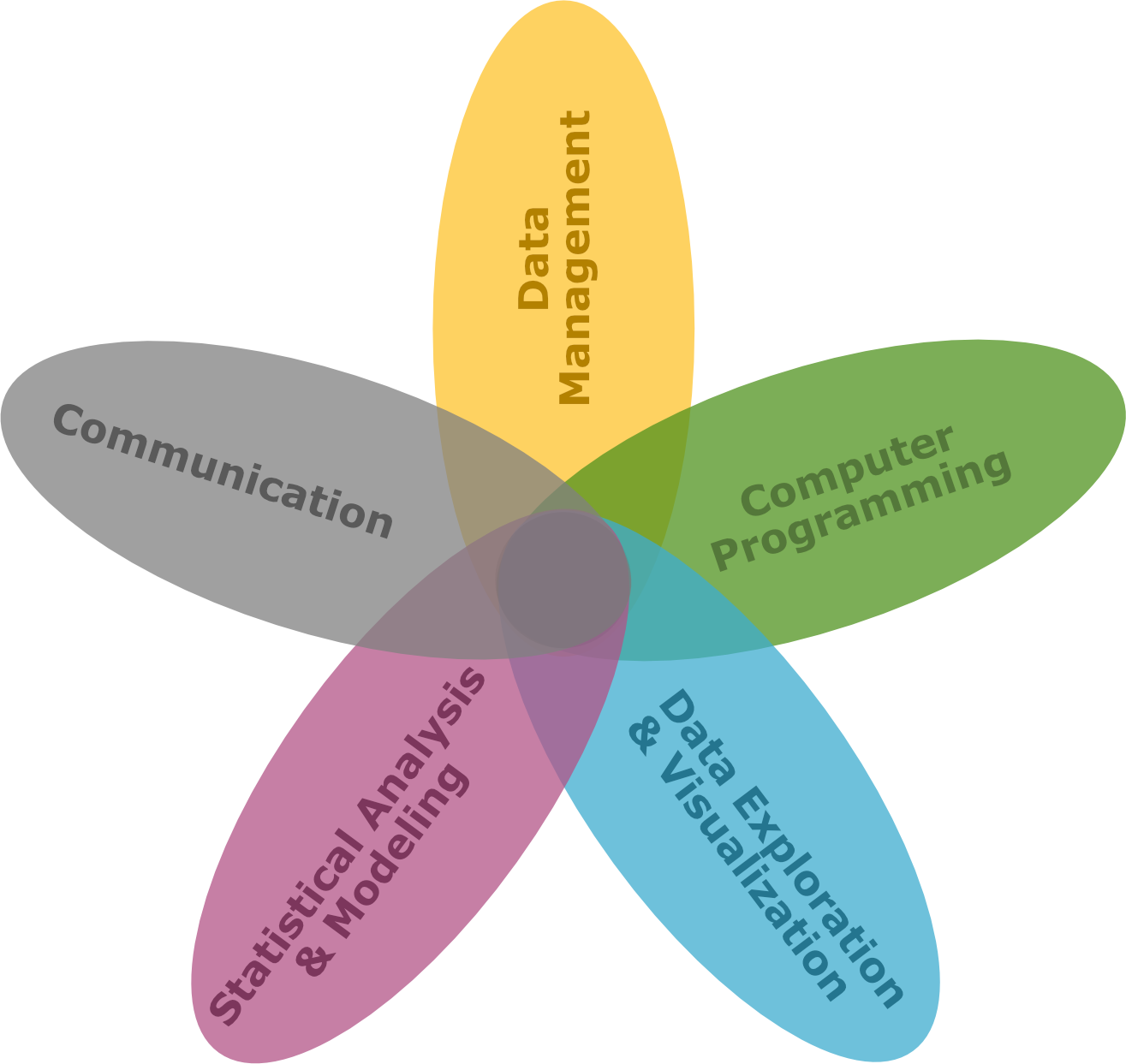
Die 5 Hauptkomponenten in Data Science
Das Data Science in Biology Programm
Übersicht der Bachelormodule


Zu diesem Modul

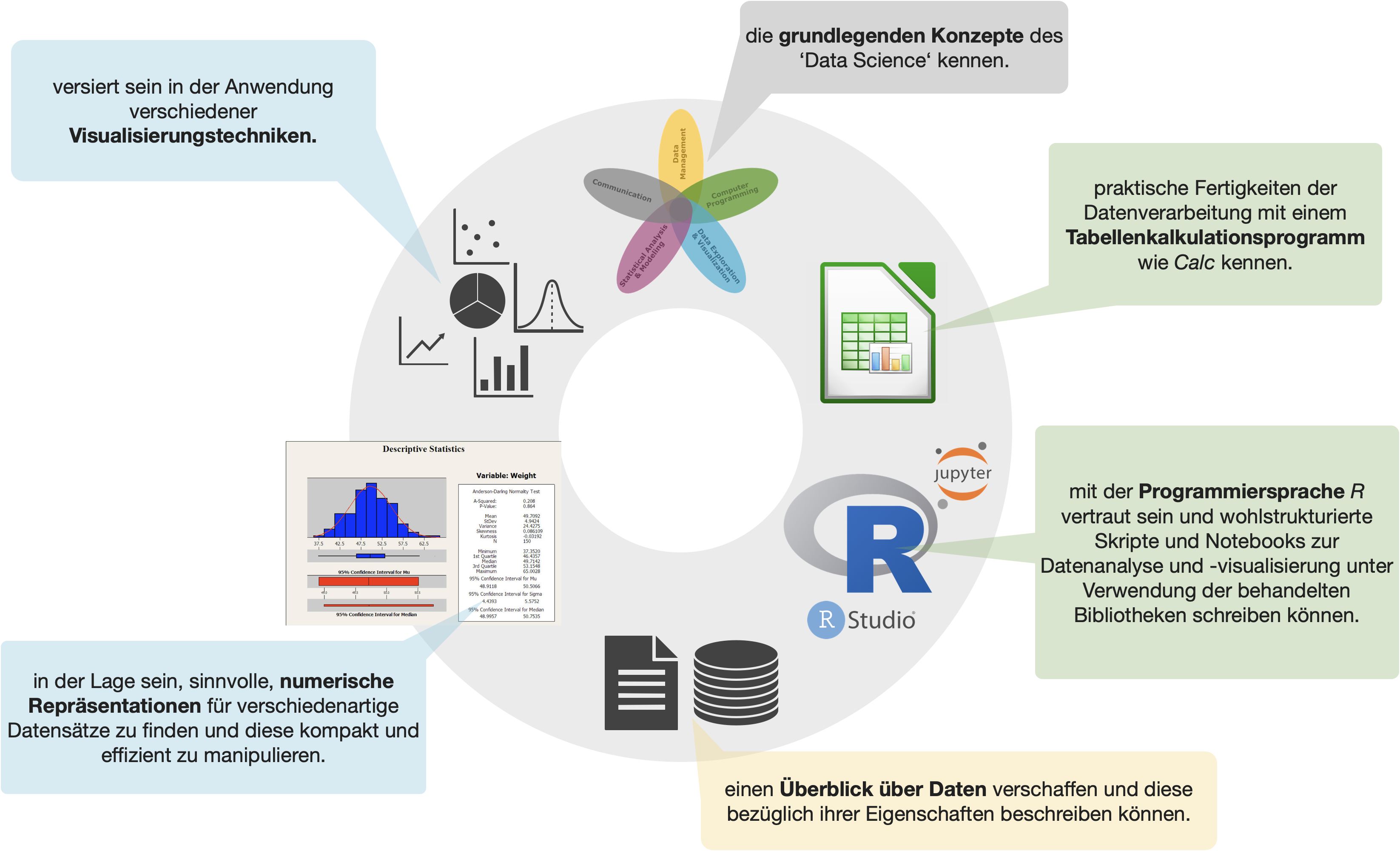
Lernziele
Am Ende dieses Moduls werden Sie…
![]()
Modulstruktur
Interaktive Vorlesungen
- Web-basierte Vorlesungen (VL)
- Quizze während der VL (HTML)
- Paralleles Programmieren (RStudio Desktop/Workbench)
- Abschließende Wissensquizze (HTML) als Vorbereitung zur Klausur
- Hilfreich: Handy, Tablet oder Computer
Übungen
- Praktische Übungen mittels swirl-Kurs bzw. R Skripten im Rahmen betreuter Gruppenarbeit
- Angewandte Online-Quizze auf Moodle als weitere Vorbereitung zur Klausur
- Bearbeitung einer Fallstudie mit abschließender Überprüfung (Moodle)
- Nötig: Computer oder Tablet
- Anwesenheitspflicht, 15% (= 2 Übungstage) Fehlzeit erlaubt
Vorlesungsthemen - Zeitplan
![]() Termin: Montags, 11:15-12:45, Gr. Hörsaal (MLK 3)
Termin: Montags, 11:15-12:45, Gr. Hörsaal (MLK 3)
- Oktober: Data Science Allgemein
- Einführung in Daten und Datenmanagement
- Grundlagen der deskriptiven Statistik
- Biologische Prozesse mathematisch beschreiben
- November/Dezember: R Programmierung
- Einführung in die (R) Programmierung
- Grundlagen in R: Variablen und Vektoren
- Grundlagen in R: Komplexere Objekte
- Datenaufbereitung mit tidyverse
- Datevisualisierung mit base R und ggplot2
- Maßgeschneiderte ggplot2 Diagramme
- Januar: Visualisierung
- Grundlagen der Visualisierung
- Kartenerstellung mit ggplot und leaflet und interaktive Grafiken
- Handhabung spezieller Datentypen
Übersicht zu den Übungen
Gruppenzeiten
| Gruppe | Tag | Zeit | Ort | Betreuung |
|---|---|---|---|---|
| A | Mo | 14:00-15:30 | IZS, gr. HS | Monika Eberhard |
| B | Mo | 14:00-15:30 | IZS, Kosswig Saal | Simon Bober |
| C | Mi | 11:00-12:30 | IPM, CvL-HS | Rebecca Oester & Katharina Meyer |
| D | Mi | 11:00-12:30 | IPM, E.004 | Julian Mittmann-Götsch |
| E | Mi | 15:15-16:45 | IZS, 017 | Alexej Sinner & Stephanie Schultz |
| F | Mi | 15:15-16:45 | IZS, Kosswig Saal | Monika Eberhard |
| G | Mi | 15:30-17:00 | IPM, E.303 | Dirk Granse |
| H | Mi | 15:30-17:00 | IPM, E.004 | Carolin Menzel |
| I | Do | 14:15-15:45 | IZS, Kosswig Saal | Joanna Malukiewicz |
| J | Do | 14:15-15:45 | IZS, gr. HS | Simon Bober |
| K | Do | 15:30-17:00 | IPM, CvL-HS | Rebecca Oester & Katharina Meyer |
| BMARSYS | Mi | 13:15-14:45 | IMF, GES107 | Saskia Otto |
Moodle Kurs
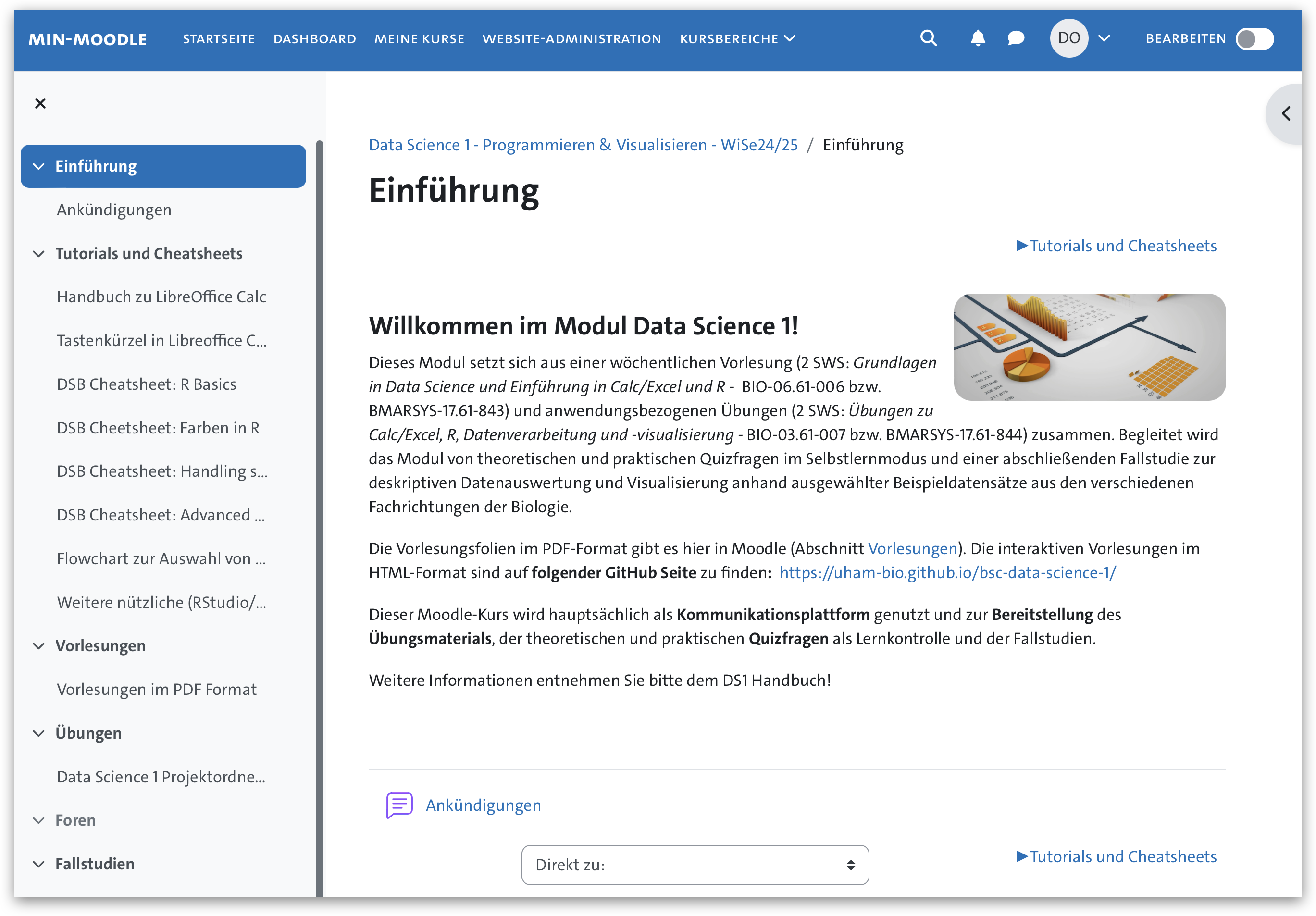

- Lernmaterial
- VL Aufzeichnungen
- Übungsdateien
- Quizfragen
- Fallstudie
- Klausur
- Kommunikation (Forum/Nachrichten)
Webseite mit Vorlesungsfolien
![]()

Kurswebseite: https://uham-bio.github.io/bsc-data-science-1/
Handbuch zum Modul

Das Handbuch enthält Hinweise und Erläuterungen zum Modul (Teil 1), eine Formelsammlung (Teil 2) und Beschreibungen der Übungsaufgaben (Teil 3).
Übungsdateien zum Download
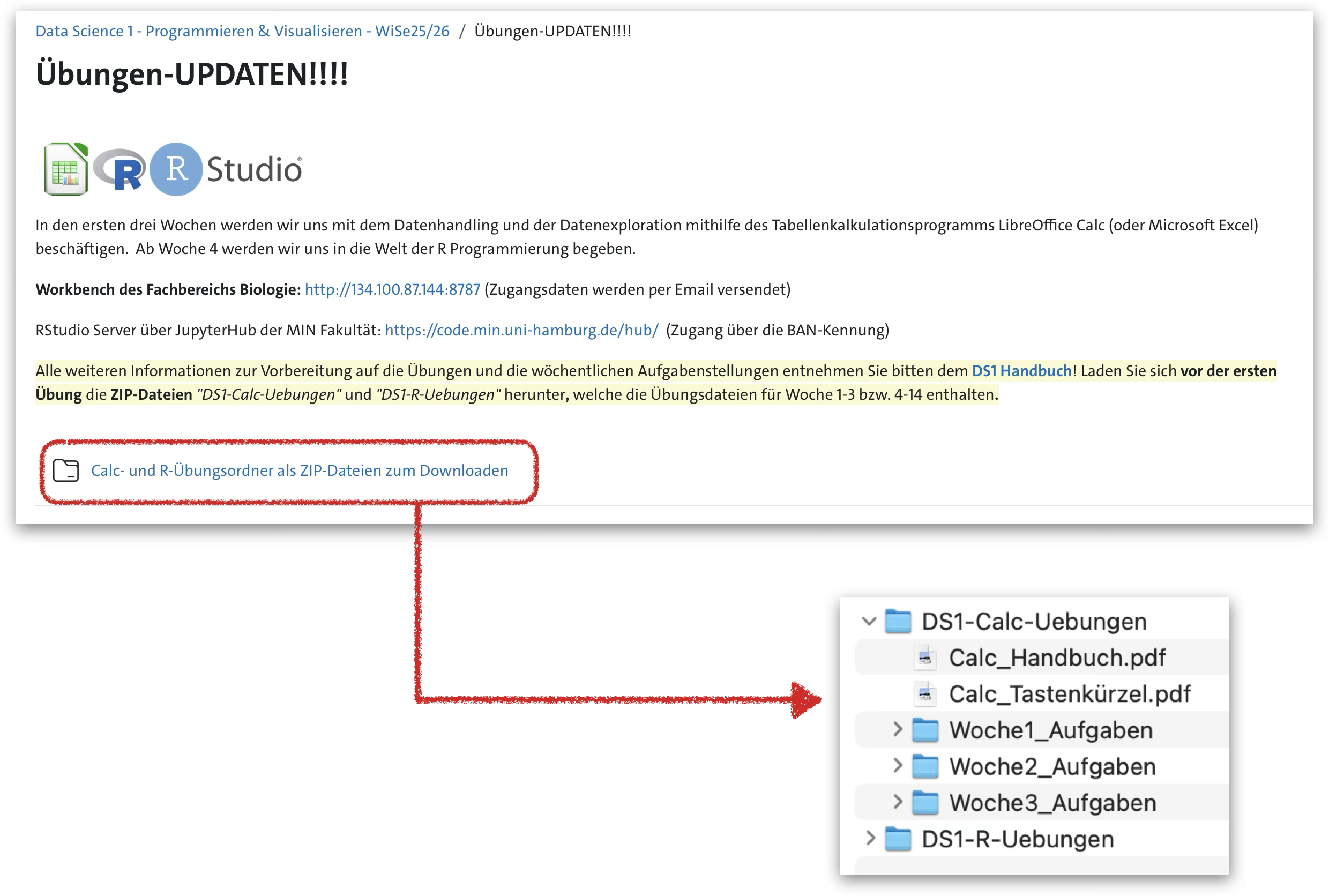
Vor der ERSTEN Übungsstunde von Moodle herunterzuladen!
Software
Vorbereitung zu den Übungen
Installieren Sie auf Ihrem Computer (Tablets funktionieren nicht):
Vor dem VIERTEN Übungsttag


- Installieren Sie R UND RStudio Desktop.
- Alternativ können Sie unsere RStudio Workbench nutzen, die auch auf Tablets/iPads funktioniert.
Mehr Informationen zur Installation der Software finden Sie im Handout.
Fallstudie (ab November)
Pilotprojekt zur Citizen Science Initiative des Projekts Lebendige Alster
- Bearbeitung einer Fallstudie im 2-3er Team
- Datenerhebung als Übungsgruppe
- Abgabe:
- Digitalisierung der Messdaten im CSV-Format
- R Skript
- Kurzbericht (MS Word/Libreoffice Writer)

Buchempfehlungen
Hier ein paar Vorschläge…
- Data Science mit R
- The R Book von Michael J. Crawley (2013), 2. Edition, Wiley & Sons, Ltd., West Sussex, UK, 975 S. → online verfügbar als PDF ( Link)
- R for Data Science von Hadley Wickham & Garret Grolemund (2017), O’Reilly Media Inc., CA, U.S.A, 494 S. → online verfügbar
- Visualisierung mit ggplot2
- ggplot2 - Elegant Graphics for Data Analysis von Hadley Wickham (2016), 2. Edition, Springer International Publishing, Switzerland, 260 S. → online verfügbar
- Guide to Create Beautiful Graphics in R von Alboukadel Kassambara (2013), 2. Edition, STHDA, 237 S.
- Visualisierung allgemein
- Data Visualisation - A Handbook for Data Driven Design von Andy Kirk (2019), 2. Auflage, SAGE Publications Ltd., 312 S.
- Good Charts Workbook von Scott Berinato (2019), HBR Press, 279 S.
Ready for the jump?
Quelle: www.pixabay.com (CCO 1.0 Lizenz)

Bei weiteren Fragen: saskia.otto(at)uni-hamburg.de

Diese Arbeit is lizenziert unter einer Creative Commons Attribution-ShareAlike 4.0 International License mit Ausnahme der entliehenen und mit Quellenangabe versehenen Abbildungen.
Kurswebseite: Data Science 1

